Таблица 1.
Использованные команды |
| Команда |
Описание |
| java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr13.1.fastq chr13.1_trim.fastq TRAILING:20 MINLEN:50 |
Чистка чтений |
| fastqc chr13.1_trim.fastq |
Анализ программой FastQ |
| hisat2 -x chr13 -U chr13.1_trim.fastq -S chr13.1_al.sam --no-softclip |
Выравнивание. Параметр --no-spliced-alignment был исключен, поскольку на вход дается РНК-транскрипт с вырезанными интронами, поэтому экзоны картируются кусками. |
| samtools view -b chr13.1_al.sam -o chr13.1_al.bam |
Перевод выравнивания в бинарный формат |
| samtools sort chr13.1_al.bam chr13.1_al_sort |
Сортировка выравниваний по координате в референсе |
| samtools index chr13_al_sort.bam |
Индексирование |
| htseq-count -f bam -s no -i gene_id -m union chr13.1_al_sort.bam gencode.v19.chr_patch_hapl_scaff.annotation.gtf > count.txt |
Подсчет чтений.
-f -формат файла sam/bam
-s цепь по которой будут выравнены риды, для одноконцевых чтений лнучше no, как следует из документации.
-i установлен по умолчанию, считает повторы в GFF файле.
-m определяет то как риды перекрываются |
| grep -wv 0 count.txt > count_good.txt |
Только гены с ненулевыми чтениями |
Была проанализировнна 13-ая хромосома.
Было получено 12985 чтений длинной 34-51 нуклеотидов.
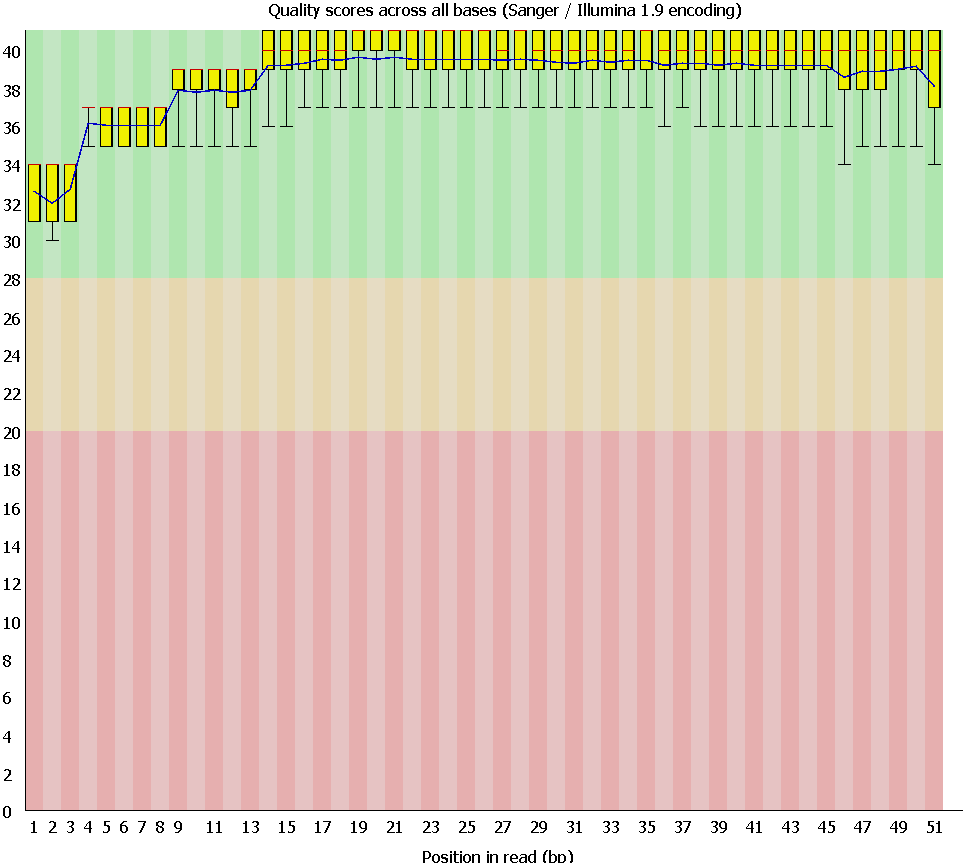
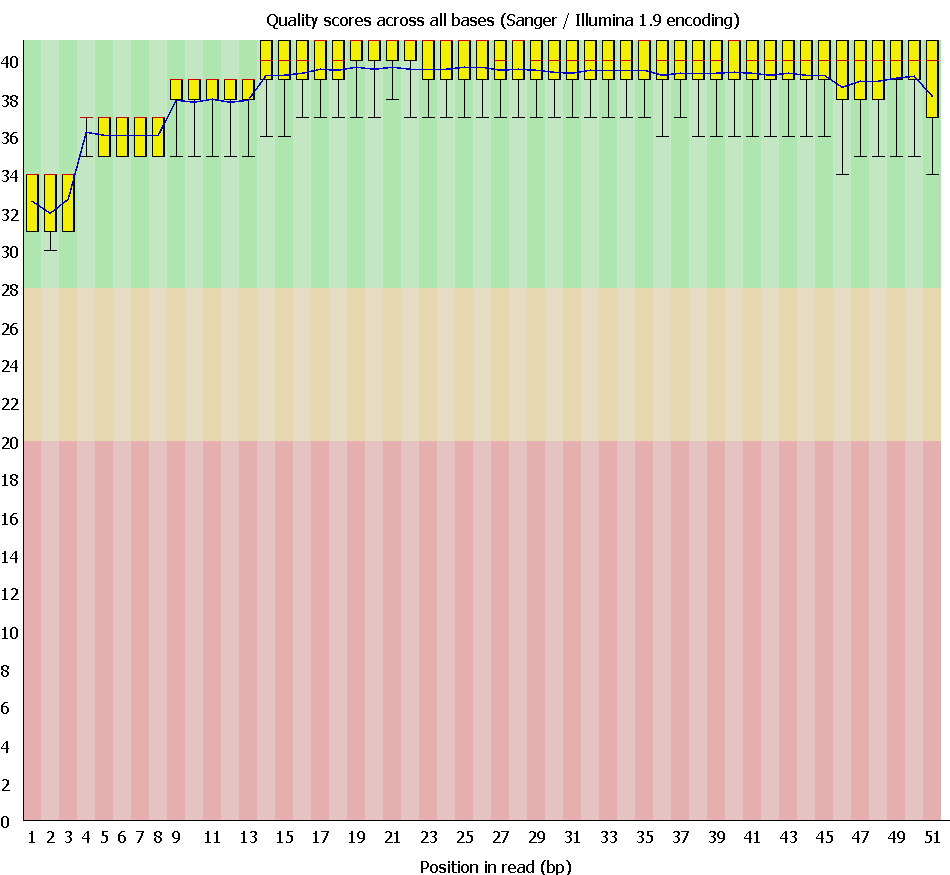
Очистка не сильно повлияла на качество, унеся с собой всего 35 чтений.
По выходу Hisat можно судить о том что качество покрытия довольно высокое, несмотря на то, что все легло лишь единожды.
12950 reads; of these:
12950 (100.00%) were unpaired; of these:
121 (0.93%) aligned 0 times
12829 (99.07%) aligned exactly 1 time
0 (0.00%) aligned >1 times
99.07% overall alignment rate
Рис. 1 Качество чтений до отчистки
Рис. 2 Качество чтений после отчистки