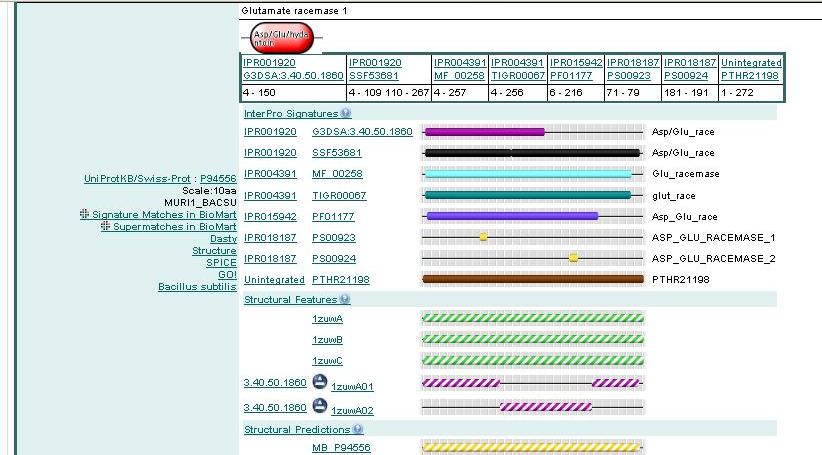
1. Самый короткий мотив Asp/Glu_race описан в БД Gene3D, использующей кластеризирующий алгоритм Маркова (Markov clustering algorithm).
2. Самый длинный мотив PTHR21198 описан в БД PANTHER, использующей скрытые марковские модели (Hidden Markov models). В данной базе последовательность не интегрирована и включает все остатки.
3. Структурные надписи: 1zuwA, 1zuwB, 1zuwC - последовательности 3-х идентичных цепей из PDB; 1zuwA01 и 1zuwA02 из БД CATH.
Очевидного соответствия структурных доменов и мотивов не наблюдается.
© В. А. Костырко
 [Silicibacter pomeroyi] Aspartate racemase, putative (485 residues)
[Silicibacter pomeroyi] Aspartate racemase, putative (485 residues)
 [Candidatus Kuenenia stuttgartiensis] Putative uncharacterized protein (916 residues)
[Candidatus Kuenenia stuttgartiensis] Putative uncharacterized protein (916 residues)
 [Talaromyces stipitatus] Hydantoin racemase (Dcg1), putative (683 residues)
[Talaromyces stipitatus] Hydantoin racemase (Dcg1), putative (683 residues)