Проверка качества ридов
Несмотря на то, что не было ридов, помеченных как некачественные, из-за провала качества per tile, а также из-за длинных усов на диаграмме качества per base, и наличия относительно большого количества чтений со средним качеством 2, была проведена очистка программой Trimmomatic (убраны риды с качеством ниже 50 и длиной меньше 36):
java -jar /usr/share/java/trimmomatic.jar SE -phred33 chipseq_chunk51.fastq chipseq_chunk51_out.fastq TRAILING:50 MINLEN:36
Таким образом, из 12221 ридов было удалено 1919 (15,70%). Исчезли риды со средним качеством ниже 18, соответствующим образом изменились показатели качества per base и per tile.
Ссылка на информацию по ридам до очистки
Ссылка на информацию по ридам после очистки
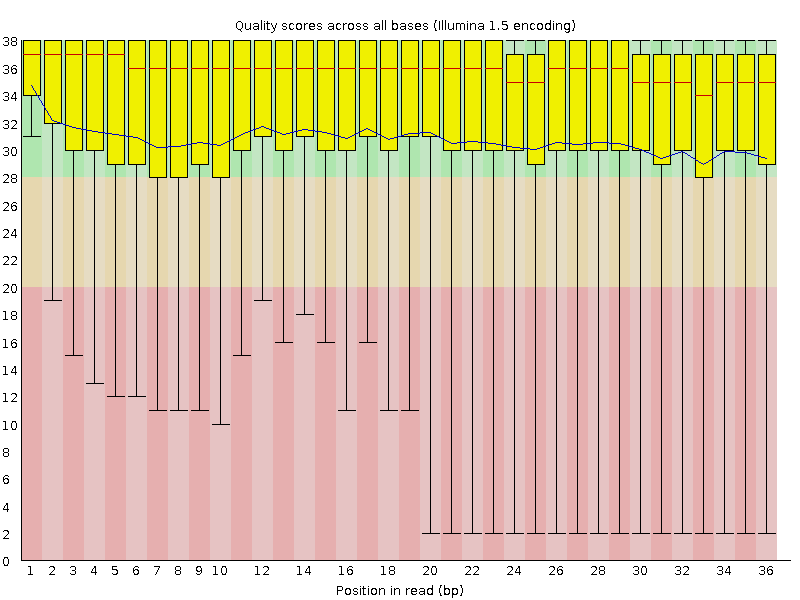
Рис.1. Диаграмма качества чтений per base до очистки
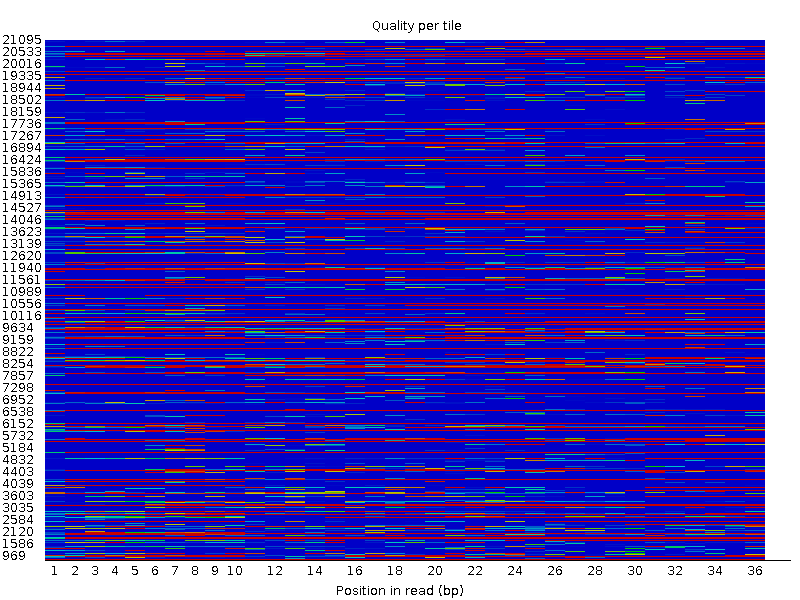
Рис.2. Диаграмма качества чтений per tile до очистки
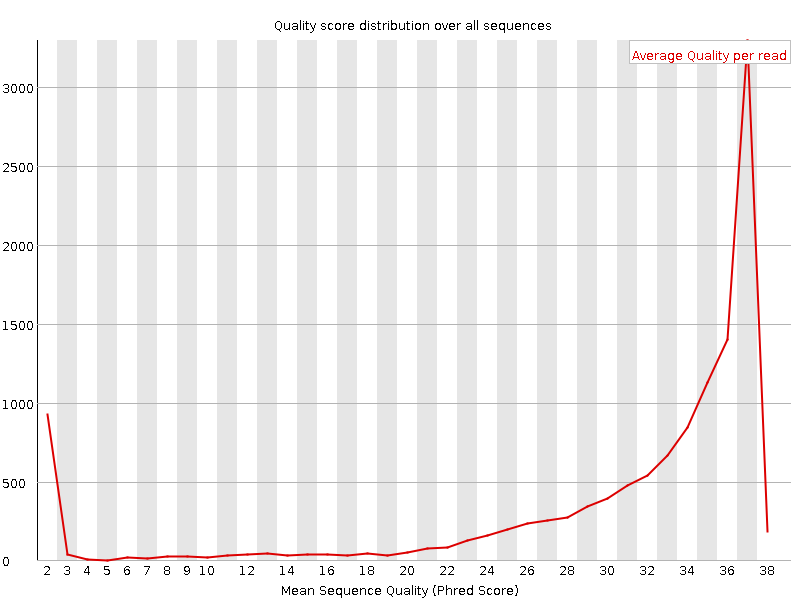
Рис.3. Распределение чтений по среднему качеству до очистки
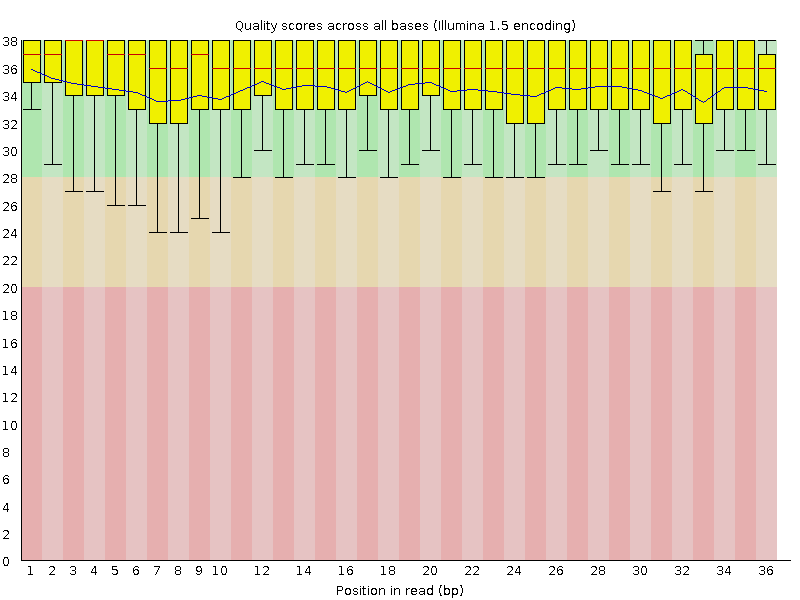
Рис.4. Диаграмма качества чтений per base после очистки
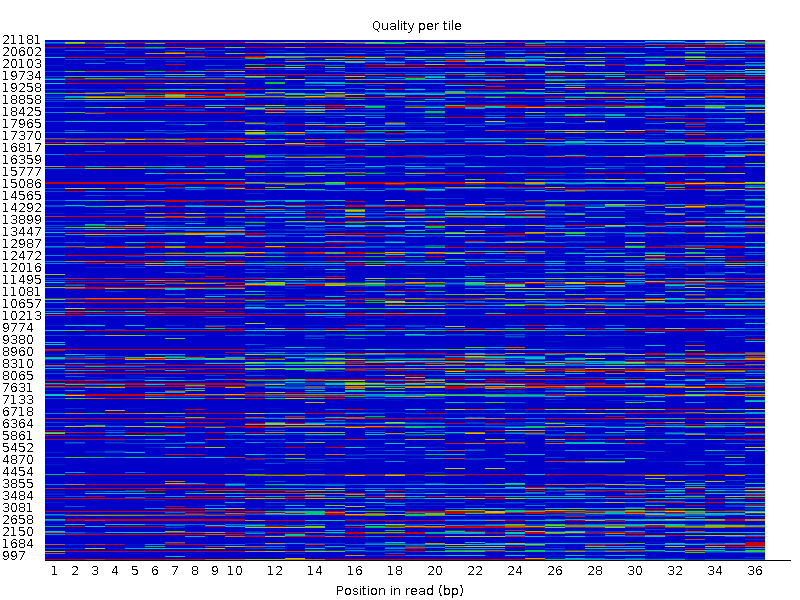
Рис.5. Диаграмма качества чтений per tile после очистки
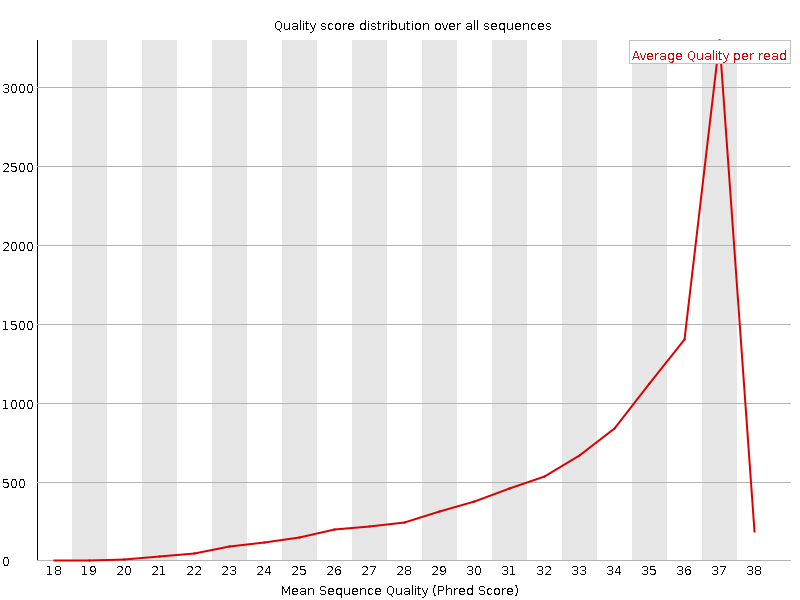
Рис.6. Распределение чтений по среднему качеству после очистки
Картирование ридов на геном человека
Картрирование выполнено с помощью команды:
bwa mem /srv/databases/ngs/hg19/GRCh37.p13.genome.fa chipseq_chunk51_out.fastq > chipseq_chunk51.sam
Далее файл переведен в бинарный формат .bam
samtools view chipseq_chunk51.sam -bSo chipseq_chunk51.bam
Проведена сортировка выравниваний ридов и референса по координанте рида в референсе
samtools sort chipseq_chunk51.bam -T chtmp -o chipseq_chunk51_sort.bam #-T chtmp делает папку для временных файлов
Индексация полученного файла
samtools index chipseq_chunk51_sort.bam
Сбор статистики по выравниванию ридов по хромосоме в файл
samtools idxstats chipseq_chunk51_sort.bam >chip_seq51.out
Ссылка на число ридов, выравненных на каждой хромосоме
Поиск пиков
Для поиска пиков использовалась команда:
callpeak -n chipseq_chunk51 -t chipseq_chunk51_sort.bam --nomode
Получены файлы:
chipseq_chunk51_peaks.narrowPeak
Итого найдено 8 пиков в хромосоме 8.
Таблица 1. Параметры пиков.
| Номер | Координаты | Ширина | Расстояние от начала до пика | -log10(p-value) |
| 1 | 12953978-12954316 | 339 | 167 | 19.08339 |
| 2 | 12998122-12998344 | 222 | 104 | 20.45434 |
| 3 | 13067811-13068140 | 330 | 183 | 26.15991 |
| 4 | 13123323-13123688 | 366 | 177 | 40.71928 |
| 5 | 13132584-13133029 | 446 | 186 | 46.27156 |
| 6 | 13187976-13188350 | 375 | 180 | 18.51964 |
| 7 | 13306704-13306975 | 272 | 185 | 14.45962 |
| 8 | 13402358-13402562 | 205 | 16 | 10.70424 |
Таким образом, самые достоверные пики - 4 и 5, т.к. e-value меньше 10^-40.
Для визуализации файл chipseq_chunk51_peaks.narrowPeak был загружен в UCSC Genome Browser с добавлением в начало файла строк:
track type=narrowPeak visibility=3 db=hg19 name="my_peaks" description="Peaks from chunk 51" browser position chr8:12950000-13410000
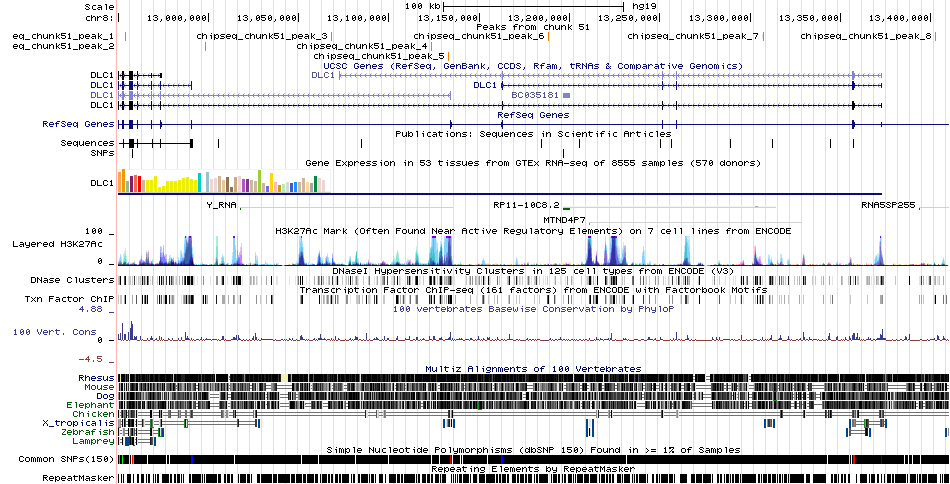
Рис.7. Визуализация пиков в геномном браузере.
Для более внимательного рассмотрения были выбраны пики 4 и 5, как наиболее достоверные.
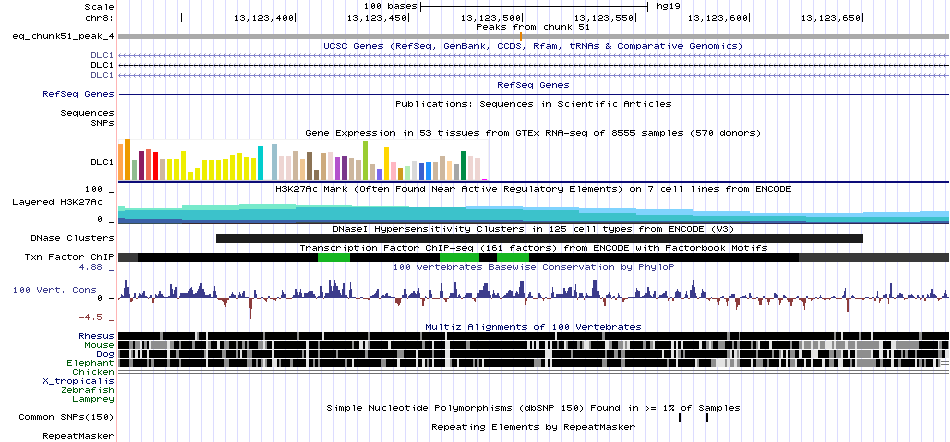
Рис.8. Визуализация пика 4 в геномном браузере.
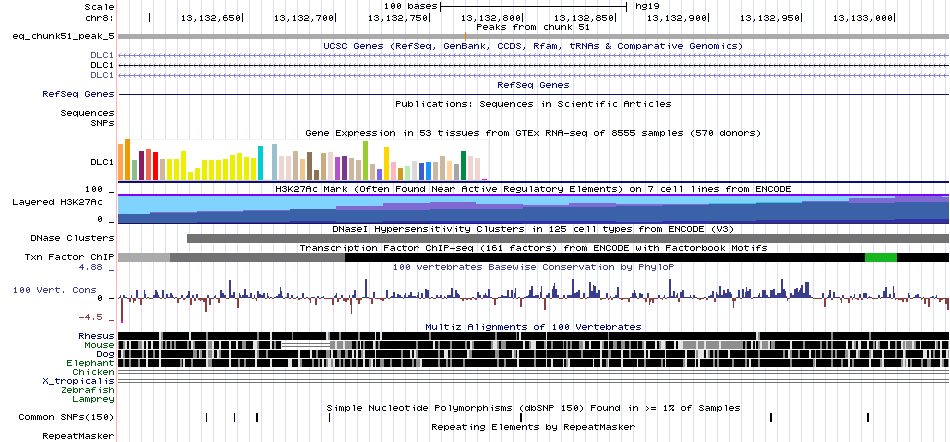
Рис.9. Визуализация пика 5 в геномном браузере.
Оба пика находятся на интроне гена DLC1, кодирующего один из белков семейства Rho ГТФ-связывающих белков. DLC1 играет значительную роль в регуляции малых ГТФ-связывающих белков, являясь супрессором опухолей. Для данного гена характерно большое количество альтернативных транскриптов благодаря наличию альтернативных промоторов и альтернативного сплайсинга. Ген располагается на комплементарной цепи, таким образом, пики располагаются в upstream области как минимум для двух вариантов транскриптов:
DLC1 (uc003wwk.1) at chr8:12940872-12990809 - Homo sapiens deleted in liver cancer 1 (DLC1), transcript variant 2, mRNA.
DLC1 (uc011kxx.1) at chr8:12940872-12973753 - Homo sapiens deleted in liver cancer 1 (DLC1), transcript variant 4, mRNA.