Дерево по нуклеотидным последовательностям
Поиском в базе NCBI Nucleotide были найдены последовательности 16S РНК бактерий из практикума, было сделано их выравнивание алгоритмом Muscle в программе AliView, после чего было построено дерево алгоритмом Neighbour Joining в программе MEGA7.
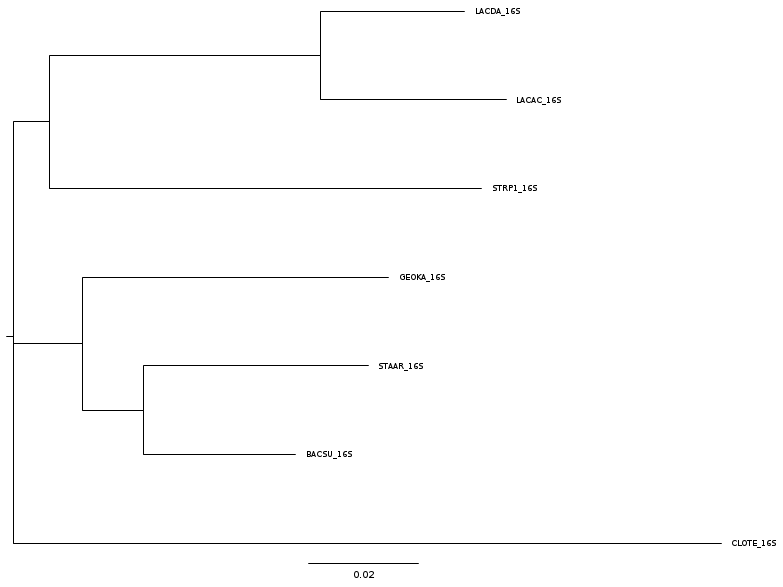
Рис.1. Дерево, построенное по последовательностям 16S РНК
Можно увидеть, что топология дерева в точности соответствует дереву из практикума 1, что подтверждает хорошие характеристики 16S РНК для реконструкции филогении у бактерий.
Ссылка на дерево в скобочном формате
Поиск паралогов и ортологов
Файлы протеомов моих бактерий были помещены в один, с помощью blastp был проведен поиск гомологов белка CLPX_BACSU, с порогом e-value = 0.001. Полученные гомологи были выравнены в программе AliView алгоритмом Muscle, по выравниванию построено дерево в программе MEGA 7 алгоритмом Neighbour Joining.
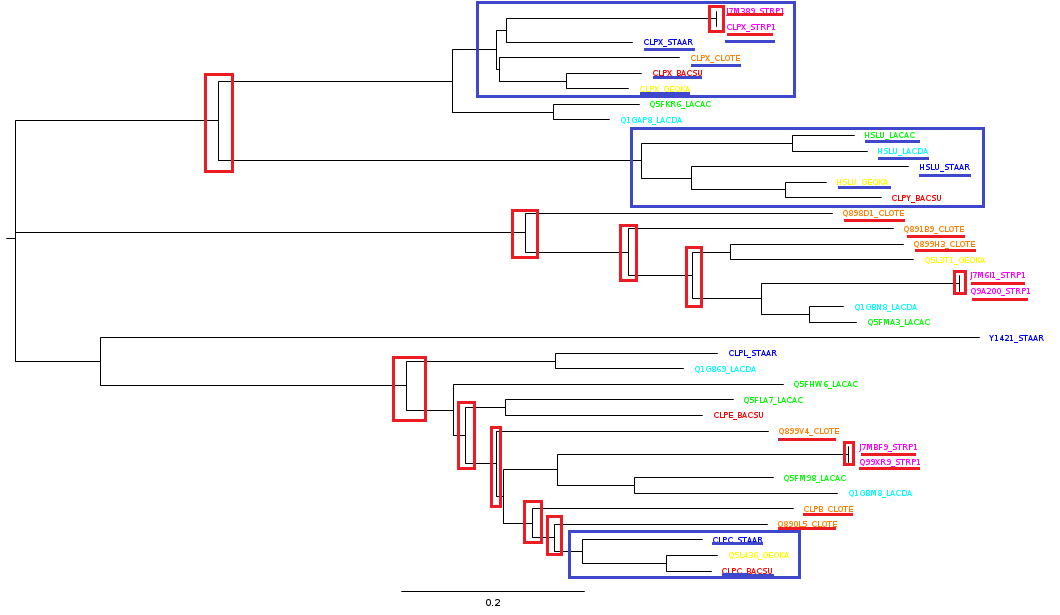
Рис.2. Дерево, построенное по гомологам CLPX_BACSU
Дупликации отмечены красной рамкой, расхождение в результате видообразования - синей. Также красным подчеркнуты паралоги (самые явные - у Streptococcus pyogenes serotype M1, STRP1, фиолетовый), синим - ортологи (гены CLPC, CLPX, HSLU.
Ссылка на выдачу алгоритма blastp
Ссылка на выравнивание последовательностей гомологов
Ссылка на дерево в скобочной форме