Изначально указанный белок был заменен на 100% идентичный WP_013634629.1 в ходе реаннотации (альфа-глюкозидаза Pseudopedobacter saltans), с ним и шла вся дальнейшая работа.
Таблица 1. Описание найденного КОГ
| Идентификатор КОГа | E-value | Длина белка | Интервал | КОГ и функциональная категория на английском | КОГ и функциональная категория на русском |
| COG1501 | 4.83e-87 | 719 | 84-714 | Alpha-glucosidase, glycosyl hydrolase family GH31, list G: carbohydrate transport and metabolism | Альфа-глюкозидаза, семейство гликозилгидролаз GH31, список G: транспорт и метаболизм углеводов |
Визуализация геномного окружения
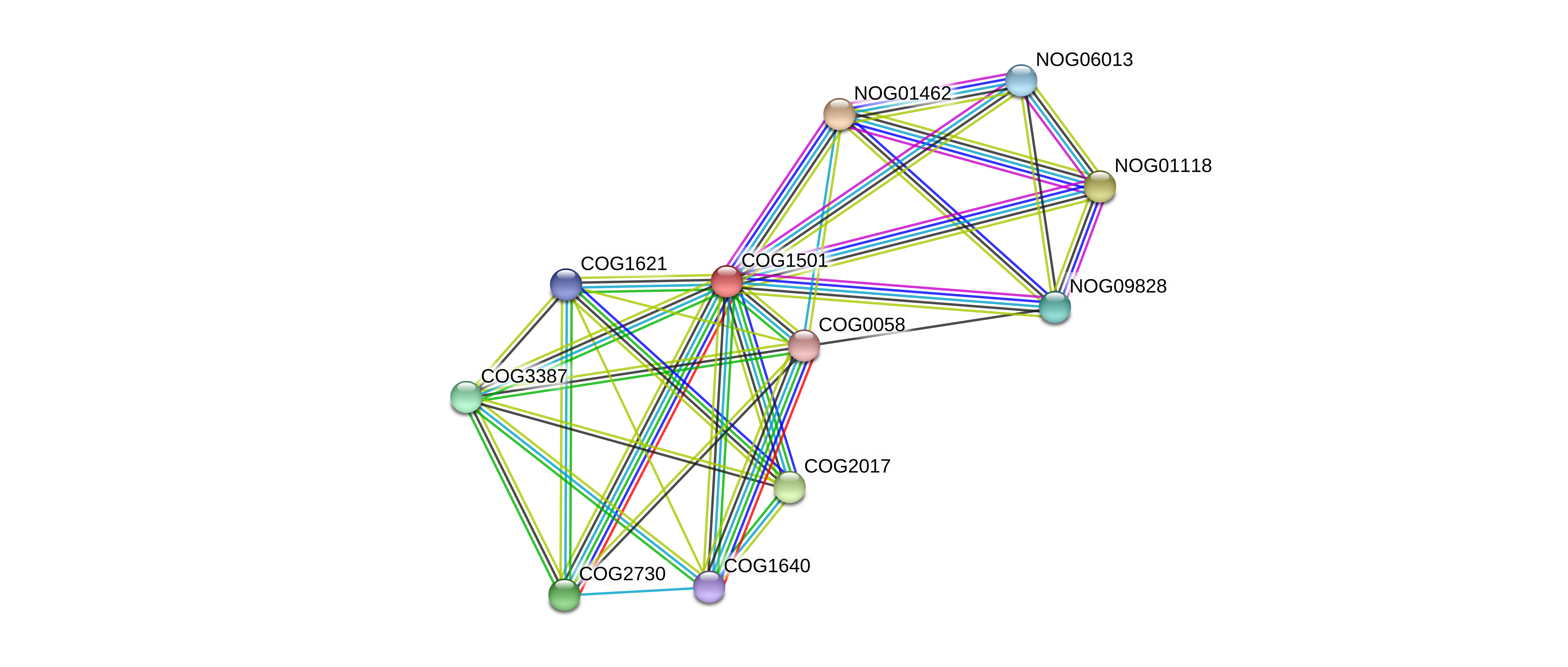
Рис.1. Визуализация геномного окружения COG с помощью программы STRING
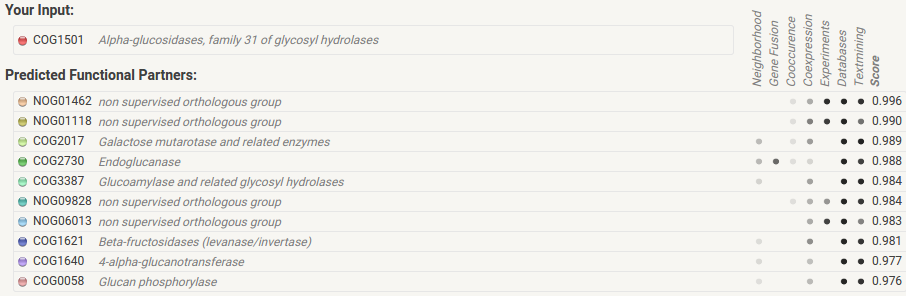
Рис.2. Найденные КОГи и их взаимодействия
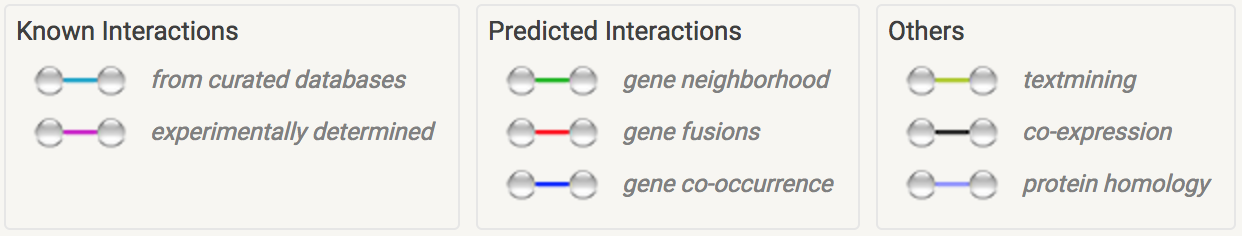
Рис.3. Легенда карты взаимодействий
Рис.1 - изображение графа, на котором отмечен наш COG1501, соединенный с другими COGами и NOGами (NOG - non-supervised orthologous groups, т.е., автоматически определенные группы ортологов, для которых не получено более подробного подтверждения ортологичности). Cвязи (см. рис.3) обозначены следующим образом: известные отношение обозначены голубым (представлены в курируемых базах данных) и пурпурным (экспериментально определены). Предсказанные обозначения обозначены так: зеленым обозначено соседство COGов в геноме, красным - слияние генов, синим - совместную встречаемость. Цвет хаки обозначает совместное упоминание белков в PubMed, сиреневым обозначены гомологичные белки, черным - коэкспрессируемые.
На рисунке 2 отношения представлены в виде списка.
Несмотря на функциональную связь (все COGи в списке относятся к списку G, см таблицу 1), вряд ли возможно утверждать, что у GOG1501 есть консервативное геномное окружение, поскольку даже наиболее представленный по соседству COG2730 (эндоглюконазы) присутствует рядом с генами из COG1501 далеко не всегда и между ними обычно присутствуют другие гены (см. рис.4), также об этом свидетельствует низкий рейтинг соседства в геноме (score 0.253).

Рис.4. Консервативное геномное окружение COG1501.
Отнесение белка F0S4X2_PSESL из Pseudopedobacter saltans к терминам GO
С помощью AmiGO BLAST был найден наиболее похожий белок: альфа-ксилозидаза из E.coli K-12, e-value находки 2.1e-32. BLAST нашел два выравниваемых фрагмента этого белка, в обоих 25% аминокислот идентичны, в первом функционально консервативны 41%, во втором 46%.
Таблица 2. Термины GO, отнесенные к белку с идентификатором Uniprot P31434 (XYLS_ECOLI).
| Аспект | Идентификатор GO | Название термина | Перевод названия термина | Код типа достоверности |
| Молекулярная функция (Molecular function) | GO:0080176 | xyloglucan 1,6-alpha-xylosidase activity | 1,6-альфа-ксилосидазная активность по отношению к ксилоглюкану | IDA |
| Молекулярная функция (Molecular function)< | GO:0042802 | identical protein binding | идентичное связывание белков | IDA |
Таблица 3. Описание кодов достоверности, использованных в Таблице 1.
| Код типа достоверности | Расшифровка кода типа достоверности | Объяснение |
| IDA | Inferred from Direct Assay (основано на прямом анализе) | Используется тогда, когда аннотация была получена при прямом экспериментальном анализе естественной функции белка. В случае, когда функции исследуется с помощью получения мутантного фенотипа, например, с помощью сверхэкспрессии, используется код IMP. IDA является кодом достоверности с высокой надежностью. |
Так как IDA является кодом высокого уровня достоверности аннотации, о функциях тех или иных белков при сравнении с XYLS_ECOLI можно судить достаточно достоверно. Как и первоначальная альфа-глюкозидаза, XYLS_ECOLI относится по функции к списку G (см. Таблицу 1), что подтверждает этот вывод.