Миниобзор бактерии Pussilibacter faecalis
ВВЕДЕНИЕ
Таксономия Pusillibacter faecalis[1]:
Надцарство: Bacteria
Царство: Bacillati
Тип: Bacillota
Класс: Clostridia
Отряд: Eubacteriales
Семейство: Oscillospiraceae
Род: Pusillibacter
Pusillibacter faecalis - грамотрицательная бактерия, палочка или палочковидная, культивируется на строго анаэробной среде с GAM агаром, колонии точечные или дисковидные, светло-серые полупрозрачные, не образуют на ней споры и неподвижны[2]. Данный вид бактерии был выделен из фекалий человека, больного раком желудка[3].
МЕТОДЫ
Подсчет количества белков с различными длинами
В таблице CDS from Pussilibacter faecalis (см. приложение 1) есть колонка Length, с помощью которой и функций Google sheets были подсчитаны длины белков, и для каждого диапазона с шагом 100 было подсчитано количество белков, длины которых лежат в данном диапазоне.
Подсчет генов с различным %GC
В таблице CDS from Pussilibacter faecalis (см. приложение 1) есть колонка %GC, для каждого диапазона с шагом 5, было подсчитано количество генов с %GC, которые лежат в данных диапазонах.
Подсчет старт-кодонов при помощи bash
Для 3.3 был написан скрипт на bash (см. приложение 2) и возможности Google Sheets. Скрипт работает с файлом формата fasta, который вырезает по две строки после каждого названия гена для CDS, pseudo- и “нормальных” генов. Затем берет вторую строку и вырезает по три нуклеотида и строки, которые являются старт-кодоном.
РЕЗУЛЬТАТЫ
Гистограмма длин белков
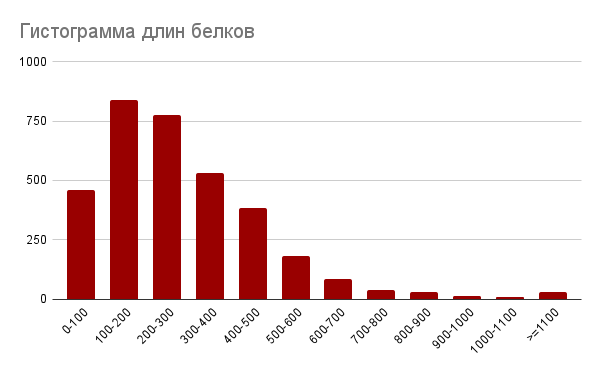
Рис. 1Гистограмма длин белков Наибольшее количество белков у Pusillibacter faecalis с длиной 100-300 аминокислот (рис. 1). Из гистограммы видна зависимость, что количество более длинных белков после 100 аминокислот уменьшается.
Гистограмма %GC в различных CDS
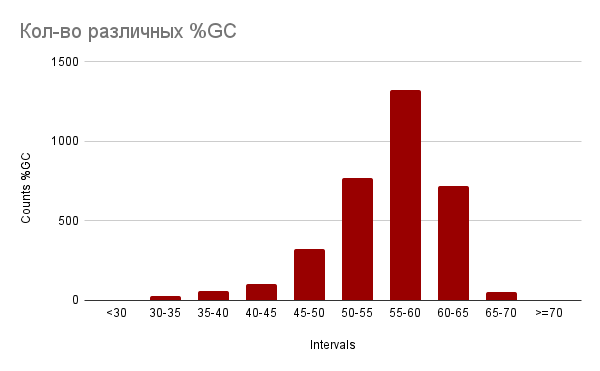
Рис. 2. Гистограмма по GC% в различных генах На гистограмме (см. приложение 1, лист gc_hist) видно, что больше всего CDS с %GC в пределах от 50 до 65%, особенно 55-60%. Можно сказать, что гуанин и цитозин немного превалируют в составе генома бактерии. Возможно увеличение %GC может свидетельствовать о том, что бактерия живет в кишечнике человека, в котором среда более агрессивная, чем, например, в пресном водоеме.
Количество старт-кодонов
Таблица 1. Количество старт-кодонов CDS NORMAL pseudogene ATG 2930 2891 39 GTG 252 252 0 TTG 163 161 2 ATT 8 6 2 CTG 8 7 1 ATA 4 4 0 ATC 4 4 0 TCA 3 0 3 TCT 3 0 3 AAG 1 0 1 AGA 1 0 1 CGG 1 1 0 GAG 1 0 1 GGG 1 0 1 GGT 1 0 1 TCG 1 0 1 TTT 1 0 1 По таблице видно, что кроме ATG, превалируют старт-кодоны GTG и TTG в нормальных генах. ATG во всех CDS 86,6%, GTG - 7,4%, TTG - 4,8%. Стоит отметить, что у них отличается только первый нуклеотид. Если посмотреть на строение нуклеотидов аденина, тимина и гуанина, можно примерно сказать, что могло привести к их изменению. Старт-кодонов GTG больше, чем старт-кодонов с тимином, оно и логично - аденин и гуанин оба пуриновые основания, для изменения аденина в гуанин нужно меньше затрат энергии, хоть и скорее это изменение связано с мутацией. Больше интересно то, что тРНК считывает такой кодон как старт-кодон, даже если основания похожи, они не полностью совпадают, просто так тРНК не может считывать GTG, как ATG. С TTG ещё сложнее, тимин это нуклеотид с пиримидиновым основанием, оно вообще не похоже на аденин.
Расположение и наименования tRNA и rRNA
Как видно из таблицы (см. приложение 3) в основной хромосоме 16S, 5S и 23S rRNA сгруппированы в одном месте и на + цепи и на - цепи. Также в плазмиде pMM59_01 на - цепи эти три rRNA находятся очень близко, но на + цепи их нет. Сложно сказать, с чем это связано. tRNA гораздо больше (см. приложение 4), чем rRNA, и все tRNA находятся в разных местах хромосом от 1 до 9 генов. Некоторые гены tRNA находятся в нескольких местах, возможно потому, что один белок могут кодировать разные кодоны, для которых нужны разные антикодоны. Также есть два tRNA-OTHER, которые возможно либо неспецифичны, либо занимаются транспортом других молекул.
ПРИЛОЖЕНИЯ
БЛАГОДАРНОСТИ
Хочу поблагодарить весь преподавательский состав за обучение методам, которые я применил в написание этого мини обзора и моего однокурсника Антона Куликова за помощь с генерацией идеи для пункта в мини обзоре.
СПИСОК ЛИТЕРАТУРЫ
- https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?mode=Info&id=2714358
- Kitahara, M., Shigeno, Y., Shime, M., Matsumoto, Y., Nakamura, S., Motooka, D., Fukuoka, S., Nishikawa, H., and Benno, Y. "Vescimonas gen. nov., Vescimonas coprocola sp. nov., Vescimonas fastidiosa sp. nov., Pusillimonas gen. nov. and Pusillimonas faecalis sp. nov. isolated from human faeces." Int. J. Syst. Evol. Microbiol. (2021) 71(11):005066.
- Kitahara M, Shigeno Y, Shime M, Matsumoto Y, Nakamura S, Motooka D, Fukuoka S, Nishikawa H, Benno Y. Corrigendum: Vescimonas gen. nov., Vescimonas coprocola sp. nov., Vescimonas fastidiosa sp. nov., Pusillimonas gen. nov. and Pusillimonas faecalis sp. nov. isolated from human faeces. Int J Syst Evol Microbiol. 2022 Apr;72(4). doi: 10.1099/ijsem.0.005317. Erratum for: Int J Syst Evol Microbiol. 2021 Nov;71(11). doi: 10.1099/ijsem.0.005066. PMID: 35442879..