Я выбрала домен PF00580 (UvrD/REP helicase N-terminal domain)
В seed выравнивании 36 последовательностей. В SwissProt 205 белков.
Польностью одинаковых последовательностей не нашлось, но при Above identity threshold= 85 осталось 32 штуки
Нашла мотив очень похожий на Walker A
Walker A мотив, также известный как P-loop, является консервативным структурным элементом белков, который отвечает за связывание фосфатных групп молекулы ATP

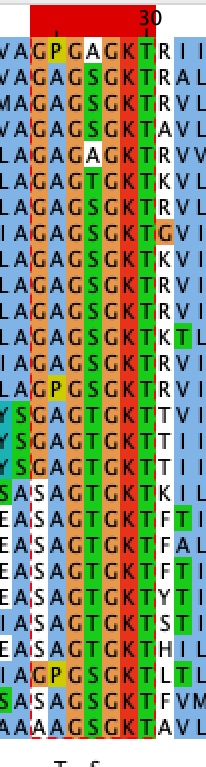
Паттерн в Jalaview G.G.GKT
Нашлось 22 совпадения. Дважды в одной поседовательности не нашлось
Паттерн для Prosite: G-x-G-x-G-K-T
Я провела поиск в SwissProt PROSITE и обнаружила 1000 находок в 1000 последовательностях
Поставила Above identity threshold 90 и из 1000 осталось 395, сделала выравнивание
С помощью поиска в jalview нашла 390 совпадений
С помощью NJ в Jalview я построила дерево
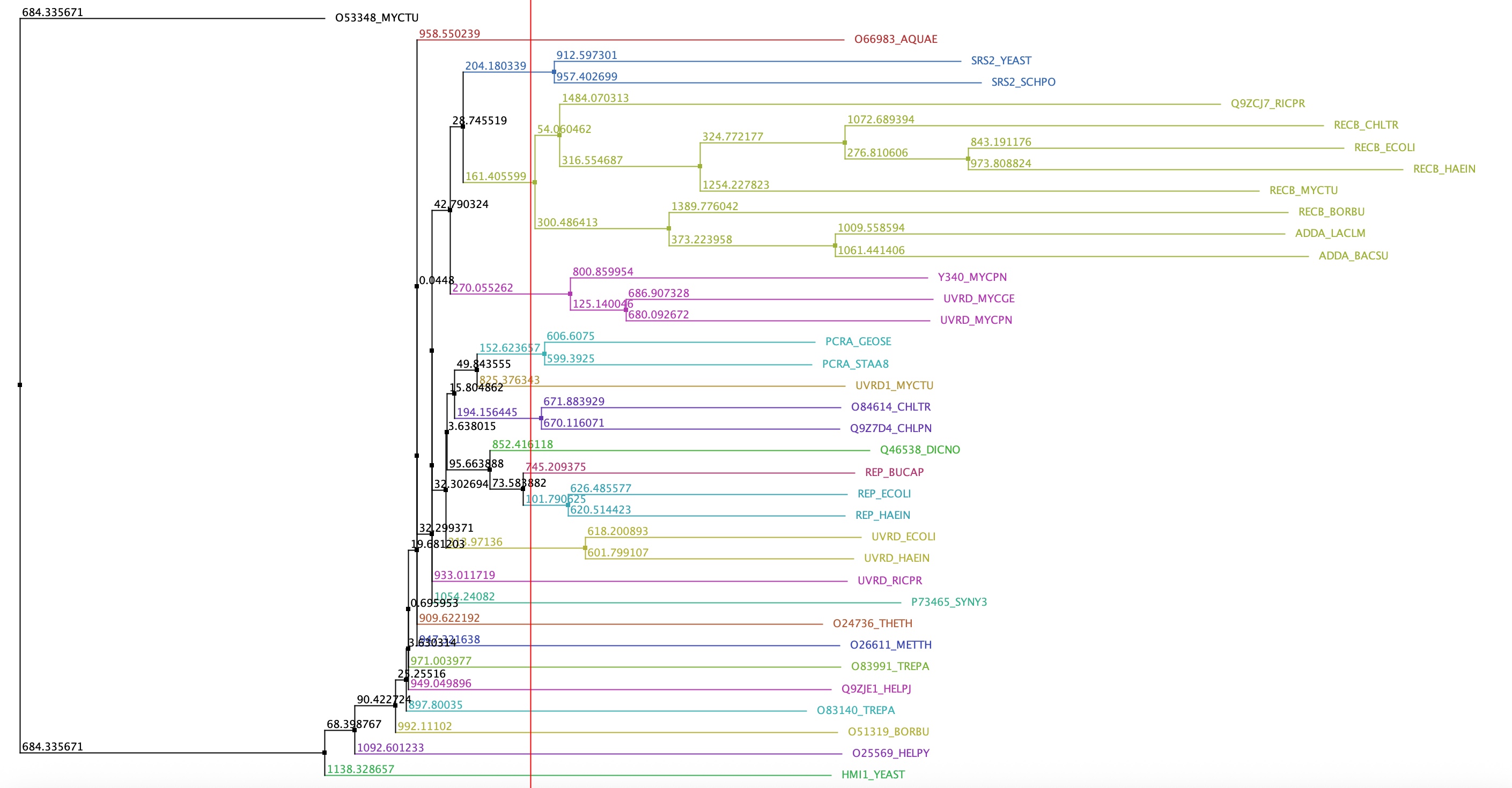
Я решила выбрать кладу состаящую из Q9ZCJ7_RICPR, RECB_CHLTR, RECB_MYCTU, RECB_ECOLI, RECB_HAEIN, RECB_BORBU, ADDA_BACSU, ADDA_LACLM
Нашла 2 мотива, но выбрала наиболее точный: ASAG[TS]GKT
В результате поиска по всему выравниванию данный мотив нашелся только в 8 случаях. Вероятно данный мотив специфичен для этой клады
Я выбрала P19954 (Ribosome-binding factor PSRP1)
Организм: Spinacia oleracea
Фактор связывания рибосом участвует в свето- и температурно-зависимой регуляции синтеза белка. Взаимодействует с нуклеотидами 16S мРНК в А- и Р-сайтах, где защищает центр декодирования и ингибирует трансляцию, предотвращая связывание тРНК
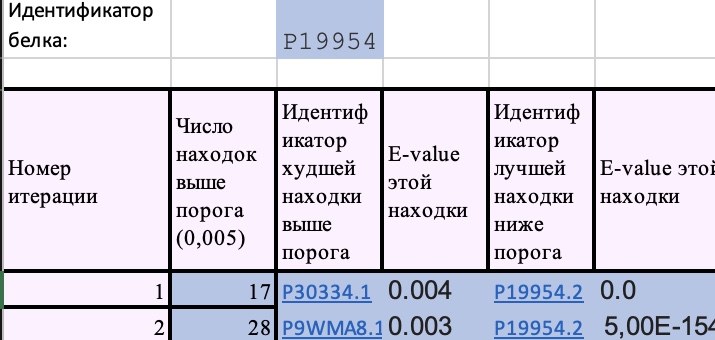
Все найденные белки относятся к белкам, взаимодействующим с рибосомой или участвующим в регулировании трансляции. Большая разница для E-value лучшей и худшей находки
Значит PSI BLAST хорошо нашел гомологи
Скачала 75 последовательностей из SwissProt
Использовала команду
meme idk1.fasta -o res -mod anr -minw 4 -maxw 8 -nmotifs 4
Результаты:
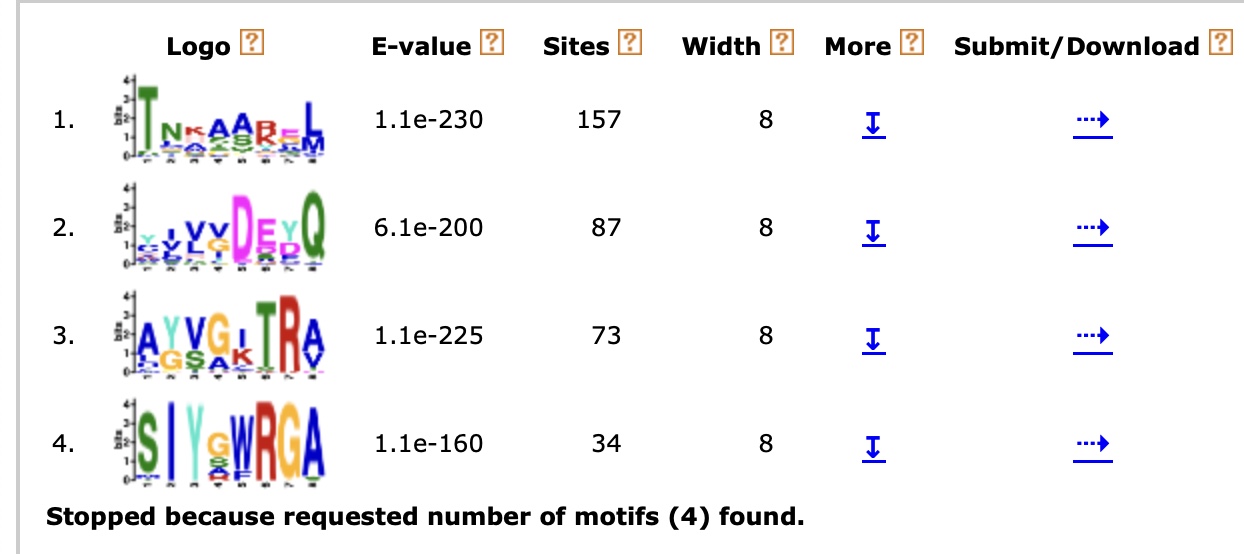
Нашлось 4 мотива
Далее была использована команда: fimo --oc fimo_do -motif TNKAAREL -thresh 0.001 res/meme.txt idk1.fasta
Мотив встречается 198 раз в 43 последовательностях
Моя бактерия - это Pseudoalteromonas piscicida
В файле sites.txt содержатся все возможные перестановки букв G, A, T, C
Я использовала следующую команду:
cbcalc -s sites.txt -M -o res.tsv GCF.fna
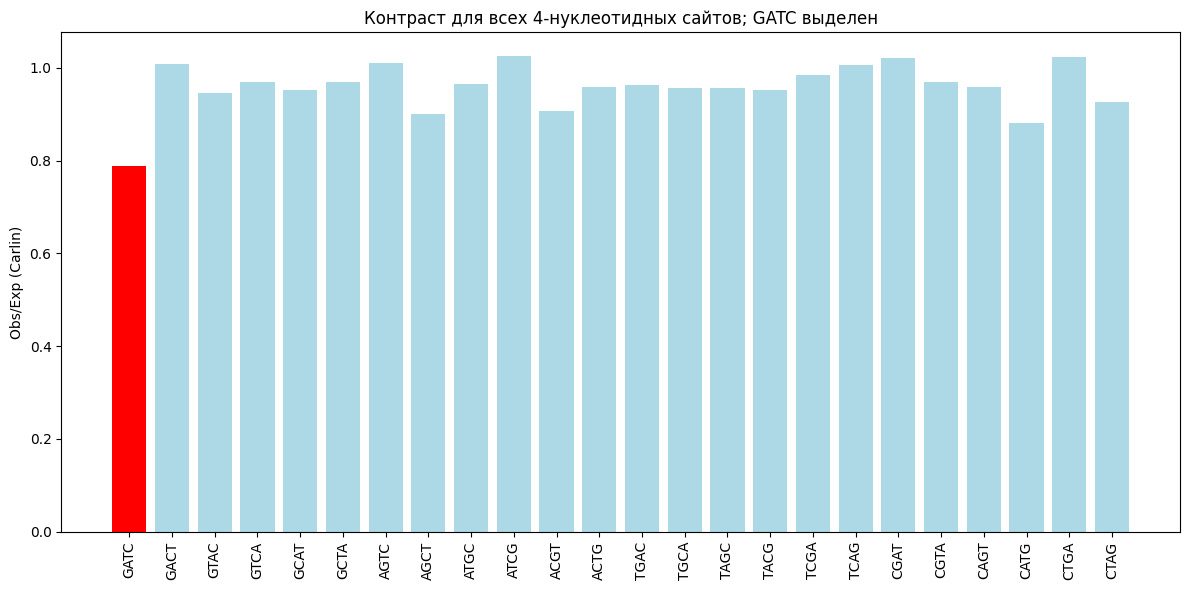
GATC меньше, чем ожидалось. Возможно из-за того, что GATC метилируется