
Я взяла протеомы со следующими мнемониками: AROAE, BARHE, BORPE, ECOLI, PSEAE, RHIME, YERPE
Записала последовательности в файл pr.fasta, произвела поиск программой blastp гомологов с порогом на E-value 0,0001. Использованные команды:
cat AROAE.fasta BARHE.fasta BORPE.fasta ECOLI.fasta PSEAE.fasta RHIME.fasta YERPE.fasta > pr.fasta
makeblastdb -dbtype prot -in pr.fasta -out col
blastp -query P0A6H1.fasta -db col -evalue 0.0001 -out result.txt
Выдача BLAST

Я изменила название находок на их мнемоники, выровняла методом muscle и построила дерево программой fastme с бутстрепом 100
Укоренила дерево в среднюю точку
Файл с формулой newick
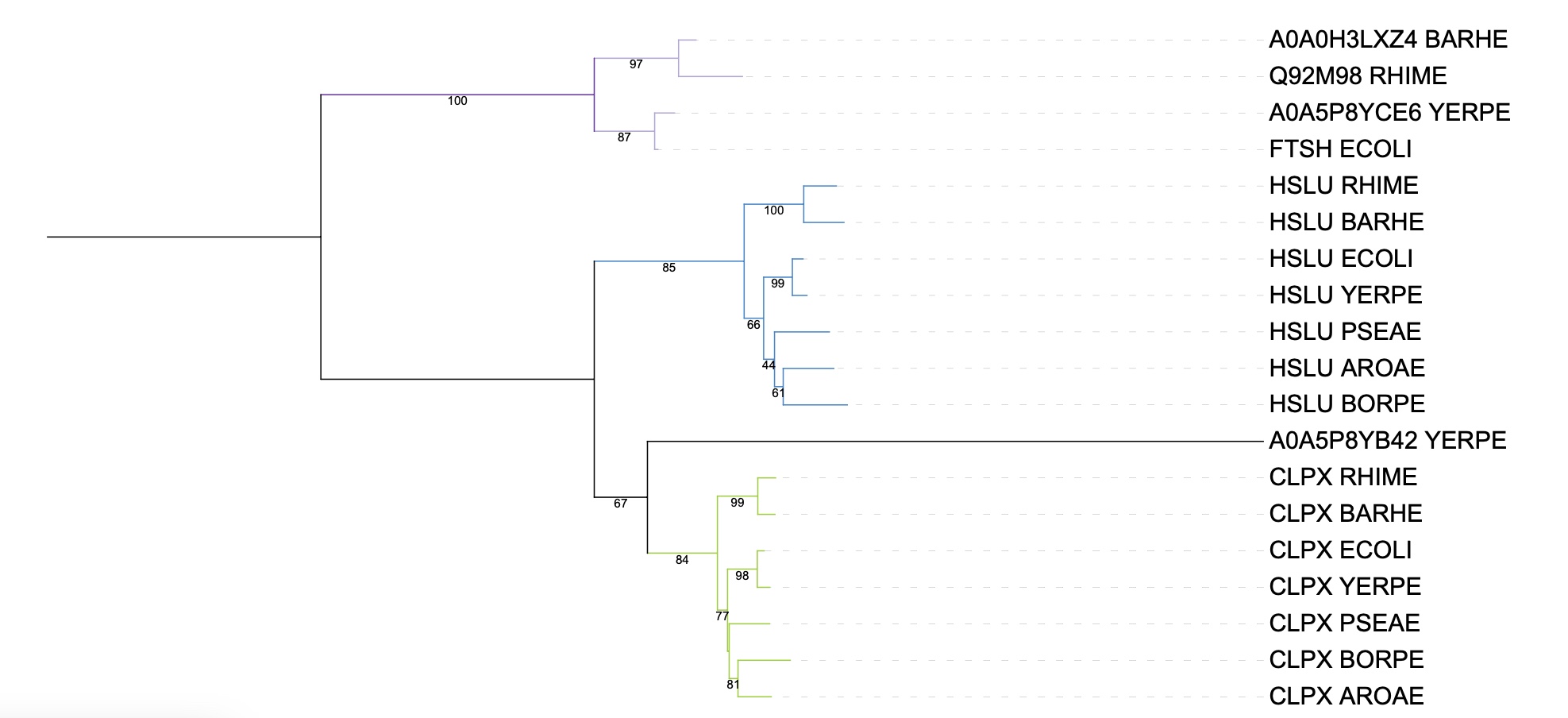
Примеры ортологов:
HSLU RHIME HSLU BARHE
HSLU ECOLI HSLU YERPE
HSLU AROAE HSLU BORPE
Примеры паралогов:
CLPX BARHE HSLU BARHE
CLPX RHIME HSLU RHIME
CLPX PSEAE HSLU PSEAE
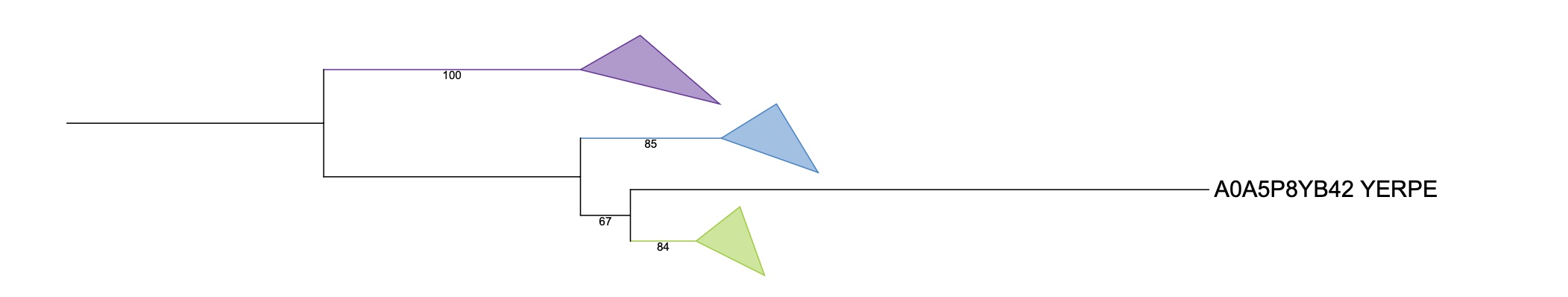
Описание состава CLPX
Включает в сябя все 7 выбранных организма
PSEAE должен быть в одной кладе с ECOLI и YERPE
Описание состава HSLU
Включает в себя все 7 выбранных организмов
PSEAE должен быть в одной кладе с ECOLI и YERPE
Описание состава FTSH
Включает в себя 4 организма: ECOLI, BARHE, RHIME, YERPE