Матрица аминокислотных замен - это матрица, в которой по строке и столбцу , в единственном порядке, расположены 20 незаменимых аминокислот.
В ij месте пересечения столбца и строки находится число. Это число - вес аминокислотной замены, показывающий примерную оценку замен одной
аминокислоты на другую. Оно получается сходя из статистических данных базы BLOKS о встречаемости замени и возможности сдвига(замен на геп)
в биологических организмах.
Матрица BLOSUM не является применимой для всех белков, в основном только для цитоплазматических (т.к. в BLOKS собранны в большинстве своем
цитоплазматические белки). Для мембранных белков существуют специальные матрицы такие как: PHAT (predicted hydrophobic and transmembrane).
Как ни странно, но различия в замене аминокислот между PHAT и BLOSUM заметно хорошо прослеживаются.
За долгое время информация базы данных BLOKS не раз успела обновиться.
После реконструкции BLOSUM62 по последним данным из BLOKS, это особо хорошо прослеживается.
Таблица BLOSUM
| Замена аминокислоты (Glu, E) | Gly | Pro | Cys | Ser | Thr | Asn | Gln | Asp | Glu | His | Arg |
Lys | Ala | Met | Ile | Leu | Val | Phe | Trp | Tyr |
| myBLOSUM62 | -2 | -1 | -3 | -1 | -1 | 0 | 6 | 0 | 2 | 1 | 1 | 1 | -1 | -1 | -2 | -2 | -2 | -2 | -2 | -2 |
| BLOSUM62 | -2 | -1 | -3 | 0 | -1 | 0 | 5 | 0 | 2 | 0 | 1 | 1 | -1 | 0 | -3 | -2 | -2 | -3 | -2 | -1 |
| PHAT_T75_B73 | -2 | -3 | -5 | -1 | -3 | 2 | 9 | 0 | 1 | 2 | -2 | -1 | -3 | -1 | -3 | -3 | -3 | -2 | 1 | 0 |
Алгоритм Нидлмана-Вунша предназначен для гловального парного выравнивания, то есть он сравнивает две последовательности целиком.
В случае,когда одна последовательность меньше другой, эта разница заполняется гэпами. Этот алгоритм реализуется программой needle.
Алгоритм Смита-Ватермана используется при локальном парном выравнивании, то есть для участков, оптимальных по весу. Его выполняет команда water.
Gap open penalty по умолчанию равен 10
Gap extension penalty по умолчанию равен 0,5
| Мутант | Выравнивание вручную | Выравнивание Нидлман-Вунш | Выравнивание Симт-Вотерман |
| 1 |  |  | 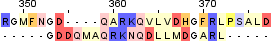 |
Identity: 11/21 (52%)
Similarity: 13/21 (62%)
Score: 54 | Identity: 8/665 (1.2%)
Similarity: 10/665 (1.5%)
Score: 22.5 | Identity: 8/20 (40.0%)
Similarity: 10/20 (50.0%)
Score: 22,5 |
| 2 |  |  | 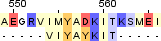 |
Identity: 11/21 (52%)
Similarity: 11/21 (52%)
Score: 39 | Identity: 10/661 (1.5%)
Similarity: 10/661 (1.5%)
Score: 23 | Identity: 5/8 (62.5%)
Similarity: 7/8 (87.5%)
Score: 26 |
| 3 | 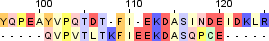 |  |  |
Identity: 12/20 (60%)
Similarity: 12/20 (60%)
Score: 45 | Identity: 12/663 (1.8%)
Similarity: 12/663 (1.8%)
Score: 25 | Identity: 11/15 (73.3%)
Similarity: 11/15 (73.3%)
Score: 29 |








