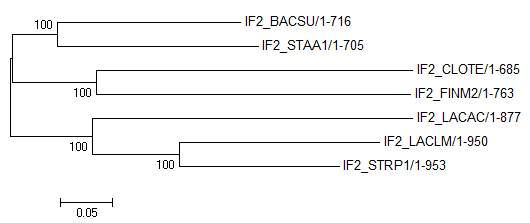
Дерево семи выбранных видов бактерий, построенное Jalview по фактору инициации трансляции 2 (IF2)
с использованием алгоритма Neighbour joining tree using blosum62. Для того, чтобы укоренить это
дерево в среднюю точку, использовалась программа retree пакета Phylip.
Укоринением в среднюю точку, мы принимаем гипотезу о молекулярных часах, для построения графа
с различной длиной ветвей. Исходное и полученное деревья были визуализированы в MEGA.
| Исходное дерево | Укорененное в среднюю точку дерево |

|
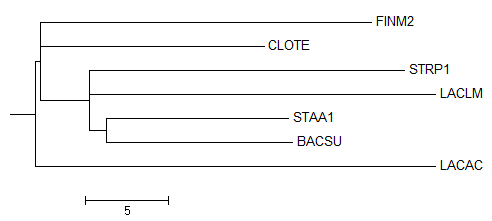
|
- Видно, что дерево было укоренено верно.
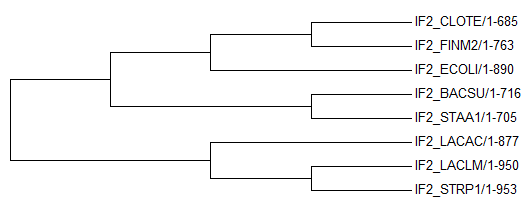
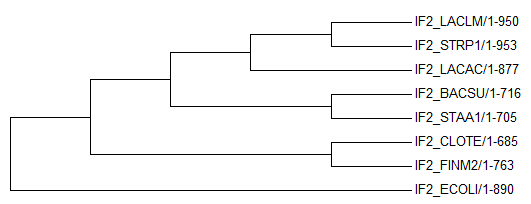
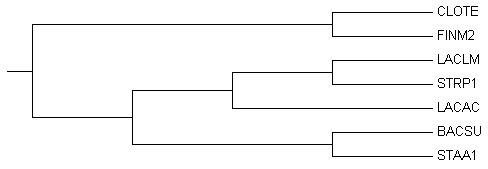 Исходное дерево.
Исходное дерево.