Построим филогенетическое дерево тех же бактерий, что в
предыдущем задании,
используя последовательности РНК малой субъединицы рибосомы (16S rRNA). Чтобы получить
последовательности 16S рибосомальной РНК каждой из бактерий, воспользуемся
записями EMBL, описывающими полный геном бактерии. Нужный же участок из EMBL
вырежем с помощью seqret. Соответствующая таблица:
|
Мнемоника
|
AC EMBL
|
Координаты
|
Цепь
|
|
BACSU
|
AL009126
|
9810-11364
|
+
|
|
CLOTE
|
AE015927
|
8715-10223
|
+
|
|
FINM2
|
AP008971
|
197837-199361
|
+
|
|
LACAC
|
CP000033
|
59255-60826
|
+
|
|
LACLM
|
CP002365
|
470580-472133
|
+
|
|
STAA1
|
CP003808
|
540419-542060
|
+
|
|
STRP1
|
AE004092
|
17170-18504
|
+
|
Нуклеотидные последовательности были собраны в одном
файле. Теперь произведем
выравнивание с помощью MUSCLE.
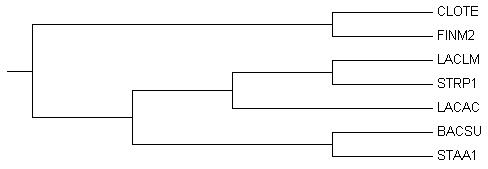
Реконструируем дерево в MEGA с помощью метода "Maximum Likelihood".
| Исходное дерево |
Реконструированное дерево |
 |
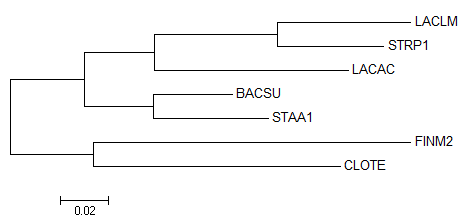 |
Как видно из таблицы, полученное дерево полностью совпадает с парвильным.
Два гомологичных белка мы будем называть ортологами, если они были получены из
разных организмов или разделение их общего предка на линии, произошло в результате
видообразования. А паралогами будем называть гомологичными белка из одного организма.
С помощью программы blastp с использованием файла с последовательностями белков
бактерий в качестве базы данных были найдены последовательности гомологов
белка CLPX_BACSU среди изучаемых бактерий. Соответствующие команды:
seqret sw:clpx_bacsu
makeblastdb -in proteo.fasta -out proteo -dbtype prot
blastp -query clpx_bacsu.fasta -db proteo -evalue 0.001 -out clpx_blastp.out -outfmt 6
Из полученного файла
отберем гомологи, относящиеся к выбранным организмам (а также уберем повторяющиеся белки).
Теперь воспользуемся последним файлом и сервисом Retrieve на сайте Uniprot,
чтобы получить нужные последовательности, которые выровняем
с помощью Muscle.
Наконец, методом Neighbor-Joining построим соответствующее дерево в MEGA.

На полученном дереве CLPX_CLOTE, CLPX_BACSU, CLPX_STAA1, CLPX_LACLM и CLPX_STRP1, например, являются ортологами,
а CLPC_BACSU, CLPE_BACSU, CLPY_BACSU, и CLPX_BACSU - паралогами.


