Построим множественное выравнивание отобранных гомологов с помощью Muscle. Для этого сначала
открыли fasta-файл в JalView.
Добавили новую пустую строку аннотации и назвали ее "TM_REAL".
Переместили исходный белок, для которого есть структура, в верхнюю строку выравнивания.
Прикрепили к нему структуру и, используя появившуюся связь между последовательностью и
структурой, пометили участки выравнивания, отвечающие трансмембранным спиралям в белке
со структурой в строке-аннотации буквой "М".
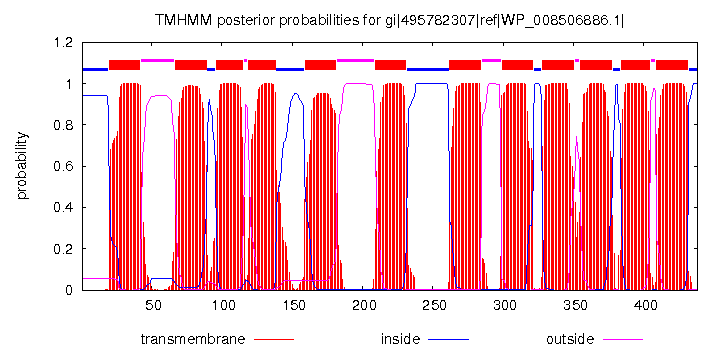
Добавили к выравниванию предсказание трансмембранных спиралей, выдаваемых программой TMHMM,
для гомолога WP_008506886.1| fucose permease [Mucilaginibacter paludis].
Взяли последовательность гомолога и получили для него результат предсказания TMHMM.
Ниже приведено изображение, выданное программой TMHMM для соответствующего гомолога:
Создали новую строку аннотации и назвали ее "TM_PREDICTED", после чего нанесли вручную участки,
предсказанные TMHMM, в эту строку.
Выбрали цветовую схему, позволяющую визуально различать гидрофобные и гидрофильные остатки.
Затем установили "By Conservation" (20%).
полученный файл в формате JAR
Покрутим белок так, чтобы его часть, ориентированная в n-сторону мембраны оказалась снизу,
а ориентированная в p-сторону - сверху. Ниже приведено соответствующее изображение:

Выбранная цветовая схема окрашивает более гидрофильные остатки
синим цветом, а более гидрофобные - красным. Порог консервативности - 20%

Изображение с полным выравниванием репрезентативной выборки
Как оказалось, в данном белке относящиеся к трансмембранным спиралям участки крайне консервативны,
а консервативность участков между самими спиралями ниже.
В трансмембранных спиралях есть как заряженные, так и полярные полярные остатки.
Положение предсказанных альфа-спиралей на выравнивании почти полностью совпадает с реальными, отмеченными для исследуемого белка.
Я считаю, что небольшие различия могут быть вызваны тем, что в белке много гидрофобных аминокислот,
которые затрудняют определение трансмембранных участков.