Для дальнейшего изучения эволюции архитектур, включающих домен RGS, были выбраны следующие (первые две):
- RGS
 (1737 последовательностей)
(1737 последовательностей) - RGS+Pkinase
 (369 последовательностей)
(369 последовательностей)
Был выбран домен RGS (информация в Pfam) (Регуляторы каскада G белков (RGS) - активирет ГТФазу для альфа-субъединицы гетеромерного G-белка, тем самым инактивируя G-белок.
В Jalview было получено выравнивание всех последовательностей Pfam домена RGS в форматах
JAR. Выравнивание было раскрашено по консервативности
(Clustalx, By conservation с порогом консервативности 10%).
Также к поледовательности ARBK1_BOVIN была добавлена 3D-структура структура 1YM7
ссылка на PDB.
С использованием скрипта swisspfam_to_xls.py и файла,
содержащего информацию об архитектуре всех последовательностей UniProt, была получена таблица
с информацией об архитектуре последовательностей, содержащих домен RGS.
Затем в таблицу были добавлены колонки с информацией о таксономической принадлежности.
Для этого по идентификаторам отобранных последовательностей в UniProt были получены AC,
по которым были получены файлы в формате UniProt (Retrieve),
которые были использованы для работы скрипта uniprot-to-taxonomy.py.
Также был добавлен столбец, содержащий информацию о длине доменов RGS и Pkinase.
Полученная таблица.
Для дальнейшего изучения эволюции архитектур, включающих домен RGS, были выбраны следующие (первые две):
 (1737 последовательностей)
(1737 последовательностей) (369 последовательностей)
(369 последовательностей)
В Metazoa были выбраны два подтаксона - филумы Chordata и Ecdysozoa.
Представители выбраны при работе с таблицей (лист PivotTable со сводной таблицей, а весь список выбранных - лист selected_seqs).
Для каждой архитектуры было отобрано по 28 последовательности (по 14 из каждого таксона).
Чтобы оставить в выравнивании нужные последовательности из двух групп был использован скрипт
filter-alignment.py.
Полученное выравнивание в формате FASTA.
Оно же и было загружено в JalView (пустые колонки были удалены).
Затем в нём были выделены группы согласно архитектуре, в каждой из них
была выполнена раскраска последовательностей ClustalX, Conservation
(порог на консервативность 10%). После удаления некоторых последовательностей (15),
удаления пустых колонок и несодержательных C- и N- концевых участков в конечном
файле выравнивания JalView содержится 41 последовательность. Скачать в формате
JAR.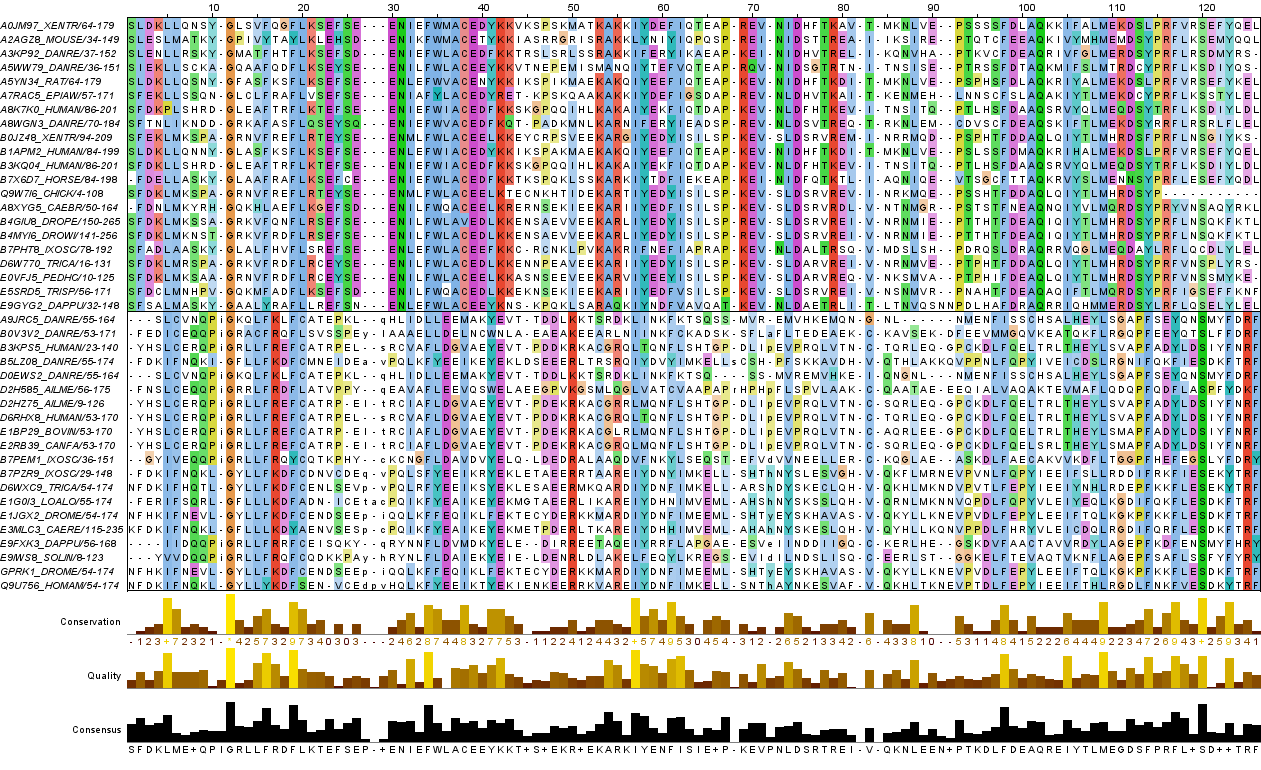
Названия последовательностей были заменены кодом и видовым названием.
Буква кода указывает на подтаксон, цифра - на количество доменов в архитектуре.
Видоизмененное выравнивание в формате FASTA.
Затем по этому выравниванию методом maximal likelihood было постороено филогенентическое дерево, использовался bootstrap.
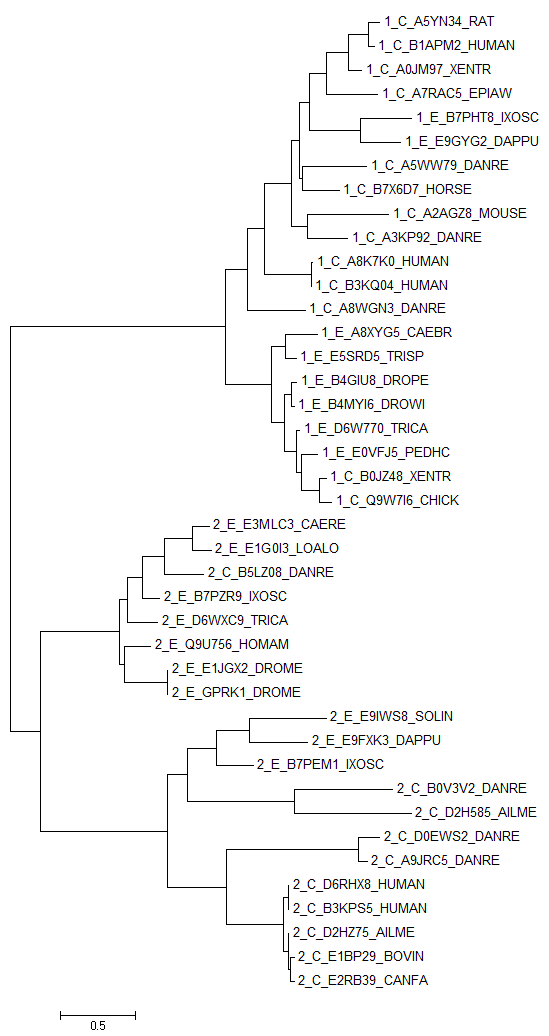
Можно заметить, что однодоменные и двухдоменные архитектуры образуют две большие клады.
В процессе эволюции, вероятнее всего, произошла делеция одного домена в корне, что
является причиной разделения на две такие большие клады.