С помощью PDBeFold были найдены структуры, гомологичные 1vi9 и были выбраны
четыре из них: 3keu, 2yxu, 3fhy, 3pzs.
Полученный файл можно скачать.
Пространственное совмещение структур было выполнено непосредственно в Jmol. Результат показан на рисунке ниже:

Стоит отметить, что хоть последовательности белков были выбраны не самые идентичные,
структуры достаточно хорошо совмещены.
Далее с помощью сервиса T-Coffee
и имеющихся последовательностей было получено
множественное выравнивание указанных белков.
Также было получено структурное выравниваниевыбранных последовательностей.
На рисунке ниже приведены кликабельные изображения из проектов в JalView, которые тоже можно
скачать.


На картинке ниже можно заметить, что выравнивания практически полностью совпадают:

Далее в PDBeFold осуществили поиск структурных гомологов домена 1dek A: 33-154. Использовали параметры по умолчанию:
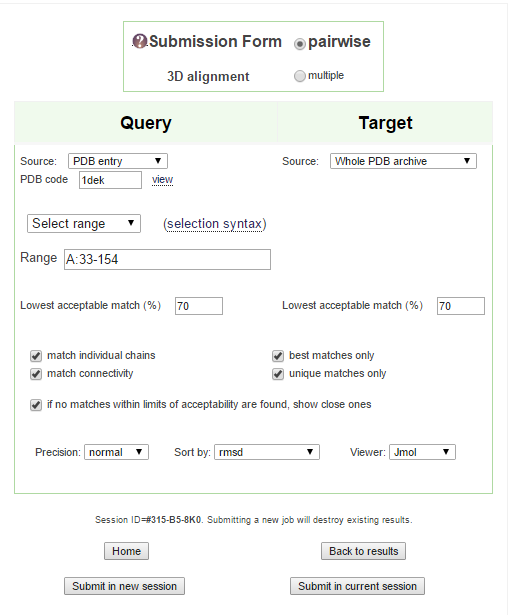
Затем выдача была отсортирована по RMSD. Из 35 структур исходного домена не оказалось.
Связано это с тем, что параметры по умолчанию требуют того, чтобы процент совпадения структур был не меньше 70, в то время как
искомый домен занимает меньше 70% общей длины белка. Именно поэтому белок и не находится.
Переходим к совмещению структур альфа- и бета- цепочек константного домена T-клеточного рецептора.
Была выбрана первая в списке структура Т-клеточного рецептора 1OGA: альфа-цепь - region d:118-202, бета-цепь - region e:119-245 и
скачаны соответствующие файлы:
1oga_d.pdb и
1oga_e.pdb
С помощью Sheep были построены карты бета-листов для альфа- и бета- цепочки, соответственно:


Видно, что бета-листу альфа-цепочки соответствует первый бета-лист бета-цепочки, так как они изображены в одной ориентации.
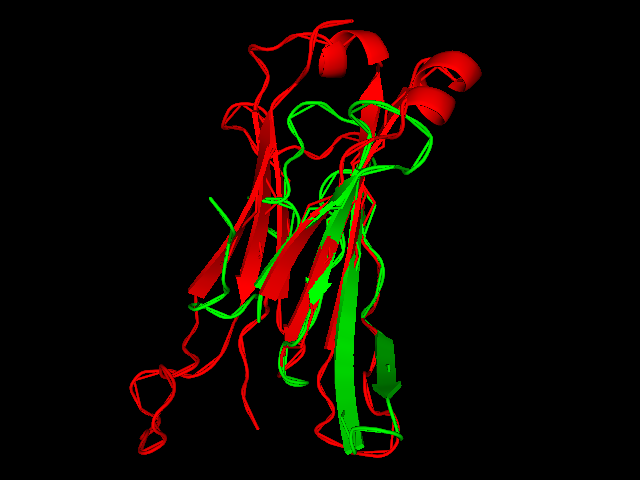
В Pymol было построено совмещение цепочек с помощью следующих команд. Ниже приведено соответствующее изображение
, где структура из цепи альфа представлена зеленым, а цепи бета красным: