Из четырех деревьев выданных программой выбрал наименее "ошибочное" - (((RPOZ_BACSU,RPOZ_BACAN),((RPOZ_STAES,RPOZ_STAA1),((RPOZ_PEDPA,
RPOZ_LACDA),RPOZ_STRP1))),RPOZ_THETN).
+--RPOZ_BACSU
+--------------7
! +--RPOZ_BACAN
!
+--6 +--RPOZ_STAES
! ! +--------5
! ! ! +--RPOZ_STAA1
! +-----4
! ! +--RPOZ_PEDPA
1 ! +--3
! +-----2 +--RPOZ_LACDA
! !
! +-----RPOZ_STRP1
!
+--------------------RPOZ_THETN
| присутствует в оригинальном дереве, но нет в полученном |
присутствует в полученном, но отсутствует в оригинальном |
| ((RPOZ_BACSU,RPOZ_BACAN),(RPOZ_STAES,RPOZ_STAA1)) |
((RPOZ_STAES,RPOZ_STAA1),((RPOZ_PEDPA,RPOZ_LACDA),RPOZ_STRP1)) |
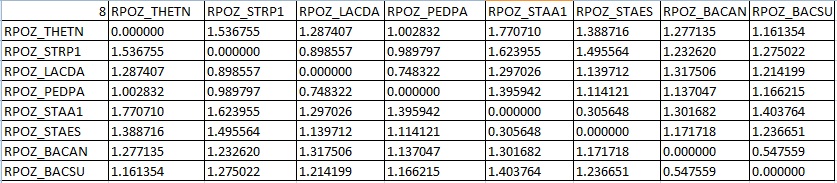
7. Оценка эволюционных расстояний между последовательностями программой fprotdist
d(RPOZ_BACAN,RPOZ_STAES) > d(RPOZ_BACAN,RPOZ_BACSU), d(RPOZ_STAES,RPOZ_BACAN)=d(RPOZ_STAES,RPOZ_BACSU)
1.171718 > 0.547559, 1.171718 == 1.236651, Отклонение = 0,064933
Аддитивность
d(RPOZ_BACAN,RPOZ_BACSU) + d(RPOZ_STAA1,RPOZ_STAES) = 0,853207
d(RPOZ_BACAN,RPOZ_STAA1) + d(RPOZ_BACSU,RPOZ_STAES) = 2,538333
d(RPOZ_BACAN,RPOZ_STAES) + d(RPOZ_BACSU,RPOZ_STAA1) = 2,575482
Отклонение = 0,037149.
8. Реконструкции дерева программой fneighbor. Neighbor-Joining method (команда fneighbor, с входным файлом pr2_mus.fprotdist)
+--------------------------------RPOZ_STRP1
+-----3
+-----4 +--------------------RPOZ_LACDA
! !
! +-------------------RPOZ_PEDPA
!
! +--------------RPOZ_STAA1
! +------------------------------------1
! ! +--RPOZ_STAES
5----6
! ! +----------------RPOZ_BACAN
! +--------------2
! +---------------RPOZ_BACSU
!
+---------------------------------------RPOZ_THETN
Это дерево имеет схожесть, но различается заметно. Нетривиальные ветви:
1){LACDA,PEDPA,STRP1} vs {BACAN,BACSU,STAA1,STAES,THETN}
2){BACAN,BACSU} vs {STAA1,STAES,LACDA,PEDPA,STRP1,THETN}
3){STAA1,STAES} vs {BACAN,BACSU,LACDA,PEDPA,STRP1,THETN}
4){RPOZ_LACDA,RPOZ_STRP1} vs {RPOZ_PEDPA,RPOZ_STAA1,RPOZ_STAES,RPOZ_BACAN,RPOZ_BACSU,RPOZ_THETN} - Эта ветвь отсутствует в оригинальном дереве.
В остальном это хороший результат.
9. Реконструкции дерева программой fneighbor. UPGMA (команда fneighbor -treetype u -trout , с входным файлом pr2_mus.fprotdist)
+------------------------------------RPOZ_THETN
+-5
! ! +----------------RPOZ_BACAN
! +-------------------2
+---6 +----------------RPOZ_BACSU
! !
! ! +---------------------------RPOZ_STRP1
! +--------4
--7 ! +----------------------RPOZ_LACDA
! +----3
! +----------------------RPOZ_PEDPA
!
! +--------RPOZ_STAA1
+-------------------------------1
+--------RPOZ_STAES
Это дерево совершенно не похоже на правильное. Нетривиальные ветви:
1){LACDA,PEDPA,STRP1} vs {BACAN,BACSU,STAA1,STAES,THETN}
2){BACAN,BACSU} vs {STAA1,STAES,LACDA,PEDPA,STRP1,THETN}
3){STAA1,STAES} vs {BACAN,BACSU,LACDA,PEDPA,STRP1,THETN}
4){LACDA,PEDPA} vs {BACAN,BACSU,STAA1,STAES,STRP1,THETN}
5){THETN,BACAN,BACSU} vs {STRP1,LACDA,PEDPA,STAA1,STAES} - эта ветвь отличает дерево от оригинального.
6)
10. TreeTop сервис.
Серверная программа реализовывала два алгоритма: cluster и topological.
В первом случае демонстрировалось дерево и скобочная формула:
(((RPOZ_THETN::0.579550,(RPOZ_LACDA::0.472536,RPOZ_PEDPA::0.472536):0.107014):0.048297,((RPOZ_STAA1::0.243084,RPOZ_STAES::0.243084):0.370875,(RPOZ_BACAN::0.404080,RPOZ_BACSU::0.404080):0.209879):0.013888):0.232268,RPOZ_STRP1::0.860115);
В другом алгоритме результатом надо считать дерево, скобочную формулу
((((RPOZ_STAA1::0.001000,RPOZ_STAES::0.312213):0.196433,(RPOZ_BACSU::0.077688,RPOZ_BACAN::0.325226):0.093944):0.024045,((RPOZ_PEDPA::0.151548,RPOZ_LACDA::0.329375):0.021518,RPOZ_STRP1::0.579367):0.017430):0.167601,RPOZ_THETN::0.167601);
и матрицу расстояний:
DISTANCE MATRIX
1 2 3 4 5 6 7 8
1 RPOZ_THETN: 0.000 0.892 0.605 0.554 0.693 0.648 0.641 0.613
2 RPOZ_STRP1: 0.892 0.000 0.780 0.808 0.896 0.903 0.845 0.865
3 RPOZ_LACDA: 0.605 0.780 0.000 0.473 0.595 0.593 0.650 0.628
4 RPOZ_PEDPA: 0.554 0.808 0.473 0.000 0.651 0.588 0.586 0.564
5 RPOZ_STAA1: 0.693 0.896 0.595 0.651 0.000 0.243 0.612 0.653
6 RPOZ_STAES: 0.648 0.903 0.593 0.588 0.243 0.000 0.584 0.608
7 RPOZ_BACAN: 0.641 0.845 0.650 0.586 0.612 0.584 0.000 0.404
8 RPOZ_BACSU: 0.613 0.865 0.628 0.564 0.653 0.608 0.404 0.000
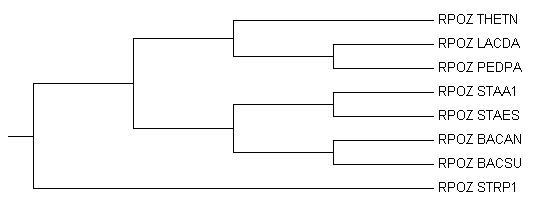
Удалив из скобочных формул расстояния я решил получить наглядное изображение деревьев и получил очень интересный результат.
| CLUSTER ALGORITHM |
TOPOLOGICAL ALGORITHM |
 |
 |
Топологический алгоритм сервера Treetop выдал правильное, без относительно расстояний, филогенетическое дерево.
Выравнивание белков.
©Джумашев