seqret embl:CP000046[529146:530700] -out 16sgene_staes.fasta

| Отсутствует ветвь | Присутствует ветвь |
| (BACSU,BACAN) vs (STAES,STAA1,LACDA,PEDPA,STRP1,THETN) | (STAES,STAA1,BACSU) vs (PEDPA,LACDA,STRP1,THETN,BACAN) |
| (LACDA,PEDPA) vs (STAES,STAA1,BACAN,BACSU,STRP1,THETN) | (STRP1,PEDPA) vs (LACDA,STAA1,BACAN,BACSU,THETN,STAES) |
| Отсутствует ветвь | Присутствует ветвь |
| (BACSU,BACAN) vs (STAES,STAA1,LACDA,PEDPA,STRP1,THETN) | (STAES,STAA1,BACSU) vs (PEDPA,LACDA,STRP1,THETN,BACAN) |
| (STRP1,LACDA) vs (STAES,STAA1,BACAN,BACSU,STRP1,THETN) | (STRP1,PEDPA) vs (LACDA,STAA1,BACAN,BACSU,THETN,STAES) |
| Отсутствует ветвь | Присутствует ветвь |
| (BACSU,BACAN) vs (STAES,STAA1,LACDA,PEDPA,STRP1,THETN) | (STAES,STAA1,BACSU) vs (PEDPA,LACDA,STRP1,THETN,BACAN) |
| (BACAN,BACSU,THETN) vs (STRP1,LACDA,PEDPA,STAA1,STAES) | (STRP1,PEDPA) vs (LACDA,STAA1,BACAN,BACSU,THETN,STAES) |
| (LACDA,PEDPA) vs (STRP1,STAA1,STAES,BACSU,BACAN,THETN) | (STAES,STAA1,BACSU,BACAN) vs (LACDA,STRP1,PEDPA,THETN) |
| Отсутствует ветвь | Присутствует ветвь |
| (BACSU,BACAN) vs (STAES,STAA1,LACDA,PEDPA,STRP1,THETN) | (STAES,STAA1,BACSU) vs (PEDPA,LACDA,STRP1,THETN,BACAN) |
| (LACDA,PEDPA) vs (STAES,STAA1,BACAN,BACSU,STRP1,THETN) | (STRP1,PEDPA) vs (LACDA,STAA1,BACAN,BACSU,THETN,STAES) |
| (STAES,STAA1,PEDPA,LACDA,STRP1) vs (THETN,BACAN,BACSU) | (STAES,STAA1,BACSU,BACAN) vs (LACDA,STRP1,PEDPA,THETN) |
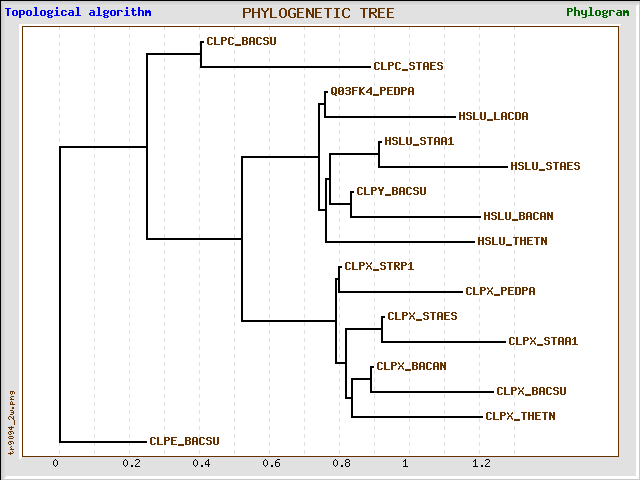
Score E Sequences producing significant alignments: (bits) Value sp|P50866|CLPX_BACSU ATP-dependent Clp protease ATP-binding subu... 770 0.0 sp|Q81LB9|CLPX_BACAN ATP-dependent Clp protease ATP-binding subu... 676 0.0 sp|Q8RC24|CLPX_THETN ATP-dependent Clp protease ATP-binding subu... 572 e-164 sp|A7X396|CLPX_STAA1 ATP-dependent Clp protease ATP-binding subu... 564 e-161 sp|Q8CNY5|CLPX_STAES ATP-dependent Clp protease ATP-binding subu... 562 e-161 sp|Q03F27|CLPX_PEDPA ATP-dependent Clp protease ATP-binding subu... 533 e-152 sp|P63793|CLPX_STRP1 ATP-dependent Clp protease ATP-binding subu... 509 e-145 sp|Q1G9V4|HSLU_LACDA ATP-dependent protease ATPase subunit HslU; 97 6e-21 tr|Q03FK4|Q03FK4_PEDPA ATP-dependent protease HslVU (ClpYQ), ATP... 96 1e-20 sp|Q81WK6|HSLU_BACAN ATP-dependent protease ATPase subunit HslU; 93 1e-19 sp|A7X1N1|HSLU_STAA1 ATP-dependent protease ATPase subunit HslU; 93 1e-19 sp|Q8CPH0|HSLU_STAES ATP-dependent protease ATPase subunit HslU; 91 6e-19 sp|P39778|CLPY_BACSU ATP-dependent protease ATPase subunit ClpY; 90 7e-19 sp|Q8R9Y3|HSLU_THETN ATP-dependent protease ATPase subunit HslU; 90 1e-18 sp|P37571|CLPC_BACSU Negative regulator of genetic competence Cl... 49 3e-06 sp|O31673|CLPE_BACSU ATP-dependent Clp protease ATP-binding subu... 48 4e-06 sp|Q8CQ88|CLPC_STAES ATP-dependent Clp protease ATP-binding subu... 44 9e-05