на главную страницу
Занятие 11
1. Подготовка файлов лиганда
На pdb.org была взята SMILES-нотация для NAG - nag.smi. 3D структура сахара - nag.pdb
2. Докинг
Входные файлы для докинга: nag.pdbqt, seq.B99990002.pdbqt,
vina.cfg
Энергия трех лучших расположений
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -4.8 0.000 0.000
2 -4.6 14.770 16.383
3 -4.4 14.911 16.009
Цифрры довольно высокие.
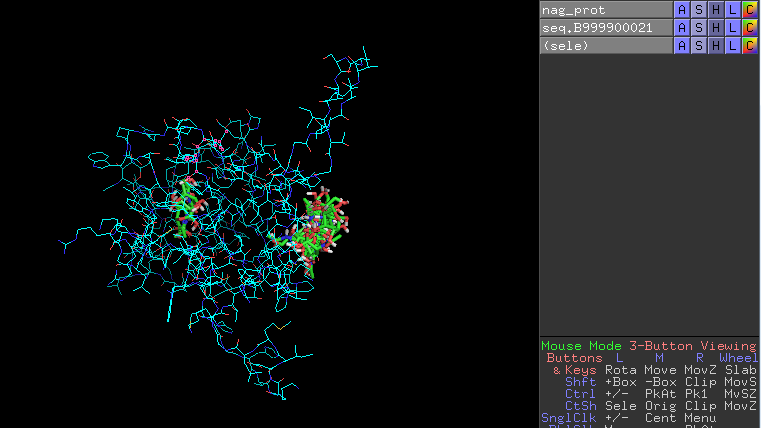
Всего есть 19 вариантов расположений лигандов. Все они представлены на следующей картинке: режим all_states.
3. Докинг с подвижными радикалами
python /usr/share/pyshared/AutoDockTools/Utilities24/prepare_flexreceptor4.py -r seq.B99990002.pdbqt -s LYS144_ASN87_GLY142
С помощью этой команды было проведено деление на подвижную неподвижную составляющие.
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -4.8 0.000 0.000
2 -4.6 1.519 2.426
3 -4.6 12.202 13.534
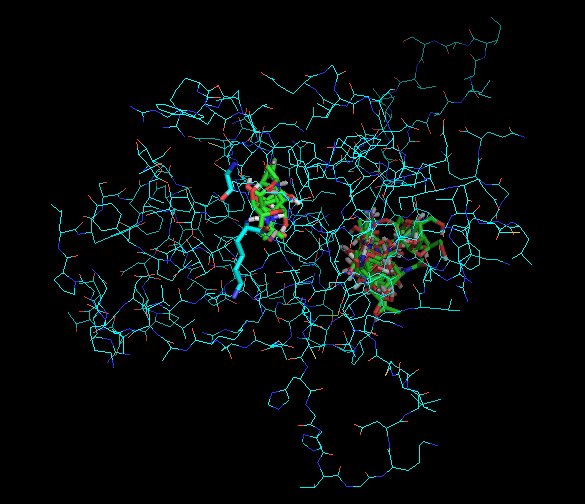
Два остатка Lys144, Gly142 выделены шарнирной формой. Изменение их радикальной части произошло, но собственно мало что изменилось после "гибкого" докинга. Нас допустим не очень интересовало как поведет
себя радикальная боковая часть лизина, если в водородной части участвует атом О пептидной связи. Это очень легко объясняется тем, что
при отборе остатков из-за низкого сходства в выравнивании я был вынужден отобрать водородные связи между атомами остова, а при докинге их положение хоть и поменялось, но незначительно.
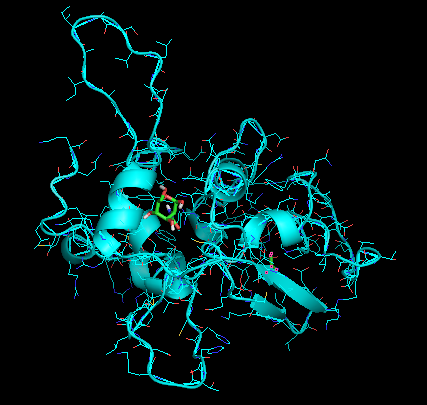
Ниже представлено изображение лучшего докинга, все-таки отобранного из случая с подвижными остатками.
©Джумашев