на главную страницу
Занятие 2
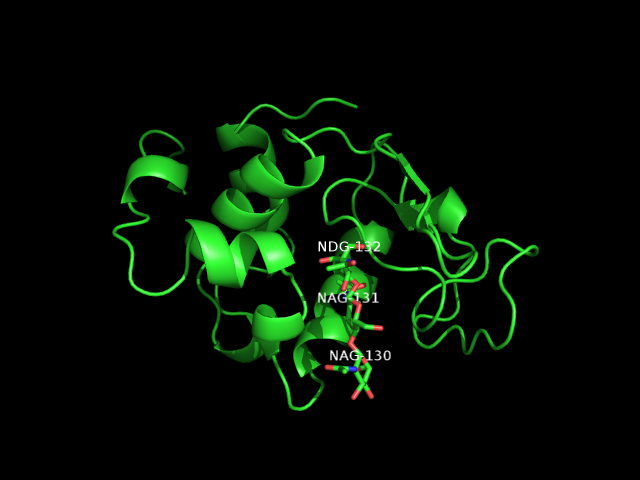
1. Объект 1LMP.pdb
Возможности Sculpting: с помощью Wizard->Demo->Sculpting производится видимый наглядно отбор тех аминокислотных остатков
или других структурных частей расположенных на каком-либо расстоянии от выбранного центра. На изображении отмечены цветом те части белка,
которые предположительно участвуют в стабилизации субстрата в белке.
Кроме того были помечены (кнопка L->Residue) те остатки, которые с высокой долей вероятности образуют водородные связи, и которые можно мутировать для проверки.
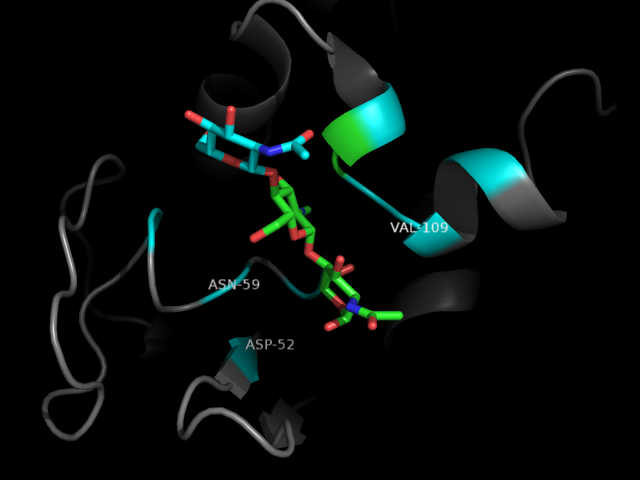
2. Мутагенез
Решено было взять остаток Asp52, по причине того, что карбонильная группа очень близка к атомам N пиррольных колец, в отличие от помеченного (label)
Asn, находится в альфа-спирали белка, к тому же на практике наиболее распространена замена заряженных групп на нейтральные(собственно Asp52Ala мутация была произведена).
Мутация в Pymol осуществляется Wizard->Mutagenesis с последующим выбором нужного остатка, нажатием No Mutation, выбором замены, apply->done.
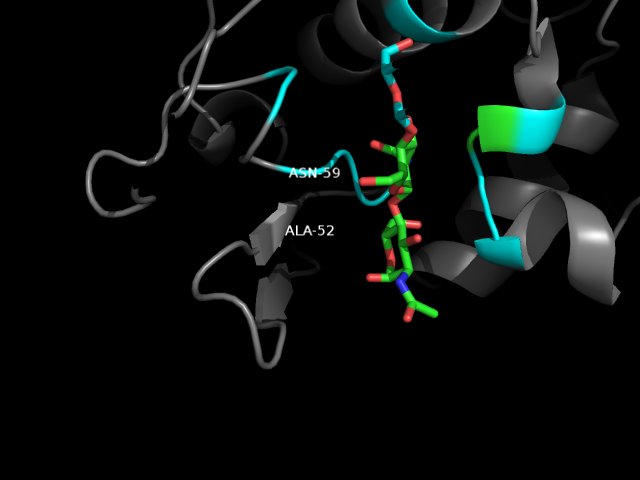
Проверку удобно было сделать Sculpting, видно что после замены Ala уже не входит в радиус возможных остатков, образующих связи с NAG.
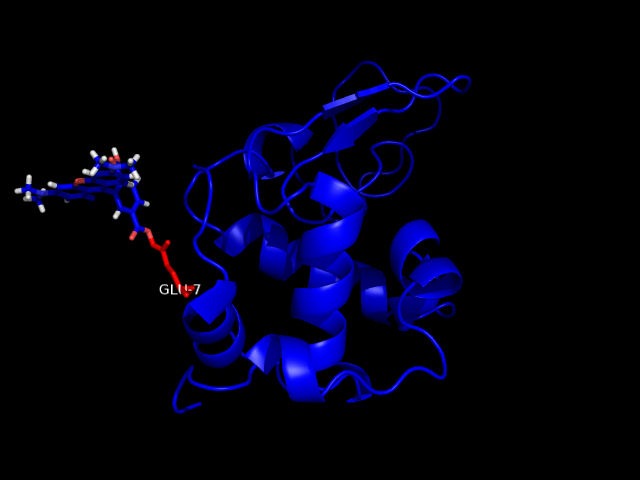
3. Флуоресцентная метка TAMRA
Подготовка файла: открыть sdf файл в pymol, сохранить как pdb. Отбор аминокислотных остатков: для сложноэфирной связи
нужна карбонильная группа(ам.остатка) и он-группа(в молекуле TAMRA). Среди не очень большого количества ASP и GLU был выбран остаток Glu7, который находится на внешней поверхности белковой
молекулы, т.ч. ничего не препятствует объединению с молекулой-флуорофора.
Fuse: открыть обе молекулы в одном окне, выбрать один атом O, затем другой, прописать fuse либо командой fuse (selection1), (selection2).
Последующие команды torsion 15
4. Полиаланиновая спираль
Скрипт для построения полиаланиновой спирали - pala.pml
Все файлы также есть в рабочей директории /term6/Practice1
©Джумашев