Мини-обзор бактерии Chromobacterium phragmitis
Чумакова Ксения Романовна
Московский государственный университет им. Ломоносова
e-mail: ksushka707@mail.ru
Аннотация
В работе представлено изучение бактерии Chromobacterium phragmitis, анализ длины белков, содержания GC% и старт-кодонов. Средняя длина белков составляет 100–300 аминокислот, а GC% варьируется от 60 до 70%. При этом около 90% кодирующих последовательностей (CDS) начинаются со старт-кодона AUG. Большая часть названий псевдогенов соответствует семейству IS5 family transposase и transposase. Полученные данные подтверждают известные закономерности в характеристиках прокариот и способствуют пониманию эволюционной связи данного вида с другими бактериями.
Введение
Chromobacterium phragmitis – вид грамотрицательных, подвижных, фиолетовых бактерий. Они были выделены из болот вдоль приливных участков рек Потомак и Джеймс в штатах Мэриленд и Вирджиния (США) 8 августа 2014 года [1].
Таксономическая принадлежность бактерии:
- Царство: Cellular Organisms
- Домен: Bacteria
- Тип: Pseudomonadota
- Класс: Betaproteobacteria
- Порядок: Neisseriales
- Семейство: Chromobacteriaceae
- Род: Chromobacterium
- Вид: Chromobacterium phragmitis [2]
Цель работы: знакомство с видом Chromobacterium phragmitis, исследование длин белков, GC%, старт-кодонов, псевдогенов данной бактерии, сравнение полученных данных с данными других исследователей.
Материалы и методы
- Для подсчета длин белков C. phragmitis была использована таблица о кодирующих участках ДНК [11], преобразованная в формат Google Table. По данным длины ДНК были найдены длины белков. После этого были созданы карманы по диапазонам значений и количеству длин белков в этих диапазонах, после чего была построена гистограмма.
- Для подсчета содержания GC C. phragmitis также была использована таблица о кодирующих участках ДНК [11], преобразованная в формат Google Table. В третьем столбце таблицы содержатся значения GC%. После этого были созданы карманы по диапазонам значений GC состава, после чего была построена гистограмма.
- Для подсчета метионина в старт-кодонах был использован состав всех белок-кодирующих участков [11] и язык программирования Python.
- Для анализа локальных особенностей генома была использована таблица локальных особенностей генома C. phragmitis [11].
- Для нахождения информации о таксономическом положении бактерии были использованы сайты NCBI, PubMed и другие.
Результаты и обсуждение
1. Длины белков
Ученые выяснили, что размеры белков влияют на их биохимическую структуру и биологические функции. Анализ статистического распределения длины белков в разных таксонах может осветить вопросы, связанные с эволюцией всех клеточных белков, поэтому может быть полезно анализировать длины белков и их функции. Достоверно известно, что средняя длина белков у бактерий составляет около 320 аминокислотных остатков[3]. На гистограмме (рисунок 1) Chromobacterium phragmitis видно, что средняя длина белков данного вида 100-300 аминокислотных остатков, что соответствует известным данным о длине белка у бактерий.
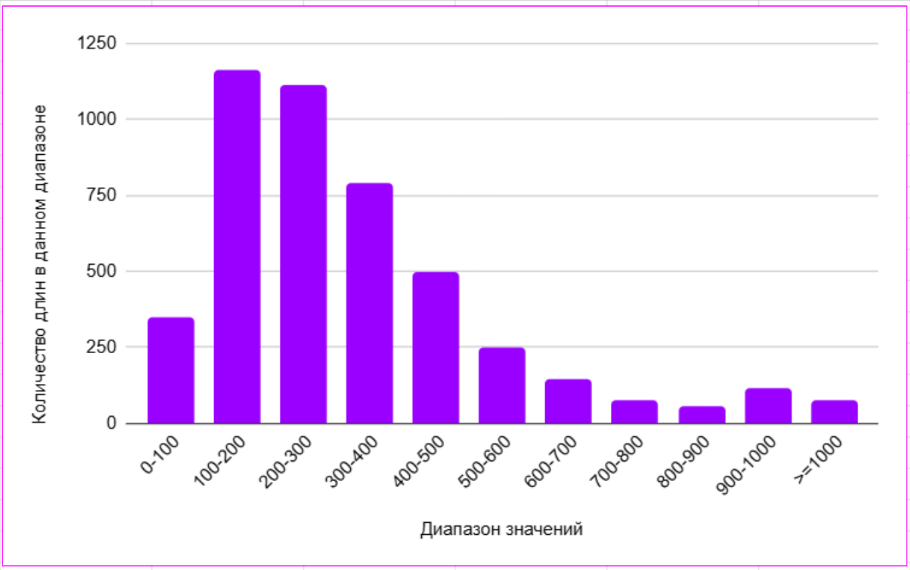
Рисунок 1
2. GC состав Chromobacterium phragmitis
В двойной спирали ДНК гуанин (G) всегда соответствует цитозину (C), а аденин (A) связан с тимином (T). Поэтому в геномах любых клеточных организмов количество G равно количеству C, и количество A соответствует количеству T. Процентное содержание GC, то есть сумма G и C, является важным показателем геномного состава нуклеотидов, пары G и C как правило более стабильны, поэтому используют именно их. Это значение может значительно варьироваться, находясь в диапазоне от 35% до 75%[4-7]. На гистограмме(рисунок 2) видно, что среднее значение процентного содержания GC находится в диапазоне 60-70%, что соответствует норме.
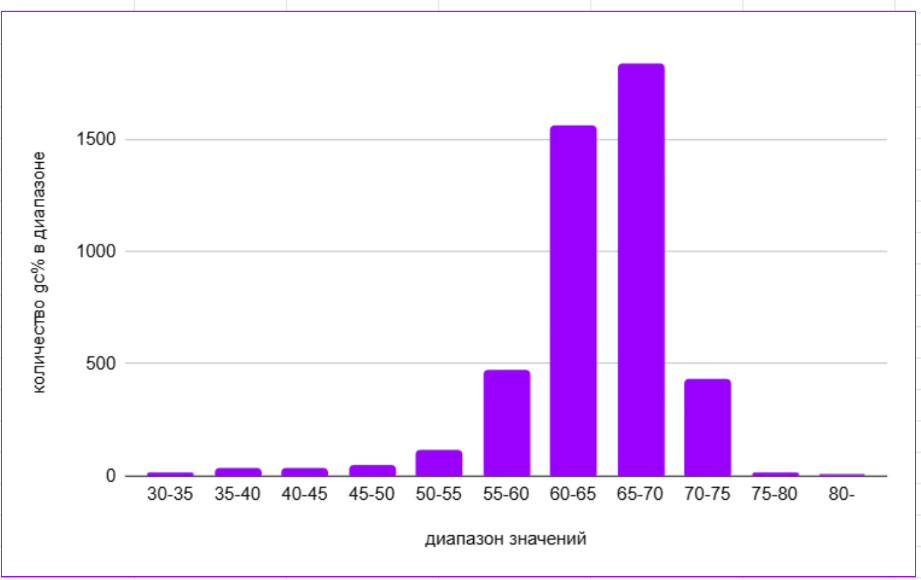
Рисунок 2
3. Старт-кодоны Chromobacterium phragmitis
Известно, что старт-кодон — первый триплет мРНК, с которого начинается трансляция белка в рибосоме. Или начало информационного промежутка соответствующей цепи ДНК. У всех организмов основным старт-кодоном является AUG(ATG для ДНК). Для прокариот характерен AUG в 80-90% случаях[8-10]. Именно поэтому я решила посмотреть характерно ли это для Chromobacterium phragmitis. Воспользовавшись данными о CDS[11] я написала программу, с помощью которой можно узнать интересующую меня информацию. Всего CDS(цепей ДНК) - 4507, из них 4083 начинаются с AUG, что составляет ≈ 90%. Можно также исследовать какие другие триплеты являются старт-кодонами, соответствуют ли они уже известным(GUG и UUG).
4. Названия псевдогенов у Chromobacterium phragmitis
Псевдогены обычно рассматриваются как неактивные копии генов, которые потеряли способность служить матрицами ДНК для производства функциональных продуктов[12]. Изучение псевдогенов, их активности, локализации, сравнение с соответствующими активными генами может быть хорошей почвой для дальнейших исследований. В своей работе я захотела изучить названия псевдогенов C. phragmitis. В таблице локальных особенностей бактерии содержится вся нужная для этого информация о псевдогенах. Для каждого из псевдогенов существует соответствующий ему without_protein, именно по ним и найдены названия семейств псевдогенов. Результаты можно увидеть в таблице 1.Имена которым соответсвуют более одного псевдогена это IS5 family transposase и transposase. Инсерционные последовательности (IS) — это мобильные повторяющиеся фрагменты ДНК, которые способны интегрироваться в различные участки генома хозяина. Это свойство вносит разнообразие в структуру генома и играет значительную роль в регуляции генов у прокариотов. В частности, было установлено, что IS5 может оказывать влияние на экспрессию близко расположенных генов[13]. Транспозаза – это фермент, который катализирует перемещение МЭ или транспозицию. Мобильные элементы (МЭ) – это участки ДНК, способные изменять свое положение в геноме, копироваться и включаться в специфические сайты[14].
Таблица 1
| Название псевдогена | Их количество |
|---|---|
| 23S rRNA pseudouridine(1911/1915/1917) synthase RluD | 1 |
| ATP-binding protein | 1 |
| dephospho-CoA kinase | 1 |
| dihydrolipoyl dehydrogenase | 1 |
| DMT family transporter | 1 |
| DUF3579 domain-containing protein | 1 |
| GNA T family N-acetyltransferase | 1 |
| IS21-like element helper ATPase IstB | 1 |
| IS3 family transposase | 1 |
| IS5 family transposase | 4 |
| transposase | 3 |
| tyrosine-type recombinase/integrase | 1 |
Список литературы
- Blackburn MB, Farrar RR Jr, Sparks ME, Kuhar D, Mowery JD, Mitchell A, Gundersen-Rindal DE. Chromobacterium phragmitis sp. nov., isolated from estuarine marshes. Int J Syst Evol Microbiol. 2019 Sep;69(9):2681-2686. doi: 10.1099/ijsem.0.003508. PMID: 31199223.
- Таксономическую принадлежность бактерии можно найти на сайте NCBI.
- Tissen A, Pérez-Rodríguez P, Delaye-Arredondo LJ. Mathematical modeling and comparison of protein size distribution in different plant, animal, fungal and microbial species reveals a negative correlation between protein size and protein number, thus providing insight into the evolution of proteomes. BMC Res Notes 5, 85 (2012).
- Basak S, Mukhopadhyay P, Gupta SK, Ghosh TC. Genomic adaptation of prokaryotic organisms at high temperature. Bioinformation. 2010;4(8):352–69.
- Nguyen DT, Wu B, Xiao S, Hao W. Evolution of a record-setting AT-rich genome: indel mutation, recombination, and substitution bias. Genome Biol Evol. 2020;12(12):2344–54.
- Mahajan S, Agashe D. Evolutionary jumps in bacterial GC content. bioRxiv. 2021; 2021.02.16.431469.
- Hu EZ, Lan XR, Liu ZL et al. A positive correlation between GC content and growth temperature in prokaryotes. BMC Genomics 23, 110 (2022).
- Peabody DS. Translation initiation at non-AUG triplets in mammalian cells. J Biol Chem. 1989 Mar 25;264(9):5031-5. PMID: 2538469.
- Lobanov AV, Turanov AA, Hatfield DL, Gladyshev VN. Dual functions of codons in the genetic code. Crit Rev Biochem Mol Biol. 2010 Aug;45(4):257-65. doi: 10.3109/10409231003786094. PMID: 20446809; PMCID: PMC3311535.
- Blattner FR, Plunkett G 3rd, Bloch CA, Perna NT, Burland V, Riley M, Collado-Vides J, Glasner JD, Rode CK, Mayhew GF, Gregor J, Davis NW, Kirkpatrick HA, Goeden MA, Rose DJ, Mau B, Shao Y. The complete genome sequence of Escherichia coli K-12. Science. 1997 Sep 5;277(5331):1453-62. doi: 10.1126/science.277.5331.1453. PMID: 9278503.
- Ссылка, по которой можно найти и скачать файлы всех белок-кодирующих участков (CDS from genomic), таблицу локальных особенностей (feature table) Chromobacterium phragmitis.
- Vanin EF. Processed pseudogenes: Characteristics and evolution. Annu Rev Genet. 1985;19:253–272. doi: 10.1146/annurev.ge.19.120185.001345.
- Tempel S, Bedo J, Talla E. From a large-scale genomic analysis of insertion sequences to insights into their regulatory roles in prokaryotes. BMC Genomics. 2022 Jun 20;23(1):451.
- Статья о транспозазах. Декабрь 2006 года, Дэвид Гудселл.