δ-субъединица АТФ-синтазы
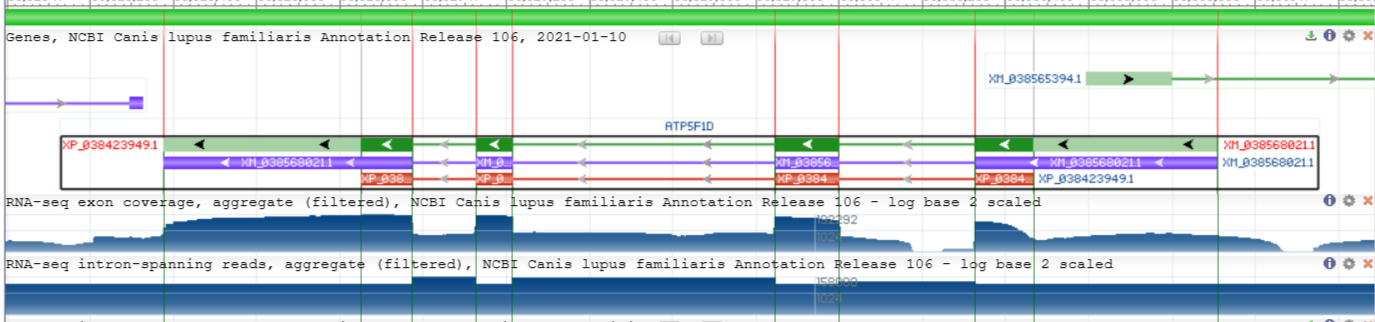
C использованием файла скачанного в прошлом практикуме *protein.faa. Найден ген, кодирующий данный белок его код XP_038423949.1.
В файле формата GBFF по поиску Locus ищем последений локус до нашего протеина - NC_049241.1, находящийся на 20 хромосоме.
Разные варианты BLAST для фрагмента ДНК
Я провела сравнение фрагмента последовательности белок-кодирующей области δ-субъединицы АТФ-синтазы собаки(Canis lupus familiaris) с таксоном пчел(Apoidea). Семейства собачьи и пчелиные достаточно сильно удалены друг от друга, поэтому выдача должна быть интересной. В качестве базы данных в обоих бластах был использован refseq_genomes, по итогу нашлось 38 сборок.
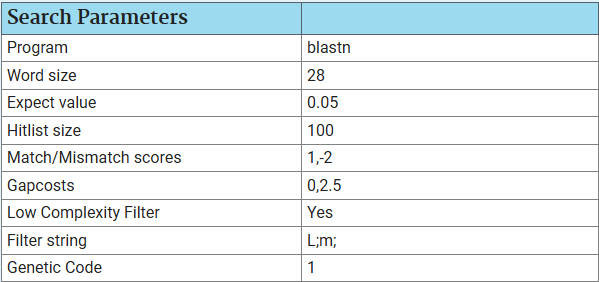
Первый используемый метод - blastn для NC_049241.1 с координатами 58027996-58031287 и Apoidea, на мой взгляд он больше подходит чем megablast для поставленной задаче, потому что рассматриваемые последовательности не являются высокогомологичными.Blastn cравнивает нуклеотидную последовательность с нуклеотидной базой данных.
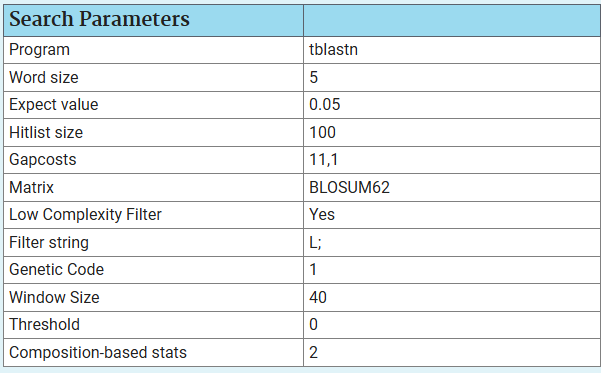
Второй используемый метод - tblastn для белка атф-синтазы и Apoidea, он сравнивает белковую последовательность (запрос) с нуклеотидной базой данных.
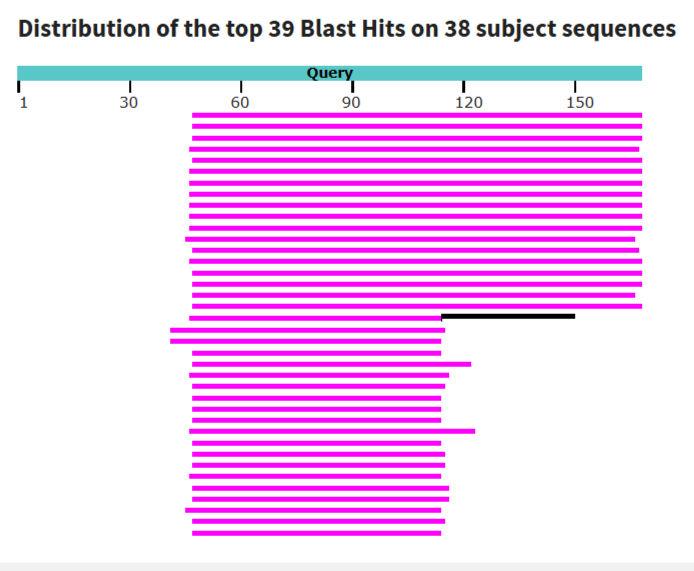
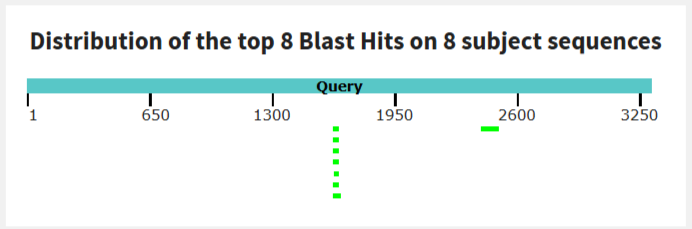
Для tblastn больше находок, чем для blastn, потому что он ищет менее консервативные варианты гомологов.
Разные варианты BLAST для фрагмента ДНК
- Индексация последовательности генома c использованием makeblastdb:
makeblastdb -in "C:\Users\user\Downloads\GCF_011100685.1_UU_Cfam_GSD_1.0_genomic.fna\GCF_011100685.1_UU_Cfam_GSD_1.0_genomic.fna" -dbtype nucl -out meow - Локальный поиск по blast для 1(16s) последовательности:
blastn -task blastn -query 1.txt -db meow -outfmt 7 > res1.txt - Локальный поиск по blast для 2(23S) последовательности:
blastn -task blastn -query 2.txt -db meow -outfmt 7 > res2.txt - Файл с результатом 1, всего 16 находок
- Файл с результатом 2, всего 23 находки
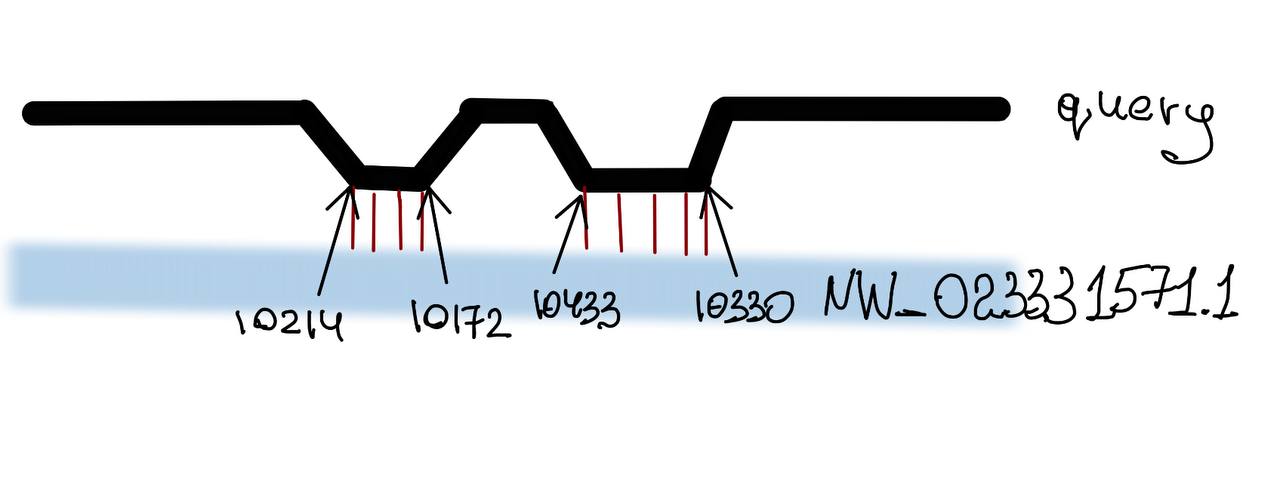
Найдена аннтоация для NW_023331571.1
- Координаты и ориентация:10169-12037
- Идентификатор гена:LOC119879332
Построение карт локального сходства
Для выполнения этого задания мной были выбраны 2 родственных организма Chromobacterium phragmitis и Chromobacterium violaceum. Для построения карт локального сходства были использованы их хромосомные ID GenBank CP029495.1 и соответственно CP069587.1
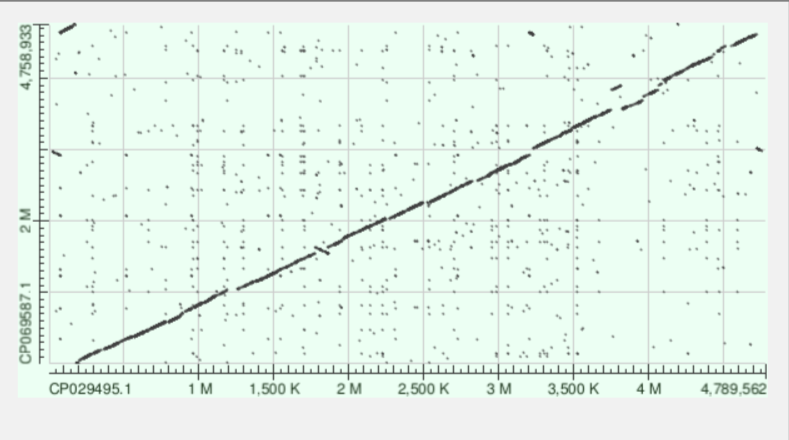
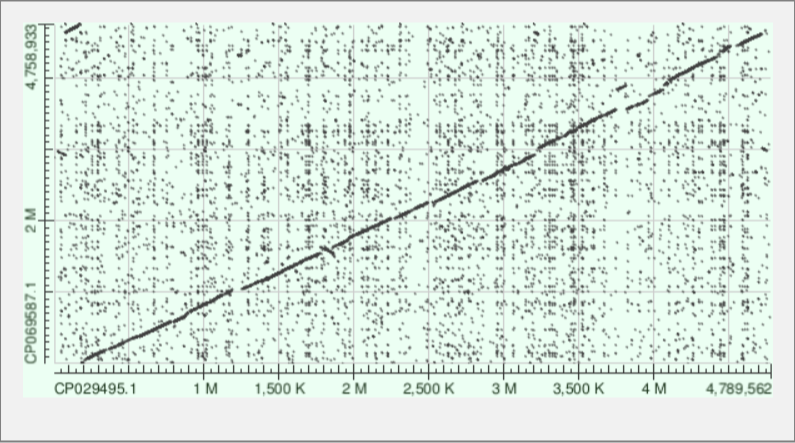
На обеих картах видно транслокацию, делецию и инверсию, хотя основная часть выровнялась. Общий вывод который можно сделать - карты локального сходства отличаются, на второй картинке видно, что между последовательностями есть точечные различия единичные нуклеотидные замены (SNP)(их очень много в отличии от первой) или очень короткие инделы (вставки/делеции), основная часть картинки совпадает, что указывает на близкое родство двух организмов.