Молекулярная динамика плавления ДНК в формамиде в GROMACS
Все рабочие файлы расположены в рабочей директорииАнализ:
-Силовое поле используемое при построении топологии: amber99sb
-Заряд системы: -10. Причины этого значения: отрицательный заряд сахаро-фосфатного остова
-Размер и форму ячейки:параллелепипед: The initial domain decomposition cell size is: X 2.51 nm Y 2.50 nm Z 2.63 nm
-Минимизация энергии:
-Алогритм минимизации энергии: l-bfgs
-Алгоритм расчёта электростатики и Ван-дер-Ваальсовых взаимодействий.
-Модель, которой описывался растворитель:
-Утряска растворителя: -Для биополимеров, укажите параметр который обуславливает неподвижность биополимера
-Число шагов: nsteps=10000
-Длина шага:dt = 0.001 ; ps !
-Алгоритм расчёта электростатики и Ван-дер-Ваальсовых взаимодействий:pme
-Алгоритмы термостата и баростата:Berendsen
-Основной расчёт МД:
-Время моделирования, количество процессоров, эффективность маштабирования
-Длину траектории
-Число шагов:nsteps=1000000
-Длина шага: dt = 0.001 ; ps !
-Алгоритм интегратора: md
-Алгоритм расчёта электростатики и Ван-дер-Ваальсовых взаимодействий: cut-off
-Алгоритмы термостата и баростата:Berendsen
Среднеквадратичное отклонение в ходе моделирования. Так как у нас происходит конформационный переход сначала расчитаем отклонение в ходе все симуляции относительно стартовой структуры:
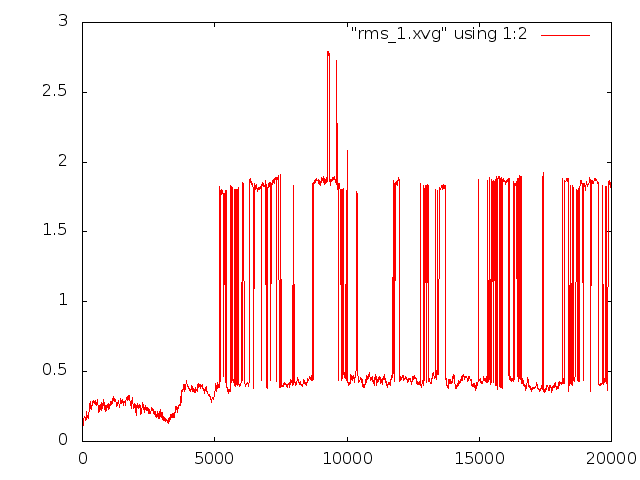
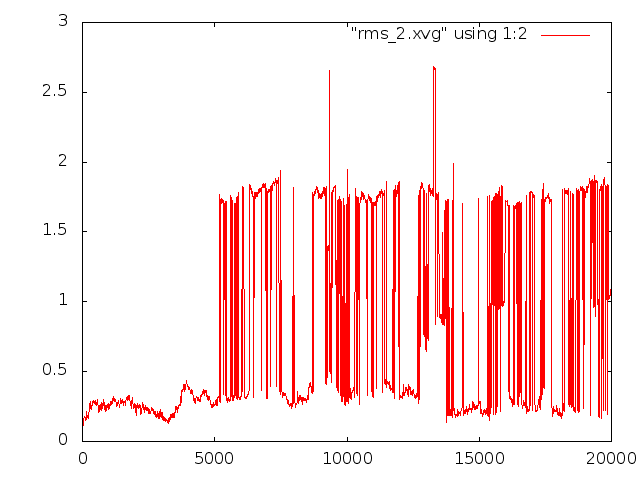
И относительно каждой предидущей структуры на растоянии 400 кадров. Если ближе к концу закончился конформационный переход, то отколнение должно уменьшаться
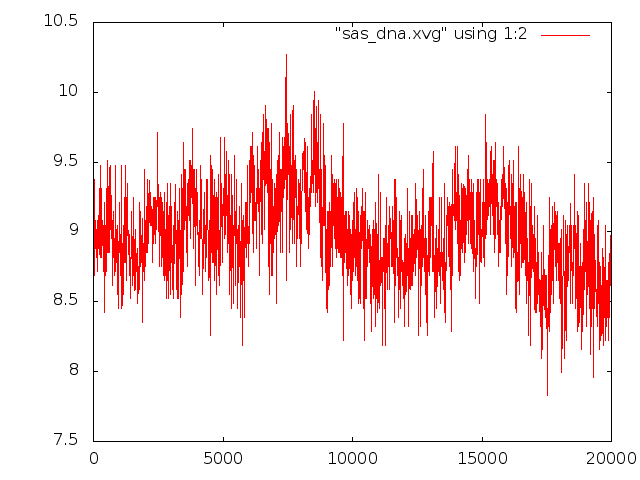
Изменение гидрофобной и гидрофильной поверхности в ходе конформационного перехода:
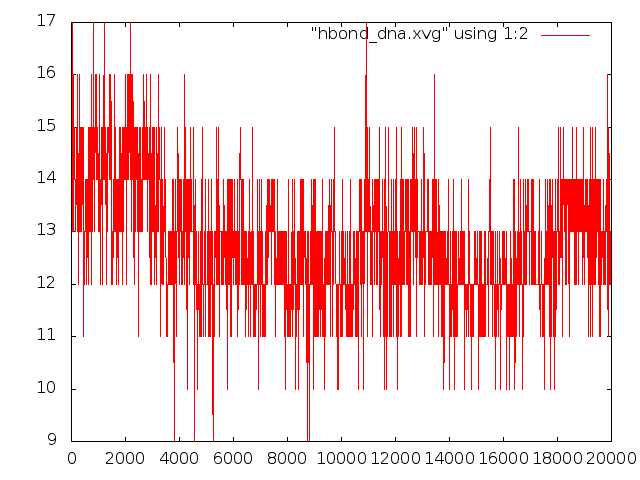
водородные связи между цепями ДНК: