Множественные выравнивания
Выравнивание сделано с помощью программы Jalview, покраска — ClustalX, консервативность ("Above identity threshold") 70%.
Исходная последовательность (в формате fasta)
Проект заданий 1-5 (в формате jar)
Задание 1
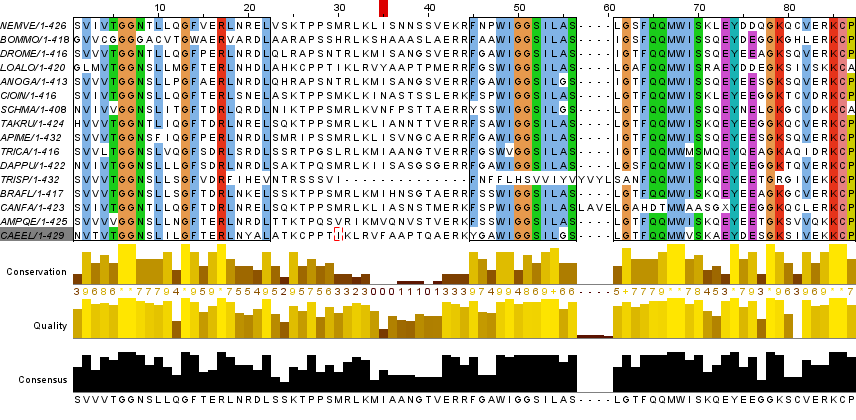
Для начала найдем в выравнивании, построенном программой Jalview, два вертикальных блока аминокислотных остатков, в которых они наиболее сходны во всех белках.
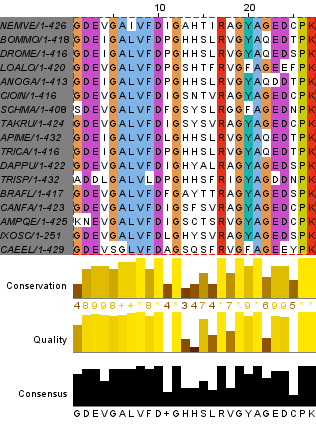
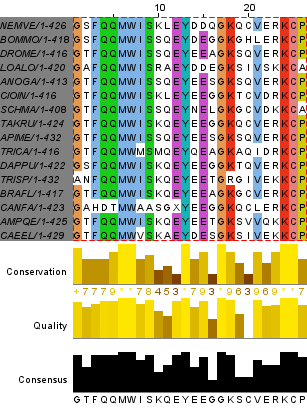
В этих блоках (рис. 1, 2) можно ожидать гомологию остатков разных последовательностей. Код последовательностей белков в базе UniProt указан в колонке слева. В блоке 2 можно заметить, что однобуквенные обозначения аминокислот в столбцах 13-16 не совпадают. Я считаю, что все же можно подозревать здесь консервативность, так как остатки часто сходны по своим свойствам и строению. Например, треонин и серин (есть гидроксильные группы); валин и лейцин (гидрофобные, только углеродный скелет в радикале).
В качестве следующего задания нужно было найти блок, где две последовательностей предположительно гомологичны, а другие сильно отличаются от них. На рисунке 3 можно увидеть, что белки ANOGA и DROME имеют сходства аминокислотных остатков между собой, в то время как остальные -- нет. Сортировка была сделана с помощью опции "Sort by pairwise identity". Можно предположить, что эти белки эволюционно ближе друг другу, чем остальным представленным.
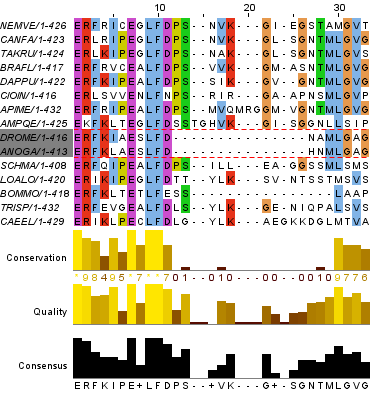
Задание 2
Рассмотрим блок 2 подробно (таблица 1). Можно ли считать его достоверным?

Таблица 1. Консервативность позиций блока 442-467 множественного выравнивания
| Количество | № позиций | Процент позиций | |
| Всего позиций | 26 | 100% | |
| Абсолютно консервативных позиций | 6 | 6, 7, 13, 17, 24, 25 | 23% |
| Абсолютно функционально консервативных позиций | 6 | 1, 8, 15, 18, 21, 23 | 23% |
| Консервативных на 70% позиций | 7 | 3, 4, 5, 9, 11, 12, 26 | 27% |
| Функционально консервативных на 70% позиций | 3 | 2, 14, 22 | 12% |
Таким образом, 85% позиций в этом блоке можно назвать гомологичными.
Задание 3
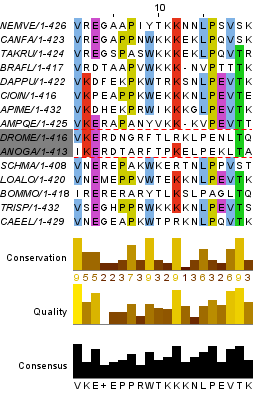
На рисунке 4 изображен участок выравнивания с тремя близко расположенными вертикальными блоками.
Всего позиций: 83
Позиций с гэпами: 4
Процент позиций с гэпами: 5%
Задание 4, 5
К блоку из задания 1 добавлена выданная последовательность и выровнена вручную. Для этого можно использовать перемещение последовательности с помощью стрелок и добавление гэпов с помощью клавишей Shift и Ctrl.
В итоге был выбран вариант, показанный на рисунке 5 (от новой последовательности оставлена только нужная часть).
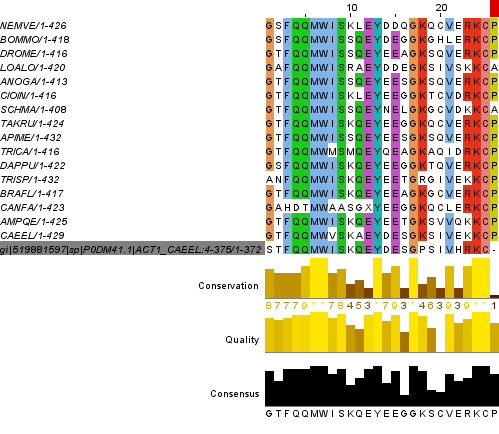
Консенсусная последовательность получившегося выравнивания, то есть последовательность превалирующих аминокислотных остатков в каждой позиции (столбце):
GTFQQMWISKQEYEEGGKSCVERKCP
LOGO — изображение этой последовательности, созданное с помощью сервиса http://weblogo.berkeley.edu/logo.cgi — показано на рисунке 6.

Задание 6
Было построено выравнивание шести заведомо негомологичных белков. Их идентификаторы базы NCBI Protein переведены в Uniprot ID с помощью сервиса ID Mapping на сайте Uniprot. После импортирования последовательности выровнены функцией Web services -> Alignment -> Muscle with default.
Выделены два "блока" в выравнивании (рис. 7, 8). Раскраска ClustalX со значением Above identity threshold >0% показывает много разных цветов, но на самом деле гомологии нет. Для достоверности поднимем процент консервативности, при котором аминокислотные остатки раскрашиваются, до 50%. В итоге цветными остаются несколько разбросанных остатков, что отнюдь не говорит о родстве последовательностей. Кроме того, в таком ложном выравнивании значительно больше гэпов.

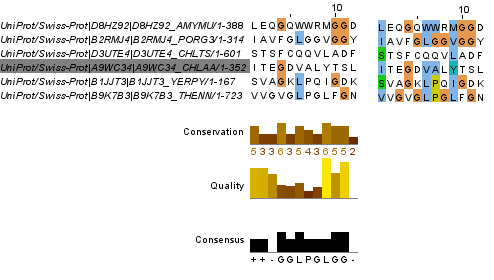
Таким образом, об истинной гомологичности последовательностей выравнивания свидетельствуют крупные блоки с хорошей консервативностью столбцов и небольшое количество гэпов.