Предсказание парных выравниваний
Задания 1, 2, 3
Для начала выберем из выданного множественного выравнивания две последовательности (первая и вторая) для дальнейшего построения парных выравниваний.
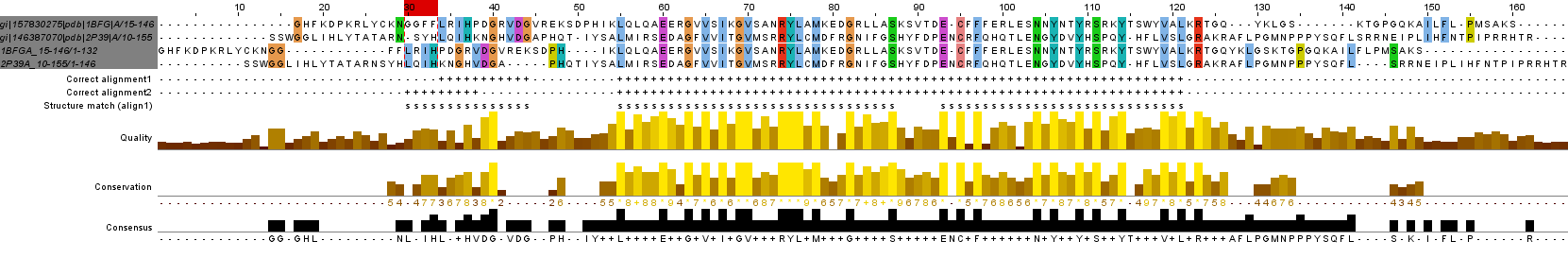
Построим глобальное парное выравнивание, используя программу EMBOSS needle (Needleman-Wunsch global alignment of two sequences). Программа вычисляет и выдает выравнивание целых последовательностей с наибольшим счетом. Необходимо задать программе две входные последовательности белков, штраф за первый гэп, штраф за последующий (в случае, если гэпы идут один за другим). Последний из упомянутых штрафов должен быть меньше, чем первый, так как чаще встречается небольшое количество длинных гэпов, чем много коротких. Для того, чтобы выходной файл был в формате fasta, нужно добавить в строчку запроса опцию -aformat3 fasta. Автоматически выдается файл .needle, где в начале есть аннотация и далее указывается полное и функциональное сходство аминокислотных остатков. На рисунке 1 можно увидеть часть выходного выравнивания этой программы.
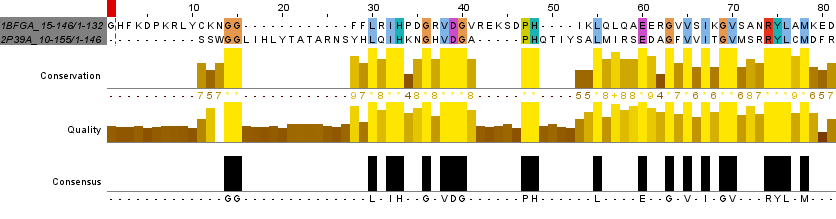
Вторая необходимая программа — EMBOSS water (Smith-Waterman local alignment of sequences). Она строит локальное выравнивание (то есть выр-е фрагмента последовательностей) также с максимальным счетом. Входные данные те же, что и для needle. На рисунке 2 изображено выходное выравнивание.
Задание 4
Возьмем два заведомо негомологичных белка (гипотетический белок A.pernix K1 и белок алкоголь дигидрогеназы P.aerophilum IM2) и применим к ним needle и water.
Как было сказано ранее, в файле автоматического формата .needle/.water показано сходство аминокислот абсолютно и функционально. Две точки означают полное сходство по функциям, а, значит (предположительно) по происхождению. Одна точка говорит о неполном сходстве. Вертикальная палка -- абсолютное совпадение.
выравнивание needle: в форматах .fasta и .needle
выравнивание water: в форматах .fasta и .water
В итоге мы получили глобальное выравнивание с большим количеством гэпов, а локальное -- очень короткое (только из 16 позиций, всего позиций ~300). Это неудивительно, ведь белки разного происхождения.
Задание 5
В таблице 1 сравнены выравнивания из предыдущих заданий.
Таблица 1. Характеристики выравниваний из предыдущих заданий (первая колонка -- последовательности).
| длина выравнивания | число и процент совпадений | число и процент сходных | число и процент гэпов | число открытий гэпов | |
| Две из множественного (глобальное) | 146 | 17 (12%) | 63 (43%) | 27 (19%) | 6 |
| Две из множественного (локальное) | 90 | 14 (16%) | 33 (37%) | 11 (12%) | 4 |
| Две негомологичные (глобальное) | 331 | 26 (8%) | 41 (12%) | 65 (20%) | 11 |
| Две негомологичные (локальное) | 16 | 1 (6%) | 9 (56%) | 4 (25%) | 1 |
Информация получена с помощью программы infoalign, которой необходимо подать на вход fasta-файл с выравниванием. По таблице сразу понятно, что последние два выравнивания намного менее достоверны. В глобальном выравнивании негомологичных белков намного больше открытий гэпов (11 vs 6), что может говорить о "подгонке" последовательностей для хорошего счета выравнивания. Кроме того, значительно меньше процент идентичных аминокислотных остатков (12% vs 43%). Может бросаться в глаза высокий процент сходных остатков в локальном выравнивании негомологичных, но длина выравнивания очень мала, что опять же говорит о том, что эти белки далеки друг от друга по происхождению.
Задание 6
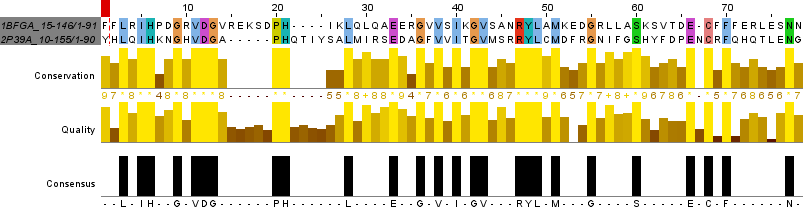
В JalView добавим к парному выравниванию, просто выделенному из множественного, выравнивание программы needle этих же двух последовательностей. Получили "двойное" выравнивание (на рис. 4 эти выравнивания -- два верхних белка и два нижних).
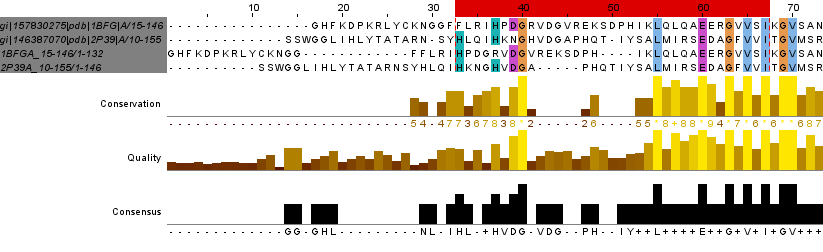
Участок явного различия двух выравниваний показан на рисунке 4. Координаты аминокислотных остатков участка: 41-54. С двух сторон от него есть столбцы с высокой консервативностью, аминокислоты в них выделены цветом.
число различающихся колонок в первом выравнивании: 23
число различающихся колонок в втором выравнивании: 24
Задание 7
Проанализируем правильность двух выравниваний, о которых говорилось в задании 6.
Во-первых, надо проверить на достоверность гомологию совпадающих аминокислотных остатков в столбцах вручную. Для этого в двух строках-аннотациях соответственно для первого и второго выравнивания проставим знак "+" или "-" (см. рисунок 5). Знак "+" проставлен, если в этом же столбце множественно выравнивая аминокислотные остатки совпадают в значительной степени (было взято Quality 4 и более) и/или этот столбец находится в кластере. Таким образом, выделенные покраской ClustalX столбцы парных выравниваний не всегда оказываются достоверными.
Во-вторых, проверим совпадение остатков (только для первого выравнивания), в трехмерной структуре с помощью Rasmol. Одна ошибка 1го рода: C_alpha атомы пары остатков в колонке выравнивания, отмеченной "+", в структуре, очевидно, не совмещаются. Сомнения трактуйте в свою пользу. Одна ошибка 2го рода: C_alpha атомы двух остатков, очевидно, хорошо совмещаются, но соответствующие буквы либо не находятся в одной колонке, либо в одной колонке, но колонка не отмечена "+". Выявлено 6 ошибки первого рода, и 4 -- второго.
На выравнивания с метками можно посмотреть в проекте
.