Мотивы. MEME. Prosite
проект JalView с множественным выравниванием и участком мотива, по которому сроился паттерн
Мотив -- консервативный участок последовательности, который, возможно, говорит о некой сходной функции или происхождении белков. Мотив можно закодировать паттерном, то есть схемой, по которой могут быть найдены идентичный или похожий мотивы в других последовательностях. Сильный паттерн определен довольно точно, задает жесткие рамки для находок. Соответственно, их может быть меньше, но достоверность гомологии в таком случае значительней. Напротив, в слабом паттерне аминокислотные остатки указаны не так определенно, такие паттерны дают больше степеней свободы для остатков. Следовательно, предполагаемых гомологов будет найдено больше, но и достоверность общего происхождения таких находок не так высока.
Для работы с мотивами можно использовать сервис ProSite. Он может находить мотивы в заданных последовательностях белка, а может действовать и наоборот, выдавая последовательности по введенному паттерну.
Для начала подадим на вход fasta-файл с последовательностью гипотетического белка археи A.pernix и ее гомологами из эукариот. Во всех белках найден так называемых PUA домен. Этот мотив учавствует в сшивании РНК, собственно, в ее метаболизме (см. pr13). На рисунке 1 показан мотив, определенный у одной из последовательностей, на рисунке 2 -- часть этого мотива, по которому и будет построен паттерн.
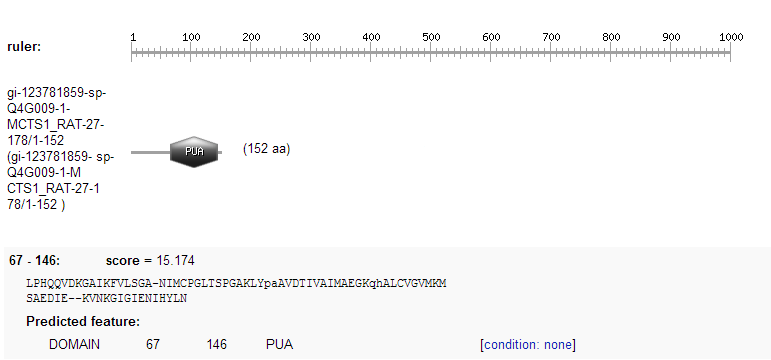
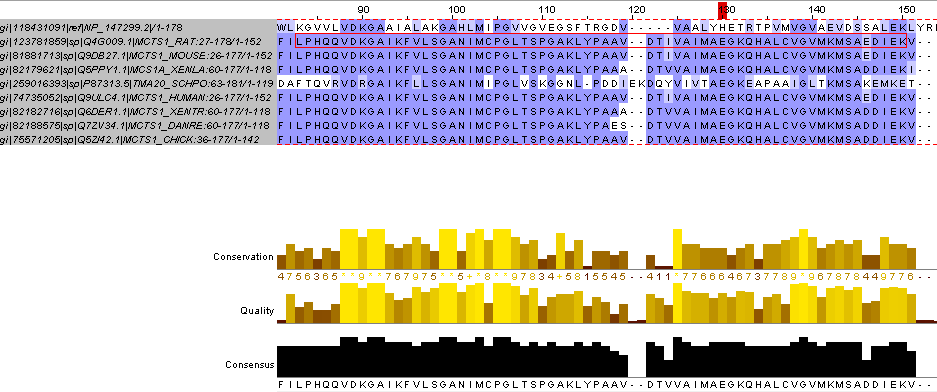
Весь мотив довольно большой, поэтому было решено сделать паттерн по части домена.
Сильный паттерн для поиска выглядел так:
V-D-[KR]-G-A-X(1,3)-[VLI]-[AL]-X(1,3)-G-A-X(1,3)-M-X(1,2)-P-G-[VL]-X(1,20)-V-[AI]-X(1,7)-[RQE]-X(4)-[VLI]-G-[VLI]-X-[KE]-[VM]-X-[AS]-X(2)-[VLI]-[EK]-[EK]
Найдено 74 последовательности (из них 13 -- в SwissProt, 61 -- в TrEMBL). Оказалось, что у первых нескольких находок найденный мотив -- тот самый домен PUA. Значит, паттерн был задан хорошо. Но длина мотива в последовательностях не выходила за рамки 178-182 аминокислот (тогда как в введенной ранее совокупности белков длина домена варьировалась сильно). Скорее всего, это и есть "цена" четких рамок сильного паттерна.
Слабый паттерн:
V-D-[KR]-G-A-X(1,5)-G-A-X(1,5)-M-X(1,5)-P-G-X(1,20)-V-X(1,7)-[RQE]-X(4)-[VLI]-G-X(1,3)-[KE]-X(6)-[EK](2)
Найдено 365 последовательностей (из них 21 -- в SwissProt, 344 -- в TrEMBL). В этом паттерне я исключила (почти все) не полностью консервативные колонки. Поставила более широкие значение для количества x-аминокислот (x -- любые). Длина мотивов в найденных последовательностях варьировалась больше.