Протокол по Uniprot
Uniprot — база данных последовательностей белков. Она делится на два сегмента. Первый - это SwissProt, куда включены последовательности белков, для которых аннотация была сделана экспертом. Второй - TrEMBL - содержит последовательности, полученные с помощью компьютерной трансляции. Аннотация TrEMBL создаётся автоматически, и последовательностей в TrEMBL во много раз больше, чем в SwissProt.
С помощью ID Mapping идентификатор Refseq заданного белка был переведен в код доступа в Uniprot. Идентификатор в банке Uniprot — Q9YEQ6_AERPE.
Ортологи — белки общего происхождения из геномов представителей, например, одного рода, которые появляются в процессе видообразования. Необходимо найти два таких организмов рода Aeropyrum. Это гипертермофильные археи.
Поисковой запрос был таким:
go:"RNA binding" AND taxonomy:Aeropyrum NOT taxonomy:pernix
Поиск происходит по одному имеющему белку A.pernix — hypothetical protein (Q9YEQ6_AERPE). Поскольку название белка не связано с функцией (оно говорит лишь о том, что существование белка теоретическое, то есть предсказано по гену), поиск идет по полю Gene Ontology (GO): RNA binding [связывание РНК].
Uniprot выдал записи белков с такой функцией только еще одного вида — A.camini (отсюда берем белок с ID U3T8L7_9CREN), и они все из TrEMBL. Чтобы найти еще один похожий белок в другом (но близком) таксоне, я воспользовалась сервисом Blastp, который нашел гомологов первой последовательности. Из них мы возьмем белок Pyrolobus fumarii c ID G0EEX4_PYRF1. Это архея другого рода, но того же порядка Desulfurococcaceales.
Для каждого из трех белков был скачан файл со всей информацией о них:
Эти данные структурированы в таблице 1.
Таблица 1. Сравнение белков трех ортологов.
| Метка поля | Содержание | |||
| Гипотетический белок Aeropyrum pernix | Гипотетический белок Aeropyrum camini | Содержащий домен PUA белок Pyrolobus fumarii | ||
| Идентификатор записи | ID | Q9YEQ6_AERPE | U3T8L7_9CREN | G0EEX4_PYRF1 |
| Код доступа первый ("Accession number") | AC | Q9YEQ6 | U3T8L7 | G0EEX4 |
| Код(ы) доступа остальные | AC | нет | Нет | нет |
| Дата создания документа | DT | 01-NOV-1999 | 11-DEC-2013 | 19-OCT-2011 |
| Дата последнего исправления аннотации | DT | 16-OCT-2013 | 16-APR-2014 | 16-OCT-2013 |
| Название (краткое описание) белка | DE | Unchacterized protein | Predicted RNA-binding protein | PUA domain containing protein |
| Название организма | OS | Aeropyrum pernix (stin ATCC 700893 / DSM 11879 / JCM 9820 / NBRC 100138 / K1). | Aeropyrum camini SY1 = JCM 12091 | Pyrolobus fumarii (strain DSM 11204 / 1A) |
| Таксономия | OC | Archaea; Crenarchaeota; Thermoprotei; Desulfurococcales; Desulfurococcacea; Aeropyrum. | Archaea; Crenarchaeota; Thermoprotei; Desulfurococcales; Desulfurococcaceae; Aeropyrum. | Archaea; Crenarchaeota; Thermoprotei; Desulfurococcales; Pyrodictiaceae; Pyrolobus. |
| Название локуса гена | GN | APE_0525.1 | ACAM_0403 | Pyrfu_0216 |
| Номер публикции | RX | PubMed=10382966; DOI=10.1093/dnares/6.2.83 | PubMed=23872576; DOI=10.1128/AEM.01089-13 | PubMed=21886865 |
| Автор(-ы) публикации | RA | Kawabayasi Y., Hino Y., Horikawa H., Yamazaki S., Haikawa Y., Jin-no K., Takahashi M., Sekine M., Baba S., Ankai A., Kosugi H., Hosoyama A., Fukui S., Nagai Y., Nishijima K., Nakazawa H., Takamiya M., Masuda S., Funahashi T., Tanaka T., Kudoh Y.,Yamazaki J., Kushida N., Oguchi A., Aoki K., Kubota K., Nakamu Y., Nomu N., Sako Y., Kikuchi H.; | Daifuku T., Yoshida T., Kitamura T., Kawaichi S., Inoue T., Nomura K., Yoshida Y., Kuno S., Sako Y.; | Anderson I., Goker M., Nolan M., Lucas S., Hammon N., Deshpande S., Cheng J.F., Tapia R., Han C., Goodwin L., Pitluck S., Huntemann M., Liolios K., Ivanova N., Pagani I., Mavromatis K., Ovchinikova G., Pati A., Chen A., Palaniappan K., Land M., Hauser L., Brambilla E.M., Huber H., Yasawong M., Rohde M., Spring S., Abt B., Sikorski J., Wirth R., Detter J.C., Woyke T., Bristow J., Eisen J.A., Markowitz V., Hugenholtz P., Kyrpides N.C., Klenk H.P., Lapidus A.; |
| Название публикации | RT | 1) "Complete genome sequence of an aerobic hyper-thermophilic crenarchaeon, Aeropyrum pernix K1."; 2) "The structure of gene product APE0525 from Aeropyrum pernix."; 3) "Crystal structure of a PUA domain (APE0525) from the Aeropyrum pernix K1 (sulfate complex)."; 4) "Crystal structure of a PUA domain (APE0525) from the Aeropyrum pernix K1 (tartrate complex)."; | "Variation of the Virus-Related Elements within Syntenic Genomes of the Hyperthermophilic Archaeon Aeropyrum." | "Complete genome sequence of the hyperthermophilic chemolithoautotroph Pyrolobus fumarii type strain (1A). |
| Журнал | RL | DNA Res. 6:83-101(1999). | Appl. Environ. Microbiol. 79:5891-5898(2013) | Stand. Genomic Sci. 4:381-392(2011) |
| Чем обосновано существование белка | PE | Evidence at protein level | Predicted | Predicted |
| Ссылка (-и) на базу 3D структур PDB | DR | 2ZX7, 2CX0, 2CX1 | нет | нет |
| Реакция, катализируемая ферментом | CC: CATALYTIC ACTIVITY | нет | нет | нет |
| Расположение фермента внутри клетки | CC: SUBCELLULAR LOCATION | нет | нет | нет |
Как видно по таблице, о белках известно нет так уж и много. Нет трехмерных PDB-структур у двух ортологов. Судя по всему, названия (Description) белков практически синонимичны: функция и домен у них одинаковые.
Ответы на вопросы из списка.
Предложите мутацию, которая, на Ваш взгляд, сильно повлияет на активность белка.
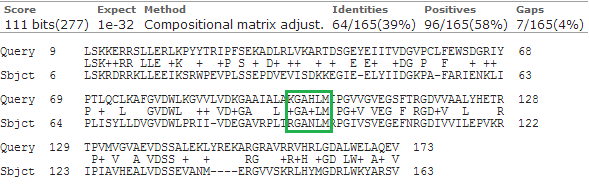
Активность белка в лице способности связывать РНК обеспечивает домен PUA. Он есть во всех трех белках. Можно поменять несколько аминокислот в нем (см. рисунок 1)
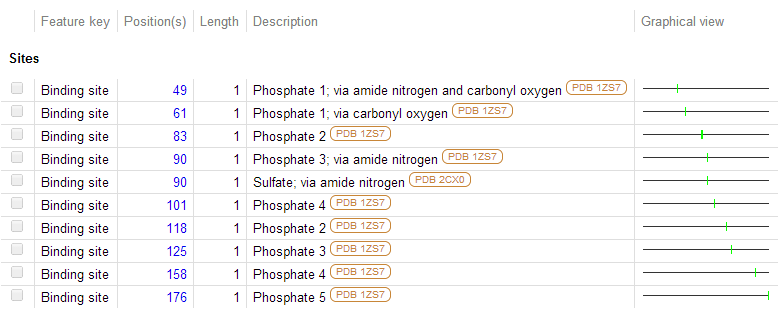
Какие ионы связываются с белком?
Для последовательности есть несколько структур в PDB. Самая богатая ионами — 2SZ7. Там есть 5 фосфат-ионов и один ион калия. Сайты связывания — это, в основном, кислород в карбониле и амидная группа (см. рисунок 2).
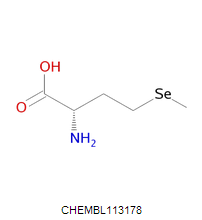
Укажите аминокислотный остаток (русское название, трехбуквенный код, номер в цепи), который модифицируется в белке после его трансляции, и охарактеризуйте эту модификацию.
В структуре 2SZ7 есть три модифицированных остатка: это селенометионин (MSE) в позициях 1, 111, 140. Это модификация метионина, в котором сера заменяется на селен (см. рисунок 3). Модификация делается намеренно для получения кристаллической структуры белка методом рентгеноструктурного анализа.