Компоненты АТФ-синтазы Aeropyrum pernix K1
Таблица 1. Таблица запросов для поиска белков-компонент АТФ-синтазы в Uniprot
| Геном или таксон | Строка запроса | Число записей | Комментарии |
| Aeropyrum pernix K1 | organism:"Aeropyrum pernix" | 1864 | Много лишних белков, полное название огранизма "Aeropyrum pernix (strain ATCC 700893 / DSM 11879 / JCM 9820 / NBRC 100138 / K1) |
| Aeropyrum pernix K1 | organism:"Aeropyrum pernix" AND organism:*K1* | 0 | |
| Aeropyrum pernix K1 | organism:"Aeropyrum pernix (strain ATCC 700893 / DSM 11879 / JCM 9820 / NBRC 100138 / K1)" | 1701 | |
| Aeropyrum pernix K1 | organism:"Aeropyrum pernix (strain ATCC 700893 / DSM 11879 / JCM 9820 / NBRC 100138 / K1)" AND name:"atp" | 54 | не все являются АТФ-синтазами |
| Aeropyrum pernix K1 | organism:"Aeropyrum pernix (strain ATCC 700893 / DSM 11879 / JCM 9820 / NBRC 100138 / K1)" AND gene:atp* | 7 | Итоговый результат |
| Aeropyrum camini | organism:"Aeropyrum camini" AND gene:atp* | 7 | Итоговый результат |
Разные попытки поиска необходимых белков отражены в таблице 1. В итоге найдены семь компонент АТФ-синтазы (рисунок 1). Из них пять более достоверных: они из базы Swissprot.
Структура шести белков выведена из гомологичных (столбец Protein existence). Структура F-субъединицы пред сказана, то есть теоретически выведена из последовательности ДНК и не найдена фактически.
Компоненты синтазы A-F ко-локализованы, то есть кодирующие гены находятся близко в геноме (столбец Gene names, APE_... - локус гена). Гены субъединицы I и P локализованы на расстоянии от них.
.png)
Ссылка на fasta-файл с последовательностями белков субъединиц.
Компоненты АТФ-синтазы Aeropyrum camini K1
Окончательный запрос показан в таблице 1. Найдены семь белков (рисунок 2).
Структура пяти субъединиц из семи заключена из гомологии, остальные два белка -- гипотетические (столбец Protein existence). Все гены субъединиц кроме I и P ко-локализованы (столбец Gene names, ACAM_... - локус гена).
.png)
Ссылка на fasta-файл с последовательностями белков субъединиц.
Гомологи белка Q9YF38 (VATD_AERPE) V-type ATP synthase subunit D
Для поиска гомологичных последовательностей в других геномах поищем белки по сходной семье (Protein family этого белка -- "V-ATPase D subunit family" [1]). Соответственно, поисковой запрос в Uniprot: family:"V-ATPase D subunit family". По нему найдено 2122 последовательности.
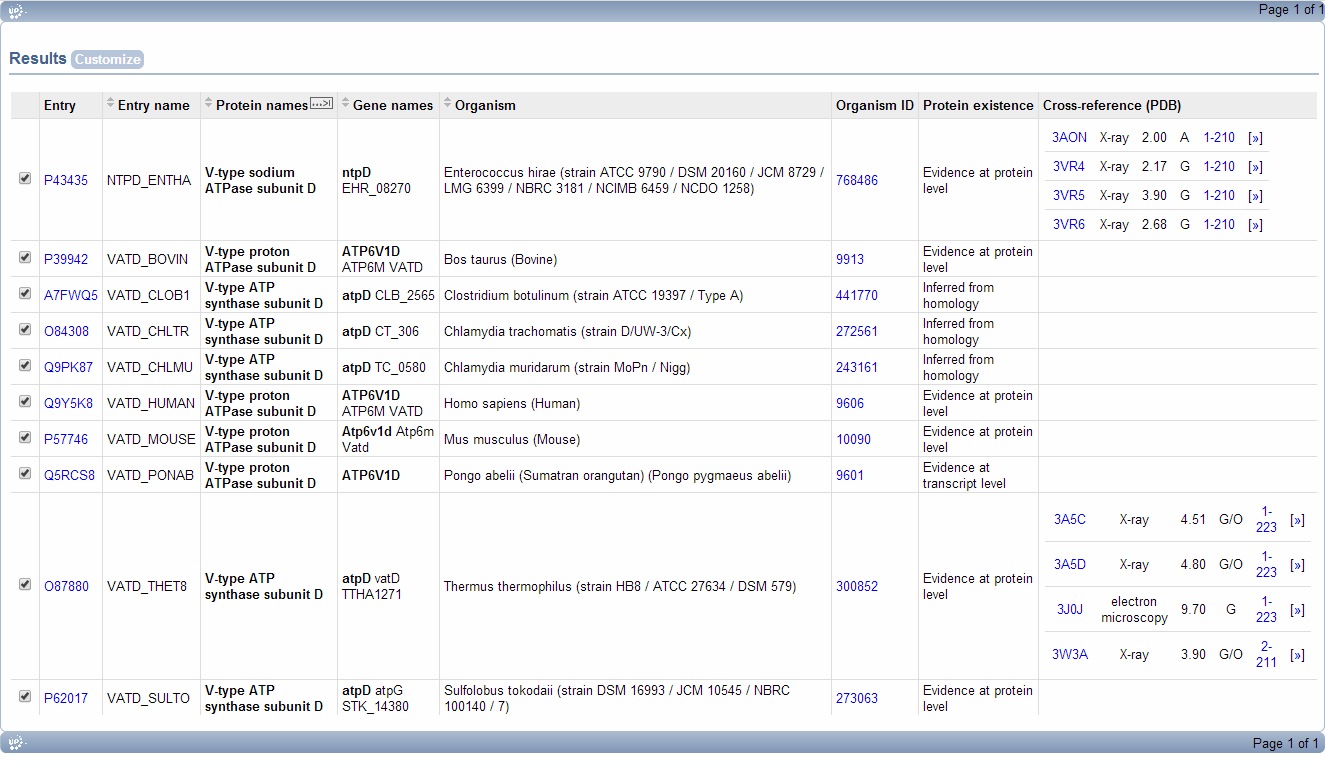
Для поиска белков с известной структурой в PDB был задан такой запрос: family:"V-ATPase D subunit family" AND database:(type:pdb *). Есть только два таких белка, они занесены в итоговый fasta-файл вместе с некоторыми другими гомологами структуры Q9YF38 (VATD_AERPE). Данные об этих белках показаны на рисунке 3.
Изученность протеома археи Aeropyrum pernix K1
Таблица 2. . Сводная таблица протеома Aeropyrum pernix K1. Пояснения в тексте.
| база UniProtKB/SwissProt | база UniProtKB/TrEMBL | |
| всего | 367 | 1334 |
| Evidence at protein level | 32 | 23 |
| Evidence at transcript level | 0 | 1 |
| Inferred from homology | 329 | 128 |
| Predicted | 6 | 1182 |
В таблице 1 приведены данные по белкам протеома. В базе UniProtKB содержатся структуры, рецензированные экспертом, в TrEMBL же они не рецензированы, то есть менее достоверны.
Некоторые пояснения к таблице:
- Evidence at protein level: есть четкие экспериментальные данные, доказывающие существование и структуру белка (секвенирование по Эдману, четкая идентификация масс-спектрометрией и др.)
- Evidence at transcript level: нет строгих доказательств существования, есть данные по экспрессии
- Inferred by homology: 'выведено из гомологии', то есть существование белка следует из похожих структур у близких видов
- Predicted: 'предсказано', то есть нет вышеперечисленных доказательств
Белков в совершенно достоверной структурой только 3% от общего числа, выведенных из гомологии -- 27%. Много структур с неизвестными доказательствами, но они из нерецензируемой базы данных TrEMBL. Можно сделать вывод, что протеом археи изучен слабо, в основном собран по белкам близких огранизмов.
Источники:
[1]http://www.uniprot.org/uniprot/Q9YF38 "Sequence similarities: Belongs to the V-ATPase D subunit family."