Хемоинформатика
Необходимо, используя пакет модулей RDkit, предложить аналог ибупрофена. Для этого нужно:
- найти формулу ибупрофена и предложить способ изменения его SMILES для эмуляции продукта Click Chemistry;
- на сайте PubChem найти все радикалы c азидом для Click Chemistry и скачать их SMILES нотации;
- заменить в найденых радикалах азидную группу на модифицированный ибупрофен;
- изобразить структуры модифицированных азидов;
- отобрать те молекулы, которые удовлетворяют правилу пяти Липински.
Загрузим RDkit — пакет для работы с хемоинформатикой.
from rdkit import Chem
from rdkit.Chem import AllChem
from rdkit import RDConfig
from rdkit.Chem.Draw import IPythonConsole
from rdkit.Chem import Draw
import numpy as np
from IPython.display import display,Image
Нарисуем ибупрофен:
ibu=Chem.MolFromSmiles('CC(C)CC1=CC=C(C=C1)C(C)C(=O)O')
AllChem.Compute2DCoords(ibu)
display(ibu)
Правило пяти Липински говорит о том, какими свойствами должна обладать молекула, чтобы быть (скорее всего) активной как лекарство при оральном приеме:
- иметь не более 5 доноров водородных связей;
- иметь не более 10 акцепторов водородных связей;
- молекулярная масса не более 500 Да;
- log P (мера липофильности, коэффициент распределения в системе октанол-вода) меньше 5.
Посчитаем параметры правила пяти Липински для молекулы ибупрофена.
import rdkit.Chem.Lipinski as Lipinksy
print Lipinksy.NumHDonors(ibu)
print Lipinksy.NumHAcceptors(ibu)
print Lipinksy.rdMolDescriptors.CalcExactMolWt(ibu)
print Lipinksy.rdMolDescriptors.CalcCrippenDescriptors(ibu)[0]
Сделаем модифицированный ибупрофен, добавив азидную группу.
ibu_mod=Chem.MolFromSmiles('N1C=C(N=N1)C1=CC=C(C=C1)C(C)C(=O)O')
AllChem.Compute2DCoords(ibu_mod)
display(ibu_mod)
Скачали структуры всех радикалов с азидом в формате SMILES из базы PubChem ('azides_smiles.txt'). Извлечем все SMILES азидов:
strings=np.genfromtxt('azides_smiles.txt',dtype=np.str)
smiles = []
for line in strings:
#formula length <30
if len(line[1]) < 30 and not '.' in line[1]:
smiles.append(line[1])
print smiles[0:100]
Теперь в скачанных SMILES азидов заменяем азидную группу на модифицированный ибупрофен (будем работать только с 1500 из азидов). Полученные структуры фильтруем по правилу Липински. А также отбираем молекулы более растворимые в воде, чем ибупрофен. Запоминаем самую водорастворимую и показываем ее в конце.
good_new_smiles=[]
n=0
min = Lipinksy.rdMolDescriptors.CalcCrippenDescriptors(ibu)[0] #logP
min_index = 0
#Новую молекулу лучше создавать в try из-за возможных битых Smiles
for smi in smiles[:1500]:
if 'N=[N+]=[N-]' in smi:
newsmi=smi.replace('N=[N+]=[N-]','N1C=C(N=N1)C1=CC=C(C=C1)C(C)C(=O)O') #replace with modified ibuprofen
else:
continue
try:
newmol=Chem.MolFromSmiles(newsmi)
#see if Lipinski rule is followed AND molecule is more water-soluble than ibuprofen
if Lipinksy.NumHDonors(newmol) <= 5 and Lipinksy.NumHAcceptors(newmol) <= 10 and Lipinksy.rdMolDescriptors.CalcExactMolWt(newmol) <=500 and Lipinksy.rdMolDescriptors.CalcCrippenDescriptors(newmol)[0] <= Lipinksy.rdMolDescriptors.CalcCrippenDescriptors(ibu)[0]:
good_new_smiles.append(newmol)
AllChem.Compute2DCoords(newmol)
display(newmol)
print n
n=n+1
if Lipinksy.rdMolDescriptors.CalcCrippenDescriptors(newmol)[0] < min:
min = Lipinksy.rdMolDescriptors.CalcCrippenDescriptors(newmol)[0]
min_index = n-1
except:
pass
print 'good molecules: ', float(n)/float(1500)*100,'%'
print 'molecule with min log P: molecule #%i with log P=%i' %(min_index,min)
display(good_new_smiles[min_index])
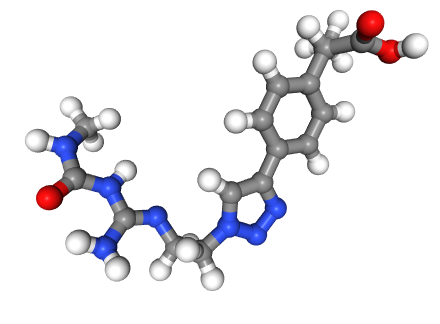
Лучшая молекула - под номером 21. Так она выглядит в 3D:
m3d=Chem.AddHs(good_new_smiles[21])
Chem.AllChem.EmbedMolecule(m3d)
AllChem.MMFFOptimizeMolecule(m3d,maxIters=500,nonBondedThresh=200 )
import nglview as nv
nv.show_rdkit(m3d)