| Название | Мнемоника |
| Agrobacterium tumefaciens | AGRRK |
| Rhodobacter sphaeroides | RHOS4 |
| Ralstonia pickettii | RALPJ |
| Pseudomonas aeruginosa | PSEAE |
| Erwinia carotovora | ERWCT |
| Escherichia coli | ECOLI |
| Yersinia pestis | YERPE |
((RHOS4,AGRRK),(RALPJ,(PSEAE,(YERPE,(ERWCT,ECOLI)))));
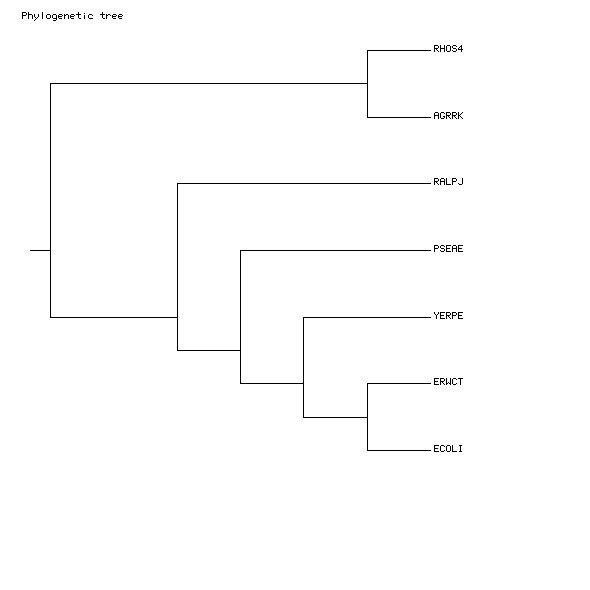
С помощью сервиса NCBI (http://www.ncbi.nlm.nih.gov/taxonomy/) определим, к каким таксонам принадлежат выбранные мной бактерии.
Agrobacterium tumefaciens (AGRRK): Bacteria; Proteobacteria; Alphaproteobacteria; Rhizobiales; Rhizobiaceae; Rhizobium/Agrobacterium group; Agrobacterium Rhodobacter sphaeroides (RHOS4): Bacteria; Proteobacteria; Alphaproteobacteria; Rhodobacterales; Rhodobacteraceae; Rhodobacter Ralstonia pickettii (RALPJ): Bacteria; Proteobacteria; Betaproteobacteria; Burkholderiales; Burkholderiaceae; Ralstonia Pseudomonas aeruginosa (PSEAE): Bacteria; Proteobacteria; Gammaproteobacteria; Pseudomonadales; Pseudomonadaceae; Pseudomonas; Pseudomonas aeruginosa group Erwinia carotovora (ERWCT): Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Pectobacterium Escherichia coli (ECOLI): Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Escherichia Yersinia pestis (YERPE): Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Yersinia
Можно заметить, что на дереве отобранных мной бактерий есть ветви, выделяющие некоторые таксоны.
Например, самая верхняя ветвь построенного дерева выделяет таксон Alphaproteobacteria, к которому относятся Agrobacterium tumefaciens и Rhodobacter sphaeroides.
Также есть нетривиальная ветвь, выделяющая таксон Gammaproteobacteria, к которому относятся
Pseudomonas aeruginosa, Erwinia carotovora, Escherichia coli и Yersinia pestis.
Еще есть нетривиальная ветвь, выделяющая таксон Enterobacteriaceae, к которому относятся
Erwinia carotovora, Escherichia coli и Yersinia pestis.
Далее из списка функций белков я выбрала одну: по белкам соответствующего семейства в дальнейшем я буду реконструировать филогенетическое дерево.
Выбор мой пал на Фактор элонгации трансляции Ts.
Получим из Swiss-Prot последовательности белков с данной функцией из отобранных бактерий
(для этого надо было вспомнить, как получить ID банка Swiss-Prot из мнемоники функции и мнемоники организма).
Я использовала следующую команду:
seqret sw:EFTS_AGRRK -outseq EFTS_AGRRK.fasta
И так для каждого организма. Далее все полученные файлы я соединила в один.
Вот полученный мной файл.
После этого я получила множественные выравнивания моих последовательностей с помощью команды:
muscle -in EFTS.fasta -out EFTS_aligned.fasta
Полученный при этом файл.
Для выполнения этого задания я воспользовалась следующей командой:
fprotpars -sequence EFTS_aligned.fasta
Скобочная формула приведена здесь.
А само неукорененное дерево можно посмотреть вот здесь.
Скобочная формула: ((((((EFTS_ECOLI,EFTS_ERWCT),EFTS_YERPE),EFTS_PSEAE),EFTS_RALPJ),
EFTS_RHOS4),EFTS_AGRRK);
Само неукорененное дерево:
+--EFTS_ECOLI
+--6
+--5 +--EFTS_ERWCT
! !
+--4 +-----EFTS_YERPE
! !
+--3 +--------EFTS_PSEAE
! !
+--2 +-----------EFTS_RALPJ
! !
1 +--------------EFTS_RHOS4
!
+-----------------EFTS_AGRRK
Полученное дерево полностью совпало с правильным.
fprotdist -sequence EFTS_aligned.fasta
была получена матрица расстояний: вот она.
AGRRK RHOS4 RALPJ PSEAE YERPE ECOLI ERWCT
AGRRK 0.000000 0.569769 0.980941 0.924960 0.818403 0.822304 0.854491
RHOS4 0.569769 0.000000 0.942776 0.877804 0.824210 0.864324 0.951312
RALPJ 0.980941 0.942776 0.000000 0.878947 0.857172 0.849837 0.857894
PSEAE 0.924960 0.877804 0.878947 0.000000 0.689033 0.635698 0.639645
YERPE 0.818403 0.824210 0.857172 0.689033 0.000000 0.236520 0.227639
ECOLI 0.822304 0.864324 0.849837 0.635698 0.236520 0.000000 0.066631
ERWCT 0.854491 0.951312 0.857894 0.639645 0.227639 0.066631 0.000000
Проверим теперь, насколько расстояния отклоняются от ультраметричности.
Свойство ультраметричности: "из трех расстояний между тремя объектами два всегда равны между собой и не меньше третьего".
d (A,B) <= max(d (A,C), d (B,C)) или Если d(A,B) > d (B,C), то d (A,C) = d (A,B) Для ECOLI=C, RALPJ=A, ERWST=B: d(ECOLI,ERWCT)=0.066631 d(ERWST,RALPJ)=0.857894 d(ECOLI,RALPJ)=0.849837 Свойство не выполняется.
Узнаем, насколько расстояния отклоняются от аддитивности
Для четырех последовательностей A,B,C,D: из 3х сумм 1) d(A,B) + d(C,D) 2) d(A,C) + d(B,D) 3) d(A,D) + d(B,C) две равны между собой и больше третьей. Для ERWCT=A, ECOLI=B, RHOS4=C, AGRRK=D: 1)d(ERWCT,ECOLI)+d(RHOS4,AGRRK)=0.066631+0.569769=0.6364 2)d(ERWCT,RHOS4)+d(ECOLI,AGRRK)=0.951312+0.822304=1.773616 3)d(ERWCT,AGRRK)+d(RHOS4,ECOLI)=0.854491+0.864324=1.718815 2) и 3) суммы больше 1), отличаются друг от друга на 0.054801.
Необходимо было получить две реконструкции дерева программой fneighbor, используя два алгоритма: UPGMA и Neighbor-Joining.
Для этого я последовательно применила две команды:
fneighbor -datafile efts_aligned.fprotdist -outfile efts.Neighbor-Joining.fneighbor -outtreefile efts.Neighbor-Joining.fneighbor.tree
fneighbor -datafile efts_aligned.fprotdist -outfile efts.UPGMA.fneighbor -outtreefile efts.UPGMA.fneighbor.tree -treetype u
Neighbor-joining:(неукорененное дерево)
Скобочная формула: (EFTS_RHOS4:0.29082,(EFTS_RALPJ:0.47740,(EFTS_PSEAE:0.35108, (EFTS_YERPE:0.11652,(EFTS_ERWCT:0.04115,EFTS_ECOLI:0.02548):0.08225):0.19624):0.05816):0.19958,EFTS_AGRRK:0.27895); +----------------EFTS_RHOS4 ! ! +----------------------------EFTS_RALPJ 1-----------4 ! ! +---------------------EFTS_PSEAE ! +--5 ! ! +------EFTS_YERPE ! +-----------3 ! ! +--EFTS_ERWCT ! +----2 ! +-EFTS_ECOLI ! +----------------EFTS_AGRRK remember: this is an unrooted tree! Between And Length ------- --- ------ 1 EFTS_RHOS4 0.29082 1 4 0.19958 4 EFTS_RALPJ 0.47740 4 5 0.05816 5 EFTS_PSEAE 0.35108 5 3 0.19624 3 EFTS_YERPE 0.11652 3 2 0.08225 2 EFTS_ERWCT 0.04115 2 EFTS_ECOLI 0.02548 1 EFTS_AGRRK 0.27895
Скобочная формула:
((EFTS_AGRRK:0.28488,EFTS_RHOS4:0.28488):0.15819,(EFTS_RALPJ:0.43048,
(EFTS_PSEAE:0.32740,(EFTS_YERPE:0.11604,(EFTS_ERWCT:0.03332,
EFTS_ECOLI:0.03332):0.08272):0.21136):0.10309):0.01259);
+-----------------EFTS_AGRRK
+--------3
! +-----------------EFTS_RHOS4
--6
! +-------------------------EFTS_RALPJ
+-5
! +-------------------EFTS_PSEAE
+-----4
! +------EFTS_YERPE
+------------2
! +-EFTS_ERWCT
+----1
+-EFTS_ECOLI
From To Length Height
---- -- ------ ------
6 3 0.15819 0.15819
3 EFTS_AGRRK 0.28488 0.44308
3 EFTS_RHOS4 0.28488 0.44308
6 5 0.01259 0.01259
5 EFTS_RALPJ 0.43048 0.44308
5 4 0.10309 0.11568
4 EFTS_PSEAE 0.32740 0.44308
4 2 0.21136 0.32704
2 EFTS_YERPE 0.11604 0.44308
2 1 0.08272 0.40976
1 EFTS_ERWCT 0.03332 0.44308
1 EFTS_ECOLI 0.03332 0.44308
Можно сделать следующий вывод: Нетривиальные ветви настоящего дерева, дерева, построенного программой fprotpars, а также этих деревьев совпадают.