Поиск в нуклеотидных банках по аннотации. Выравнивание геномов.
Для построения карт локального сходства были выбраны геномы вирусов из порядка Mimiviridae: 1. Acanthamoeba polyphaga mimivirus (strain Kroon) 2. Acanthamoeba polyphaga moumouvirus Эти вирусы являются одними из немногих дцДНК-вирусов и имеют объёмный (более миллиона пар оснований) сложноструктурированный геном, содержащий гомологи некоторых генов бактерий, архей и эукариот. Поиск осуществлялся с помощью NCBI Genomes по запросу «Viruses». Я установила фильтры по уровню сборки (Chromosome и выше) и по аннотации (Annotated by NCBI RefSeq). Из полученных сборок был выбран геном Acanthamoeba polyphaga mimivirus. При сравнении его генома с помощью megablast с вирусами того же семейства на DotPlot была в основном одна прямая линия, поэтому в качестве второго объекта был выбран Acanthamoeba polyphaga moumouvirus. Далее было проведено выравнивание полных геномов этих вирусов с помощью magablast (word size 28) и blastn (word size 11). Идентификаторы геномов в RefSeq: NC_075035.1 - Acanthamoeba polyphaga mimivirus NC_020104.1 - Acanthamoeba polyphaga moumouvirus Полученные карты локального сходства (DotPlot) представлены на рисунках 1, 2.
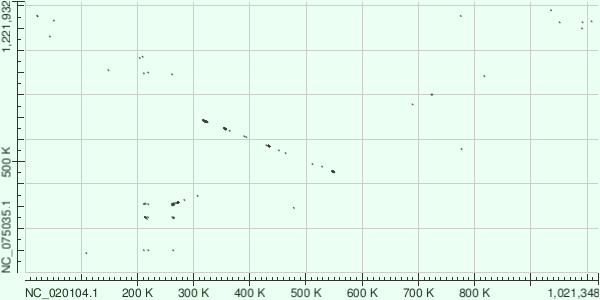
При использовании megablast почти не было обнаружено схожих участков, так как я решила рассматривать относительно неблизкородственные вирусы, однако результат blastn сильно отличается – эта карта DotPlot описана ниже.
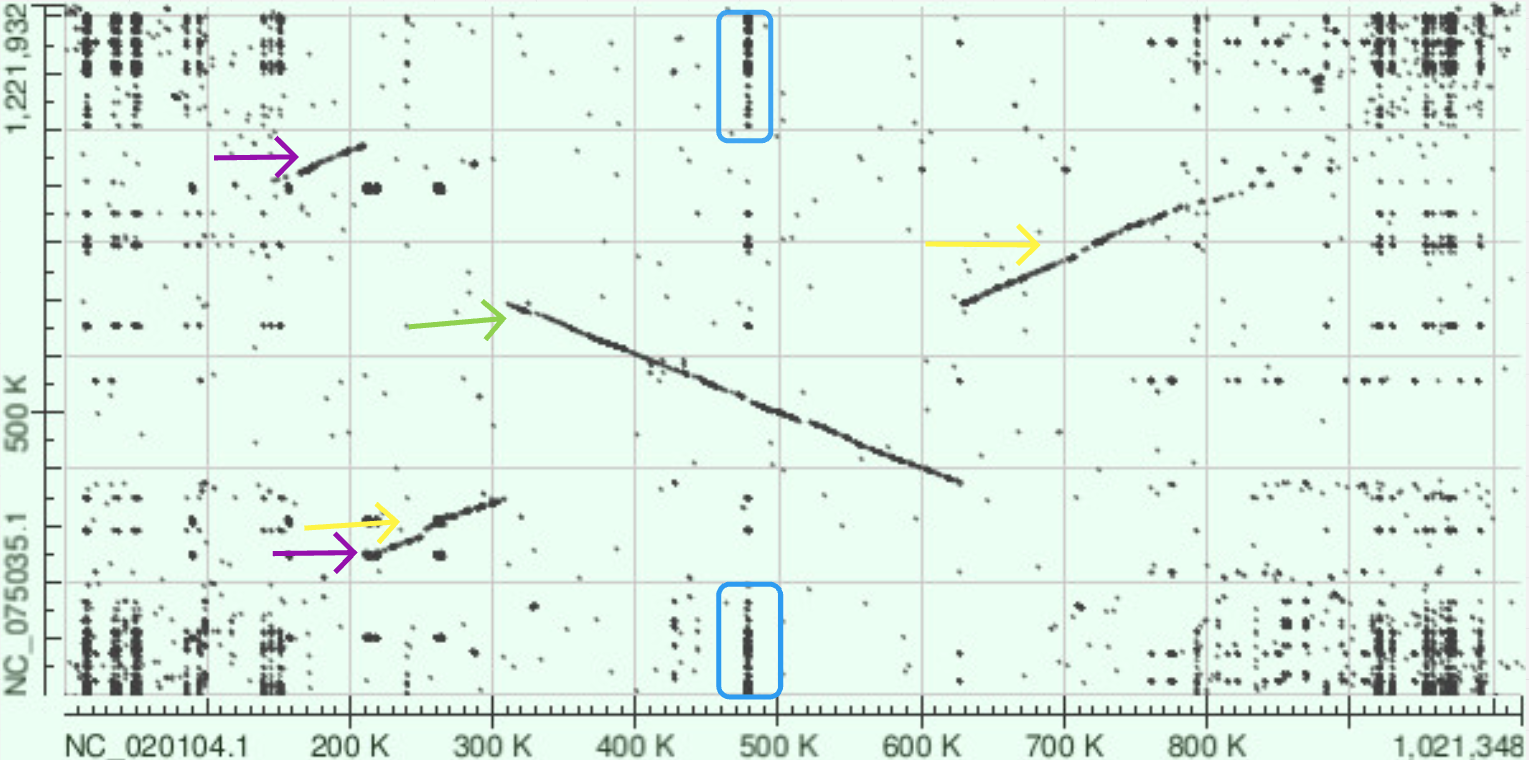
DotPlot полученный алгоритмом blastn (см. Рис. 3) позволяет провести анализ выравнивания выбранных вирусных геномов, так как способен обнаруживать участки с меньшим сходством, что больше подходит для сравнения геномов с меньшим процентом идентичности.
Описание DotPlot
Присутствует большое количество схожих фрагментов, хотя многие участки представляют собой совокупность отдельных точек, не лежащих точно на одной прямой, то есть длинных участков с идеальным совпадением практически нет. Самая четкая и длинная линия (на участке 310К-620К) находится на побочной диагонали, значит у вирусов были выбраны разные цепи. (зеленая стрелка) Крупные перестройки генома заметны на участках 200K-300K и 630K-800K: линии, расположенные на главной диагонали – произошли инверсии. Также это соответствует транслокациям – фрагменты сдвинуты относительно основной линии выравнивания. (желтые стрелки) Двумя фиолетовыми стрелками обозначены линии, расположенные друг над другом на главной диагонали – это указывает на инверсию и дупликацию (в геноме Acanthamoeba polyphaga mimivirus) достаточно большого фрагмента. В начале и в конце выравнивания, а также в середине (обозначено голубым) есть вертикальные линии/группы точек. Возможно, здесь участки генома Acanthamoeba polyphaga moumouvirus выровнялись с повторами в геноме Acanthamoeba polyphaga mimivirus, причем эти повторы есть и в конце, и в начале его генома (линии симметричны сверху и снизу). Еще на карте локального сходства достаточно много отдельных небольших точек, которые соответствуют повторам в последовательностях.