Нуклеотидный BLAST
Поиск гена, кодирующего δ-субъединицу АТФ-синтазы
В файлах с последовательностями белков и аннотированным геномом тринадцатиполосого суслика была найдена следующая информация о нужном белке: δ-субъединице АТФ-синтазы. Идентификатор белка: XP_005332020.1 Файл с последовательностью белка Идентификатор нуклеотидной записи: NW_024405118 (к ней относится ген, кодирующий данный белок). Файл с последовательностью нужного гена с небольшой окрестностью (см. рис. 1).
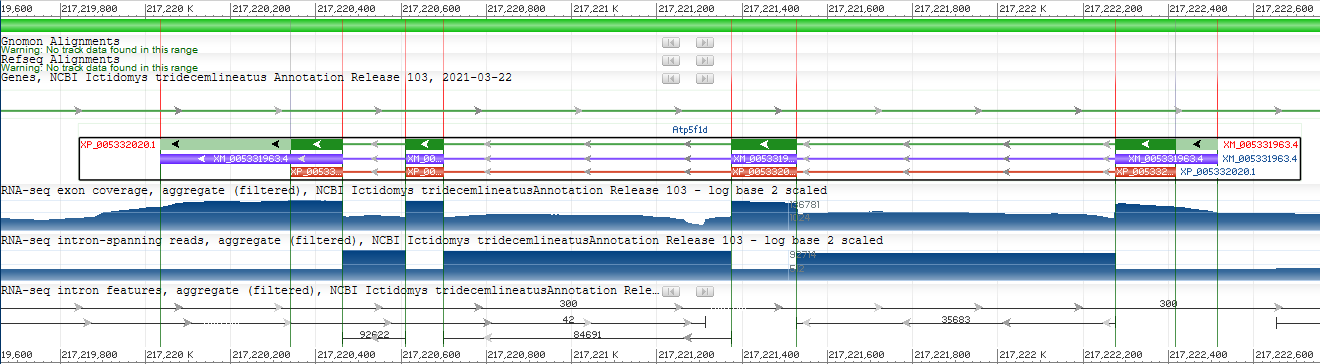
Разные варианты BLAST для фрагмента ДНК
Тринадцатиполосый суслик (Ictidomys tridecemlineatus) относится к вторичноротым, поэтому в качестве достаточно удаленного таксона для поиска в нем гомологичных интересующему белку последовательностей с помощью BLAST я выбрала семейство Пчёлы (Apoidea).
1. Blastn
Blastn осуществляет поиск нуклеотидных последовательностей, схожих с заданной по нуклеотидной базе данных, используется для поиска гомологичных последовательностей. В отличие от megablast он позволит также найти варианты с относительно меньшим процентом совпадения (поэтому он был выбран, ведь организмы не близкородственные), но его работа занимает немного больше времени. Параметры blastn: База данных для поиска: refseq_genomes. Число сборок в ней, входящих в выбранный таксон: 28 (число геномов, по которым проводился поиск) Word size: 7 - находок 79, но у них небольшие score и query coverage. (см. рис. 2 и текстовую выдачу blastn по ссылке) Word size: 11 - 4 находки, и они не самые лучшие из полученных с word size 7. (см. рис. 3 и текстовую выдачу blastn по ссылке)
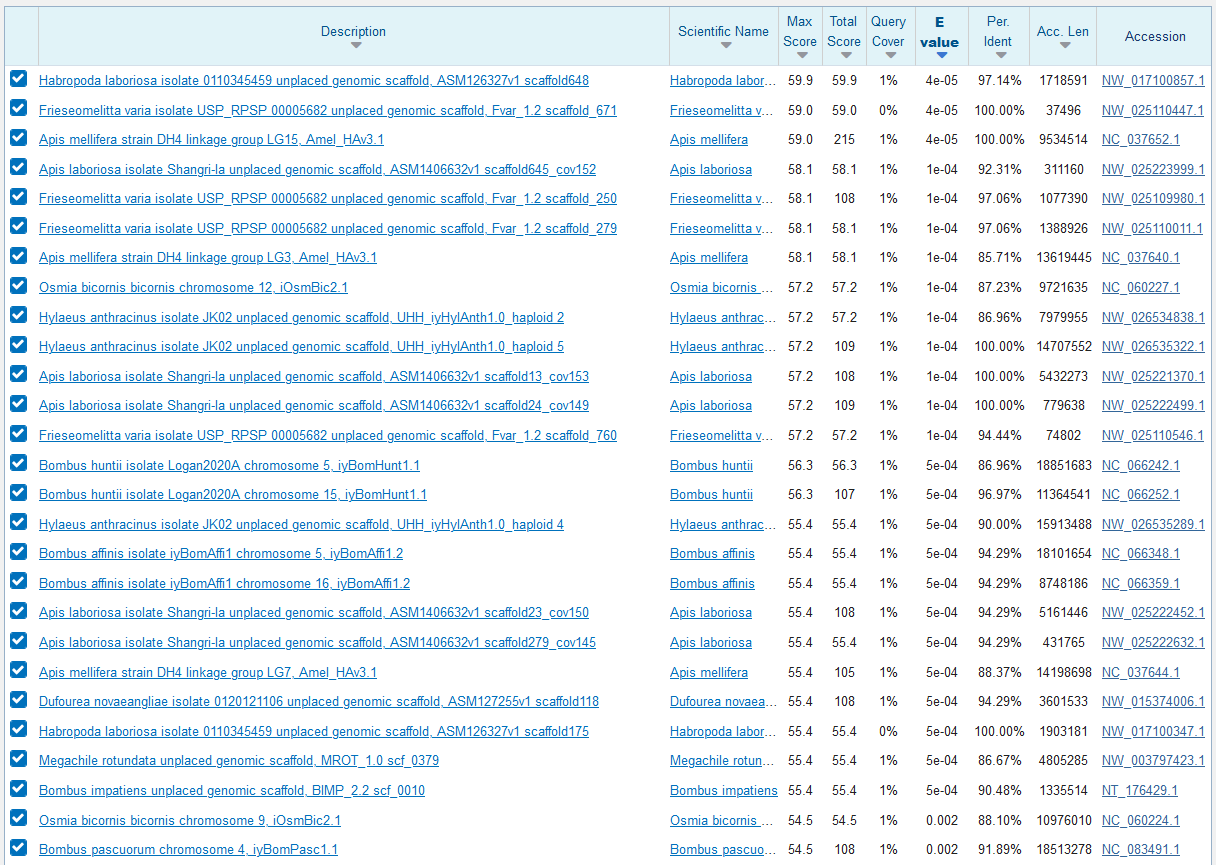
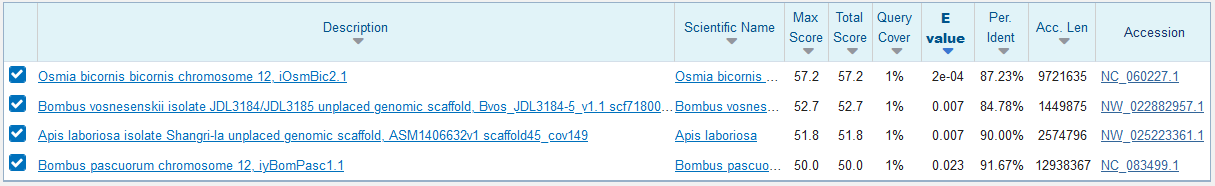
2. Tblastx
На вход тоже дается нуклеотидная последовательность, но поиск происходит по транслированной нуклеотидной базе данных. С помощью этого алгоритма должно получится больше находок, так как искомая последовательность сначала транслируется, что учитывает возможность получения одного белка из разных нуклеотидных последовательностей. Этот алгоритм был выбран, так как для использования tblastn нужно сначала транслировать последовательность (на вход дается уже последовательность белка). Параметры tblastx: База данных для поиска: refseq_genomes. Word size: 3 – 39 находок. У них больше процент покрытия – от 5 до 15% (при использовании blastn было 0-1%), значения Score тоже заметно выше, e-value меньше. (см. рис. 4 и текстовую выдачу tblastx по ссылке). Таким образом с помощью tblastx было получено больше значимых находок.
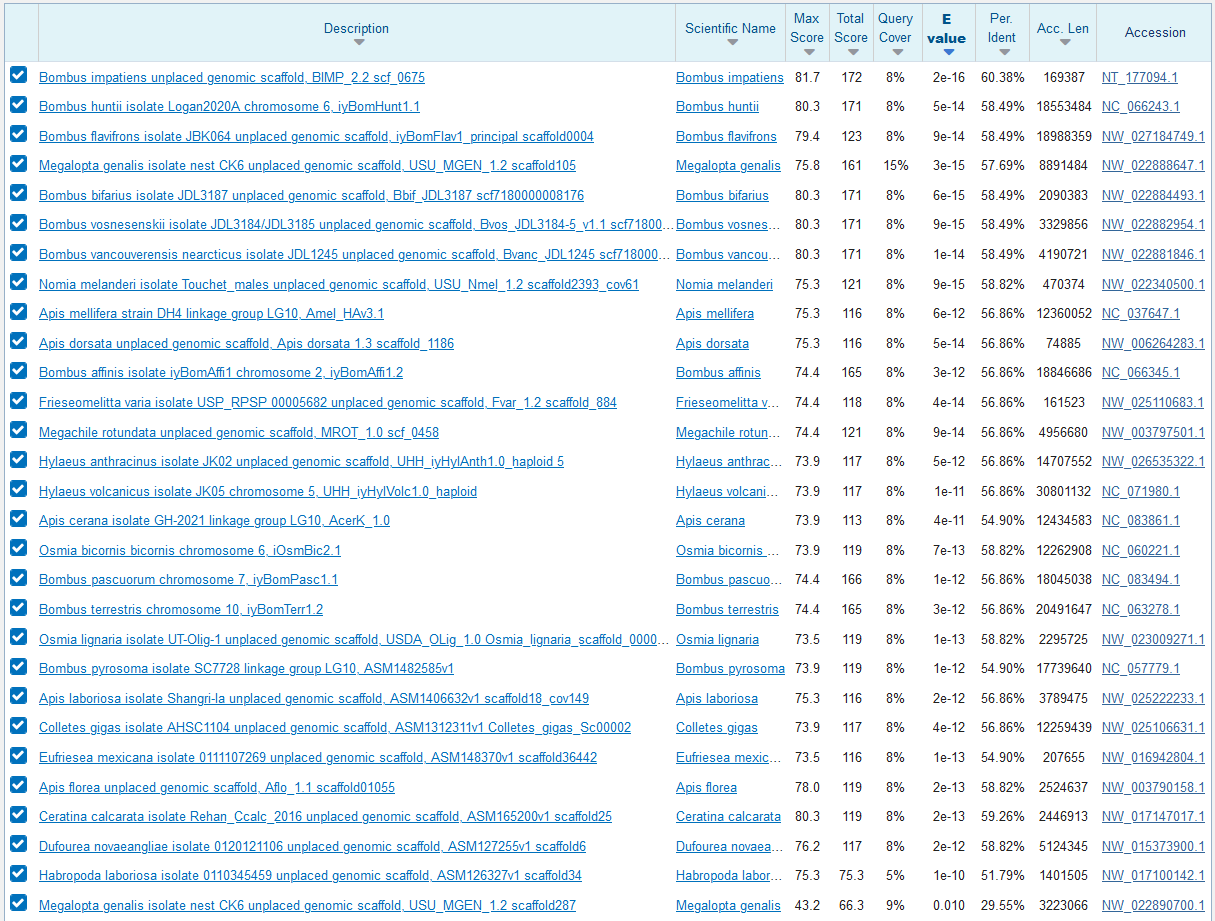
Поиск в геноме Ictidomys tridecemlineatus генов основных рибосомальных РНК по далекому гомологу
1. Создание локальной базы данных
Команда для индексации последовательности генома:
makeblastdb -in GCF_016881025.1_HiC_Itri_2_genomic.fna -dbtype nucl
2. Локальный поиск BLAST
Локальный поиск проводился по последовательностям 16S рРНК и 23S рРНК Escherichia coli. 16S рРНК входит в состав малой субъединицы рибосомы 70S. Она узнает последовательность Шайна-Дальгарно на мРНК, связывает некоторые трансляционные факторы, участвует в стабилизации трехмерной структуры рибосомы и связывании большой и малой субъединиц. 23S рРНК входит в состав большой субъединицы рибосомы 70S, является ключевым компонентом пептидилтрансферазного центра рибосомы, участвует в стабилизации трехмерной структуры рибосомы. Был выбран blastn, поскольку поиск проводился по не кодирующим белок нуклеотидным последовательностям, и организмы не близкородственные. Использованные команды:
blastn -task blastn -query 16S.fasta -db GCF_016881025.1_HiC_Itri_2_genomic.fna.fna -out blastn_16S.out -evalue 0.05 -outfmt 7
blastn -task blastn -query 23S.fasta -db GCF_016881025.1_HiC_Itri_2_genomic.fna.fna -out blastn_23S.out -evalue 0.05 -outfmt 7
Результаты: 16S рРНК- нет находок даже с самыми разными word size и e-value. 23S рРНК- 6 hits, но 4 гомолога (файл с результатами). Эти гомологи находятся в митохондриальном геноме тринадцатиполосого суслика (так как там вероятнее будут содержаться гены, кодирующие компоненты рибосомы 70S). Действительно, в файле со сборкой его генома указано, что эти записи относятся либо к mitochondrion, либо к unplaced genomic scaffold.