Мотивы в белках, паттерны и PSSM для их поиска.
1. Поиск консервативных мотивов в выравнивании.
Был выбран соответствующий критериям домен - PF10681 (Chaperone for protein-folding within the ER, fungal); short name - Rot1. Белки этого семейства - шапероны в эндоплазматическом ретикулуме грибов, они временно взаимодействуют с развернутыми белками, ингибируя их самоагрегацию и поддерживая правильную борку. Далее было скачано выравнивание seed, состоящее из 59 последовательностей. Последовательностей, идентичных более, чем на 90% не было. После постепенного снижения Above identity threshold был найден мотив, имеющий визуально высокое информационное содержание, он встречается в 56 последовательностях. Его координаты: 99-108 (см. Рис.1) паттерн Jalview: DGR[QKM][LQI].S.[PR]C паттерн Prosite: D-G-R-[QKM]-[LQI]-x-S-x-[PR]-C
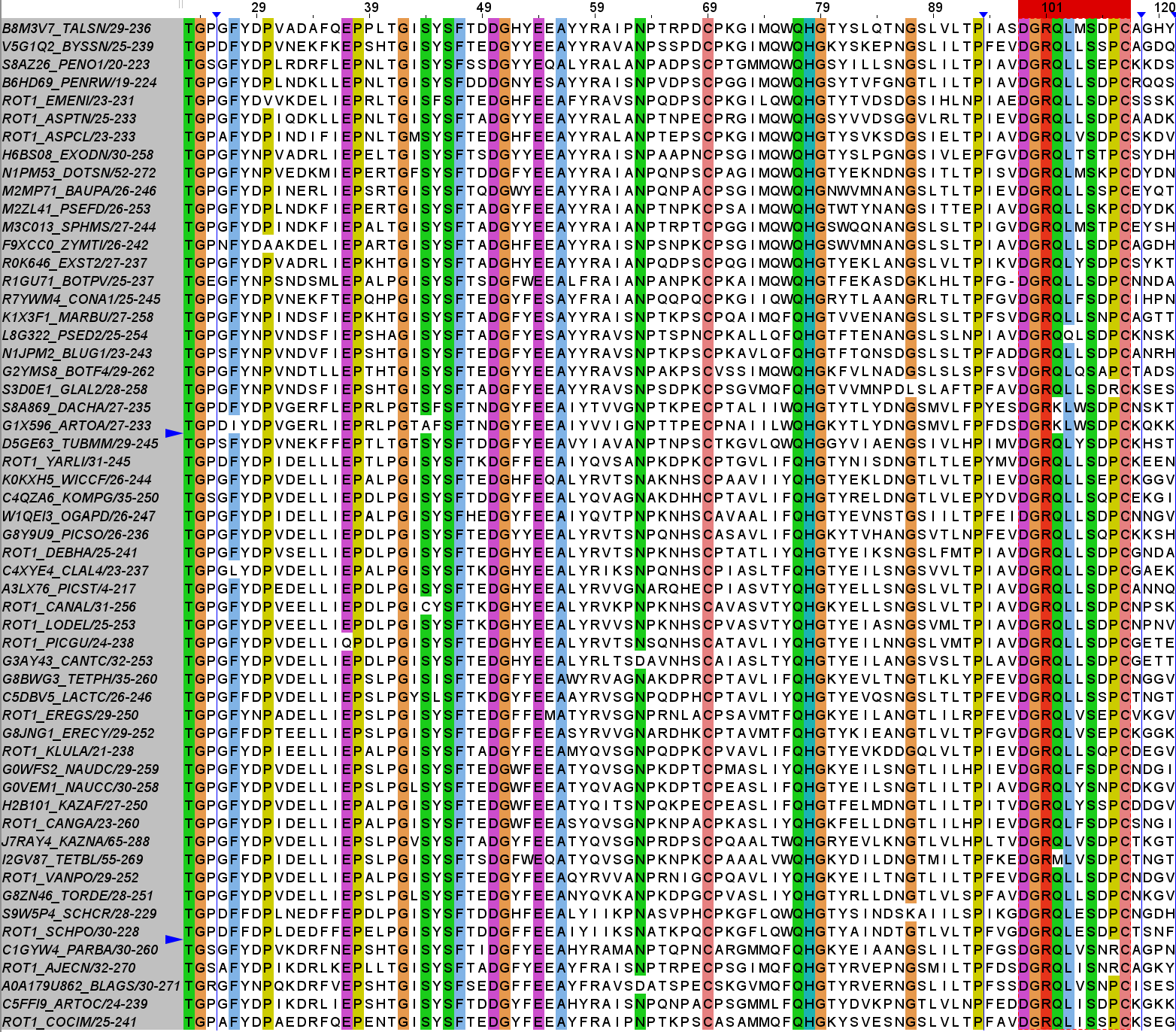
Был запущен поиск паттерна при помощи сервиса ScanProsite (база данных Swiss-Prot). Нашлось 22 совпадения в 22 последовательностях, все эти белки являются белками ROT1 (это также Short name выбранного домена) из разных грибов. Последовательности были скачаны и выровнены программой Muscle. Мотивы выровнялись (см. Рис. 2).

Таким образом, найденный мотив консервативен для данного семейства белков, является необходимой частью их структуры и, возможно, участвует в шапероновой активности.
2. Поиск мотива, специфичного для одной клады филогенетического дерева.
С помощью метода NJ в Jalview было построено филогенетическое дерево для данного набора последовательностей из выравнивания seed. Была выбрана клада, состоящая из 10 последовательностей (см. Рис. 3)
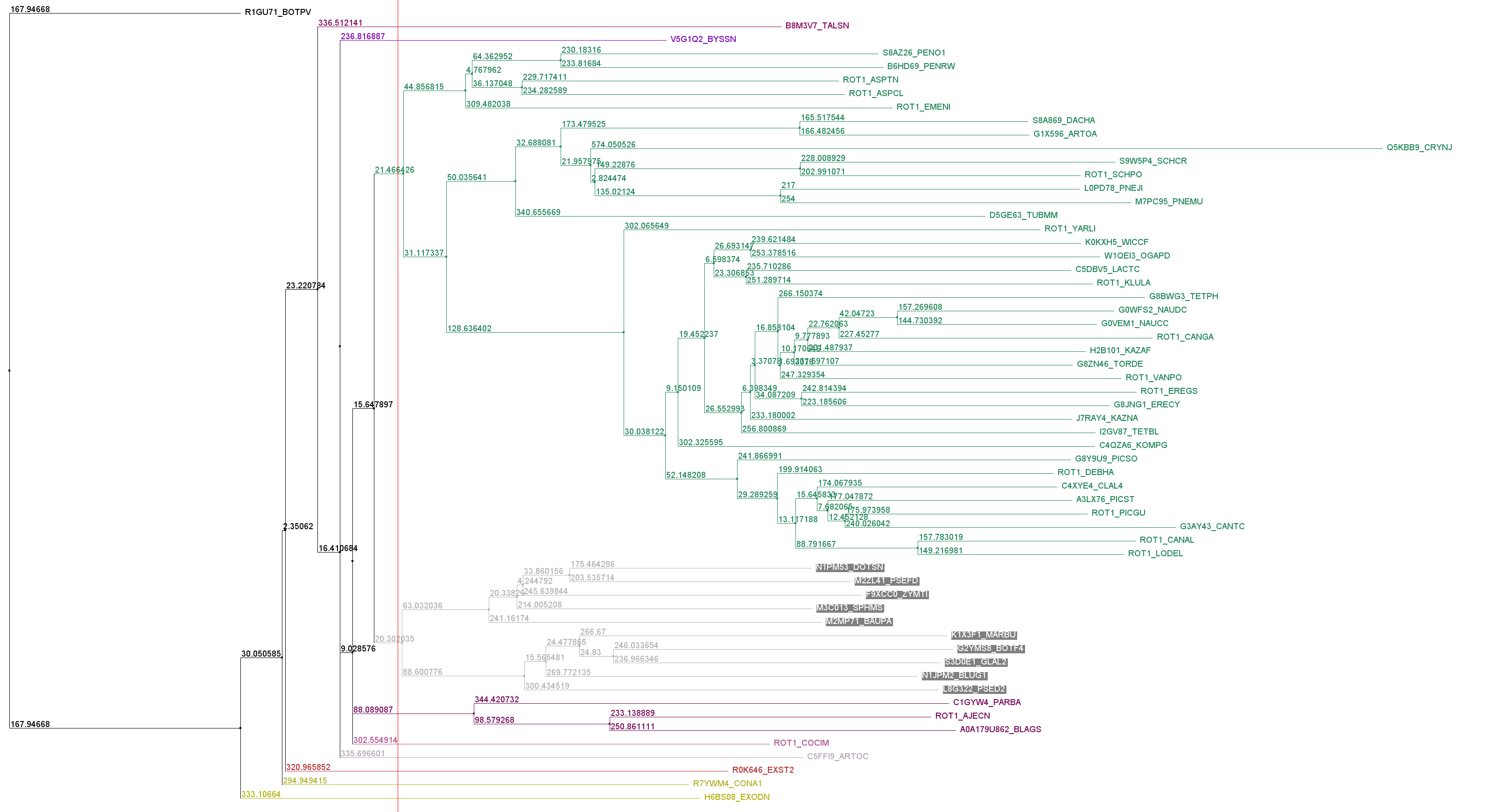
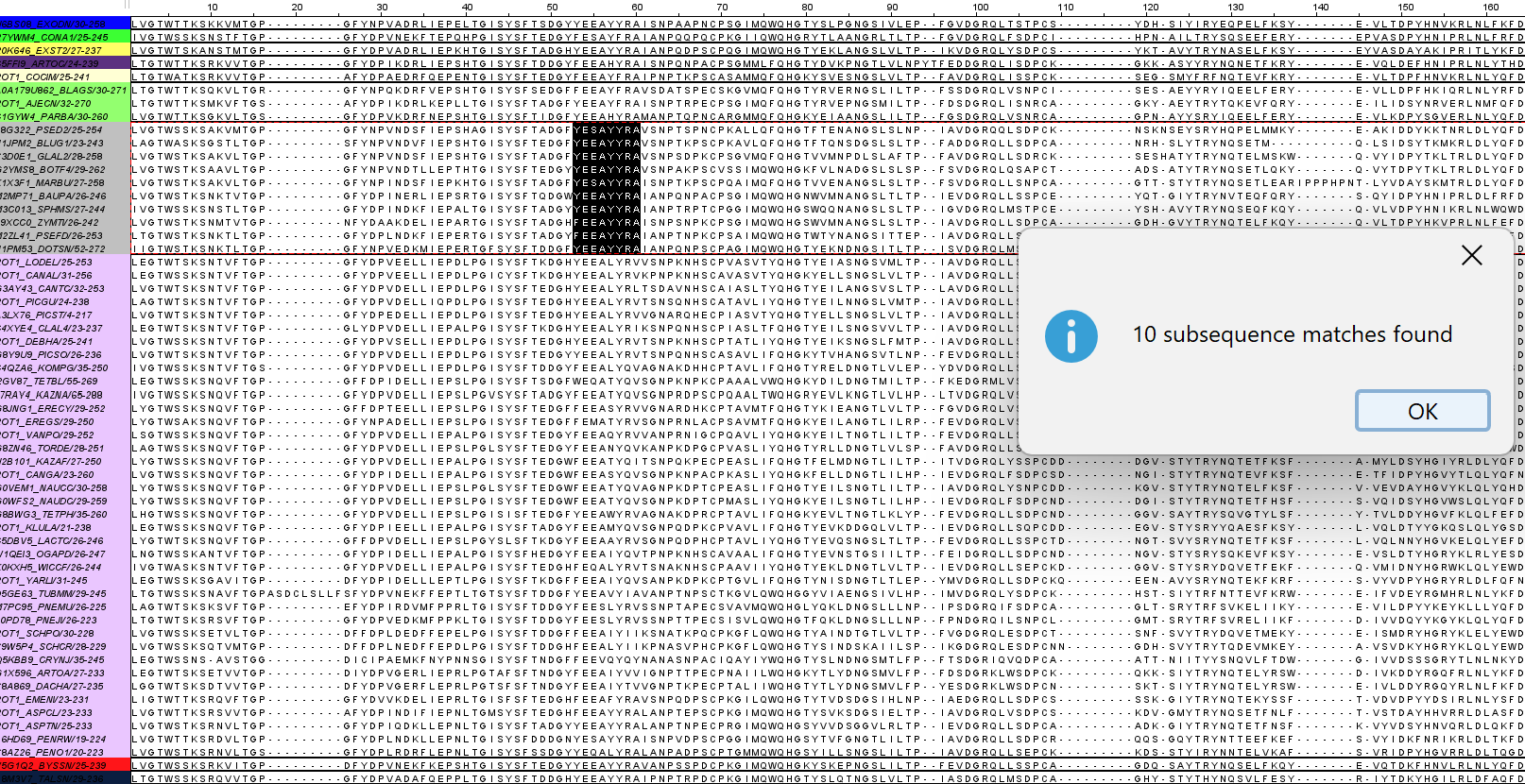
Далее выравнивание этой клады было вынесено в отдельное окно и найден наиболее консервативный мотив: Координаты: 53-60 паттерн Jalview: [YF]E[ES]AYYRA При поиске мотива во всем выравнивании он был найден только в выбранных 10 последовательностях (см. Рис. 4), следовательно, мотив специфичен для данной клады.
3. PSI-BLAST
Выбранный AC - C4Z088: Probable septum site-determining protein MinC из бактерии Lachnospira eligens. Это ингибитор клеточного деления, который блокирует сборку протофиламентов FtsZ, которые необходимы для формирования Z-кольца в месте деления клетки, также этот белок предотвращает полимеризацию FtsZ. Был запущен алгоритм PSI-BLAST по базе данных Swiss-Prot. Результаты представлены в Таблице 1.
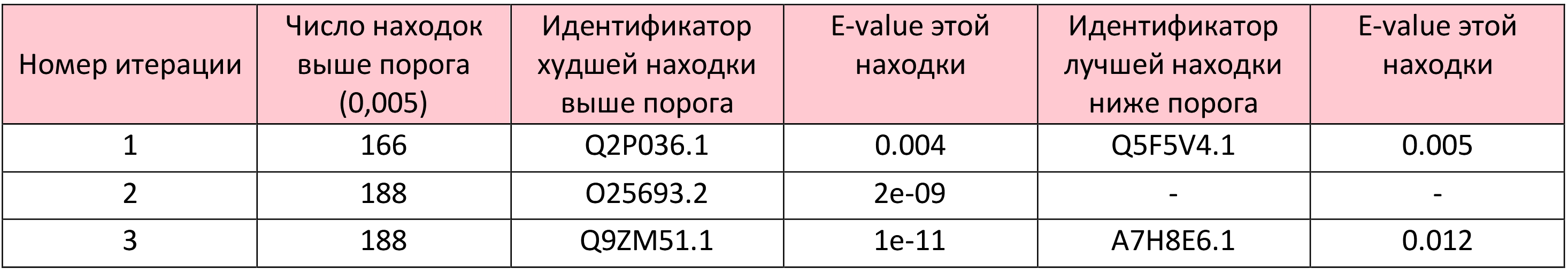
На третьей итерации не нашлось новых белков - удалось стабилизировать результат. Разница e-value между худшей находкой выше порога и лучшей находкой ниже порога достаточно большая, следовательно, найдено семейство гомологичных белков; все из бактерий. Я просмотрела названия белков – все они имеют ту же функцию, что и C4Z088. Таким образом, PSI-BLAST хорошо выделил семейство гомологичных белков.
4. Поиск de novo мотивов с помощью MEME в выборке последовательностей с доменом из SwissProt.
Я выбрала последовательности белков с доменом PF10681 из грибов класса Dothideomycetes (Fungi; Ascomycota; Dothideomycetes) – 191 последовательность. Далее последовательности были выровнены. После удаления схожих последовательностей их осталось 58 (файл с последовательностями). Для поиска мотивов была запущена программа MEME. Команда:
meme doth_red.fasta -o meme.res -protein -mod anr -minw 4 -maxw 10 -nmotifs 4Выдача meme Один из найденных мотивов (см. Рис. 5) очень похож на тот, который был описан в первом задании (визуально это также заметно, если сравнить Рис. 6 и Рис. 1). Также, у этой находки достаточно низкое e-value, что подтверждает значимость данного мотива
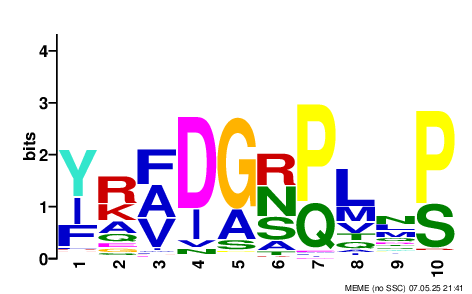

Далее для поиска этого мотива была запущена программа FIMO. Команда:
fimo --oc fimo_do -motif YRFDGRPLNP -thresh 0.001 meme.res/meme.txt doth_red.fastaВыдача fimo – получилось 195 находок в 58 последовательностях, то есть этот мотив почти всегда встречался несколько раз. Возможно, это связано с тем, что в этом мотиве много вариабельных позиций, и в одной последовательности находились несколько вариантов мотива, как видно из выдачи.
5. Оценка представленности сайта GATC в геноме бактерии.
Рассматриваемая бактерия - Tenacibaculum finnmarkense. Для вычисления контрастов была использована программа cbcalc. На вход подается геном бактерии и список возможных 24х четырехнуклеотидных сайтов без повторений. Команда:
cbcalc -s sites.txt -M -o res.tsv genome.fna-s sites.txt файл с сайтами для анализа -M – использование метода Карлина -o res.tsv -выходной файл По результату была построена гистограмма (см. Рис. 7)
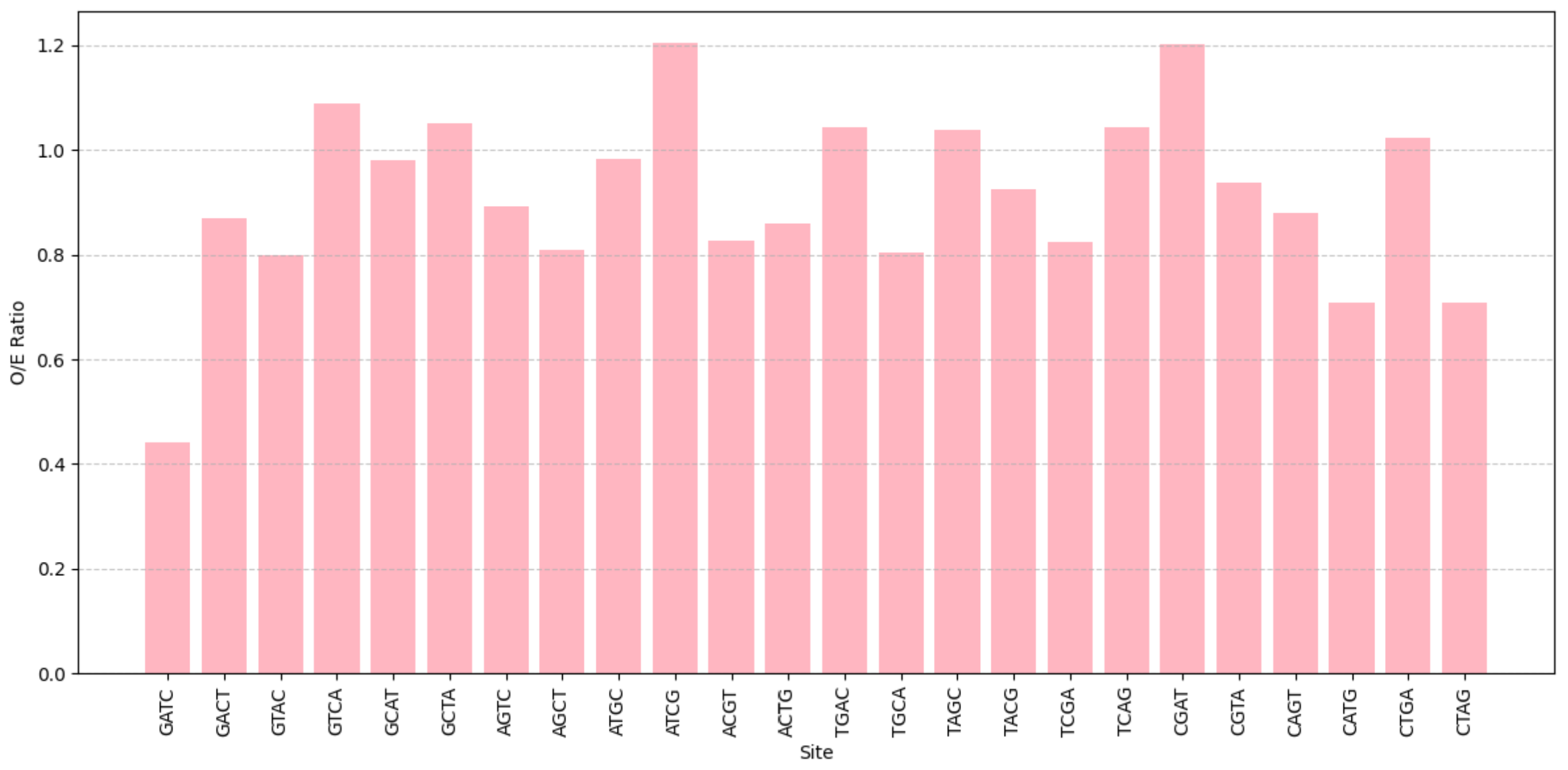
Из гистограммы видно, что самые распространенные сайты – это ATCG и CGAT. Возможно, они являются сайтами метилирования (или узнаются другими ферментами) у Tenacibaculum finnmarkense. Распространенный у бактерий сайт метилирования GATC недопредставлен – отношение obs/exp = 0.44 значит, что наблюдаемая частота сайта составляет примерно 44% от ожидаемой. Возможно, это связано с наличием у Tenacibaculum finnmarkense таких рестриктаз, которые распознают именно этот сайт (и разрезают ДНК) и чтобы защитить геном от своих ферментов рестрикции представленность сайта GATC сильно понижена.