Алгоритмы филогенетической реконструкции
Выравнивание цитохромов B из выбранных животных было построено с помощью сайта Uniprot. Затем была проведена реконструкция дерева программой FastME (оценка эволюционных расстояний с помощью модели p-distance (Рис.2) и модели MtREV (Рис.3)) и программой IQ-Tree. (Рис.4)
Сравнение деревьев
Все виды, составляющие эти деревья, относятся к Млекопитающим.
FastME
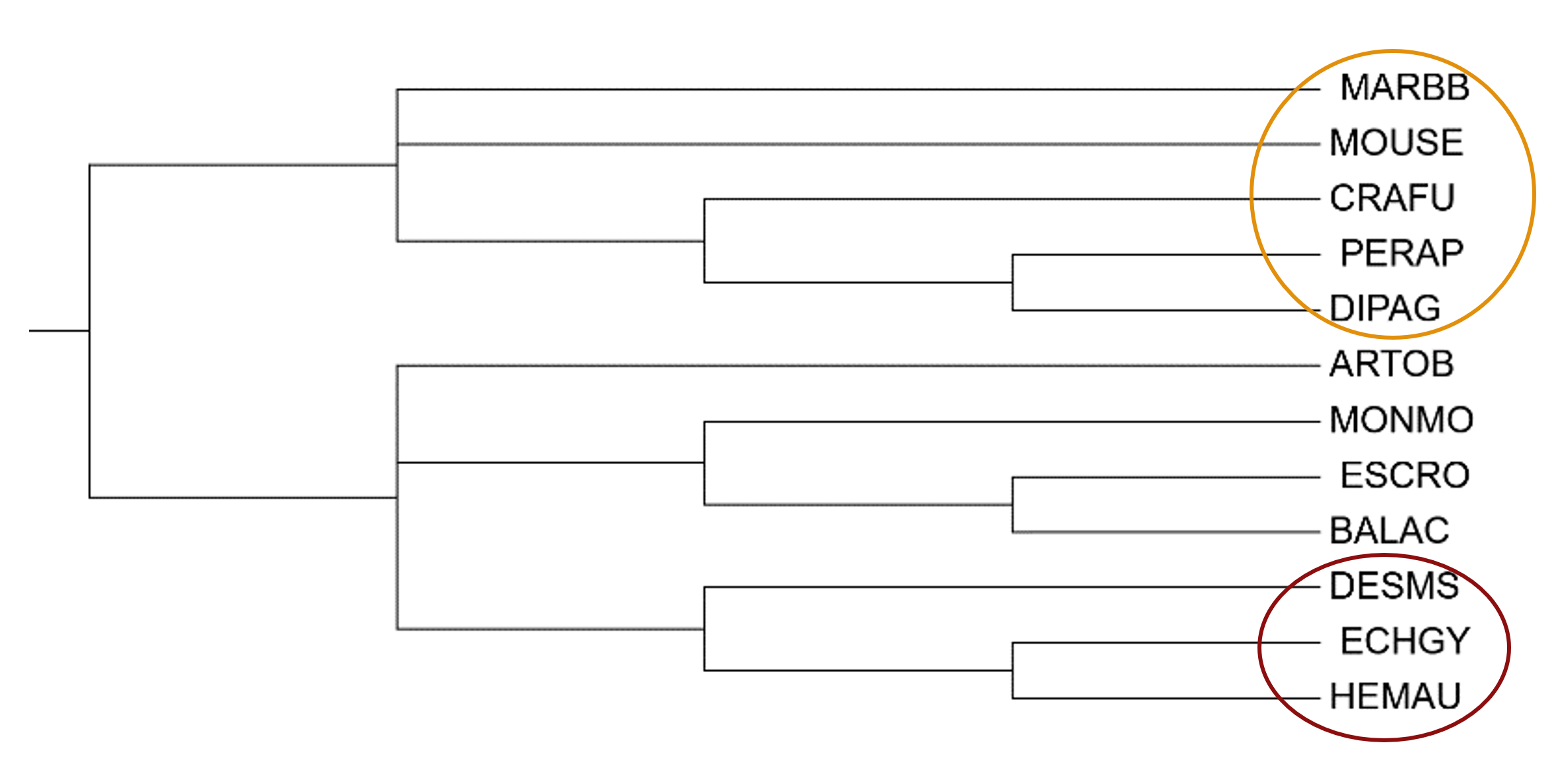
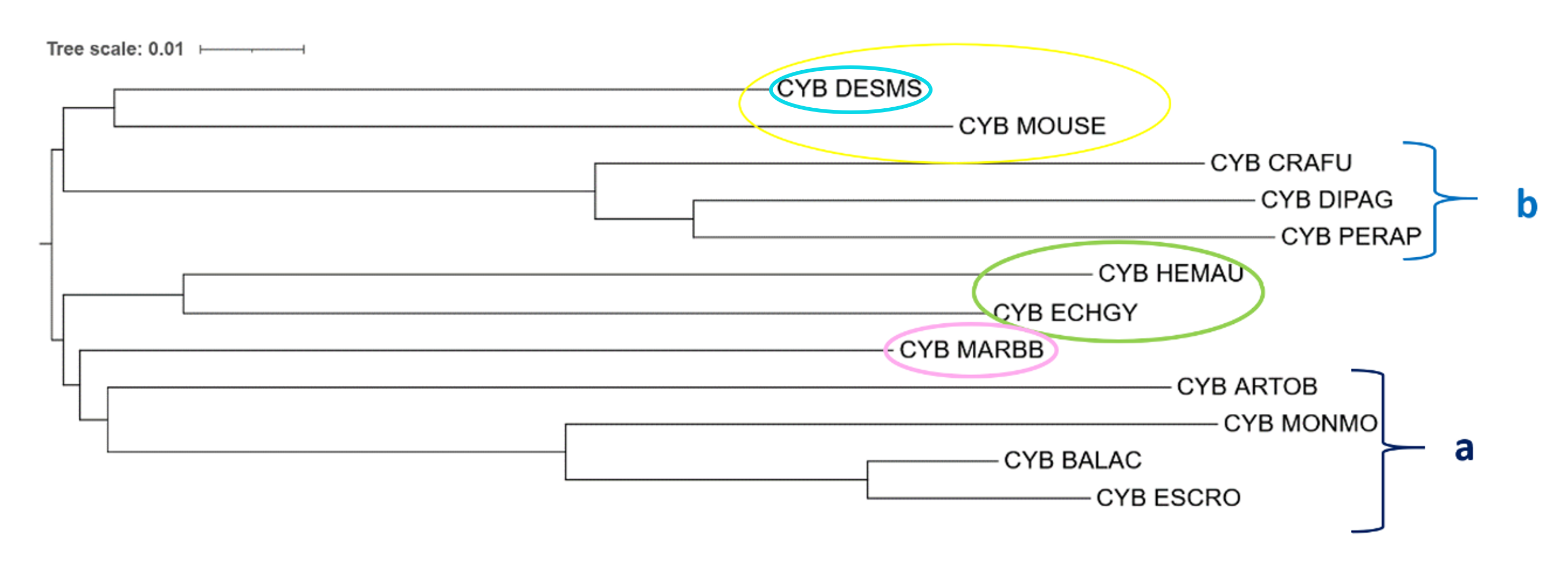
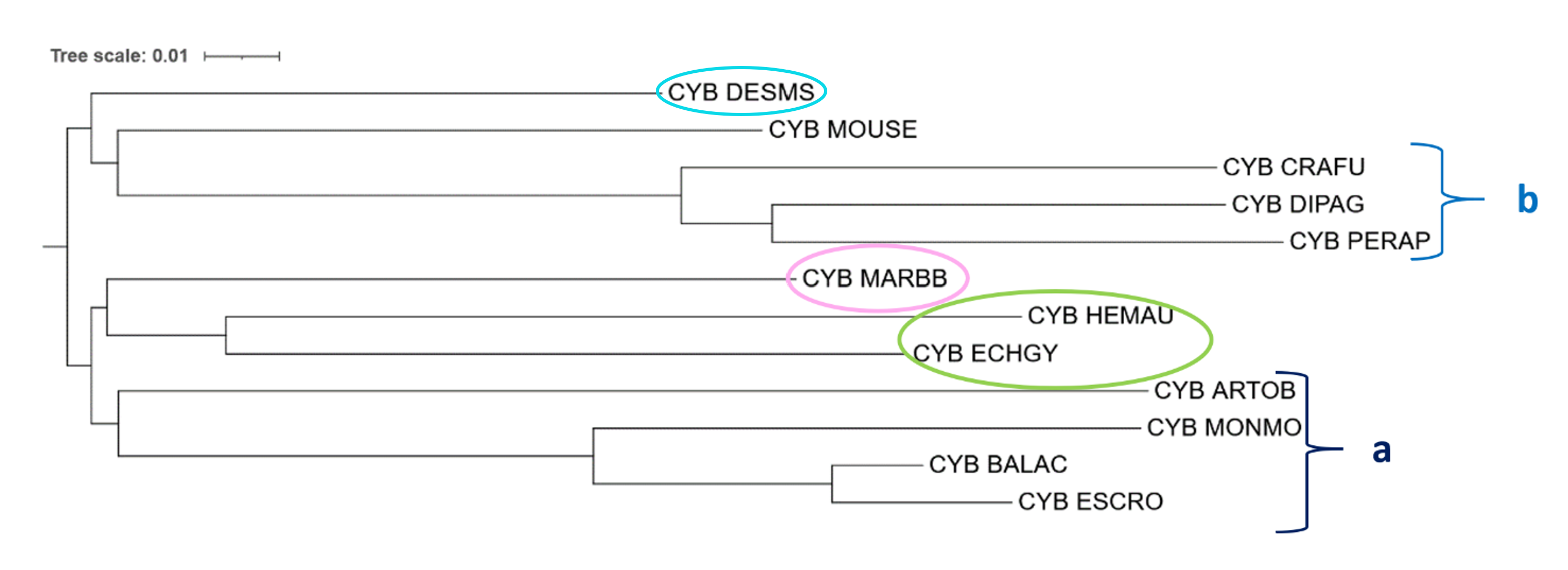
На рисунках 2 и 3 фигурными скобками выделены совпадающие клады (a и b), полученные FastME независимо от модели. Деревья, построенные программой FastME с помощью разных моделей оценки эволюционных расстояний, отличаются длинами ветвей. Можно заметить, что эти деревья существенно отличаются от дерева, построенного на основе NCBI Taxonomy: 1) Ошибочно не реконструирована ветвь, включающая Eulipotyphla (Насекомоядных): HEMAU (Ушастый ёж), ECHGY (Гимнура) и DESMS (Выхухоль) – выделена темно-красным. На деревьях, построенных программой FastME есть клада из HEMAU и ECHGY (выделена зеленым), но DESMS (выделена голубым) расположена неверно: в случае модели p-distance DESMS объединена в кладу с MOUSE (выделена желтым), сестринскую с кладой b; а в случае модели MtREV DESMS является сестринской к кладе из 4х видов Грызунов (MOUSE + клада b). 2) Ошибочно не реконструирована ветвь, включающая Rodentia (Грызунов): MOUSE (Домовая мышь), MARBB (Байбак), CRAFU (Дымчатый суслик), DIPAG (Прыткая кенгуровая крыса), PERAP (Аризонский мешотчатый прыгун) – выделена оранжевым. Верно реконструирована клада b, но MARBB (выделен розовым), находится совсем в другой части дерева в случае обоих моделей - p-distance: (MARBB + a) составляют кладу, сестринскую к кладе из Насекомоядных (HEMAU и ECHGY); MtREV: MARBB является сестринским к кладе из Насекомоядных (HEMAU и ECHGY). Также в дереве, использующем модель MtREV, MOUSE верно является сестринским видом к кладе b, но в случае p-distance MOUSE является сестринской к кладе b только в составе клады, выделенной желтым, что описано выше. Таким образом, похоже, что при использовании программы FastME можно условно сказать, что DESMS (Выхухоль) и MARBB (Байбак) поменялись местами относительно того, к каким видам они на самом деле родственны.
IQ-Tree

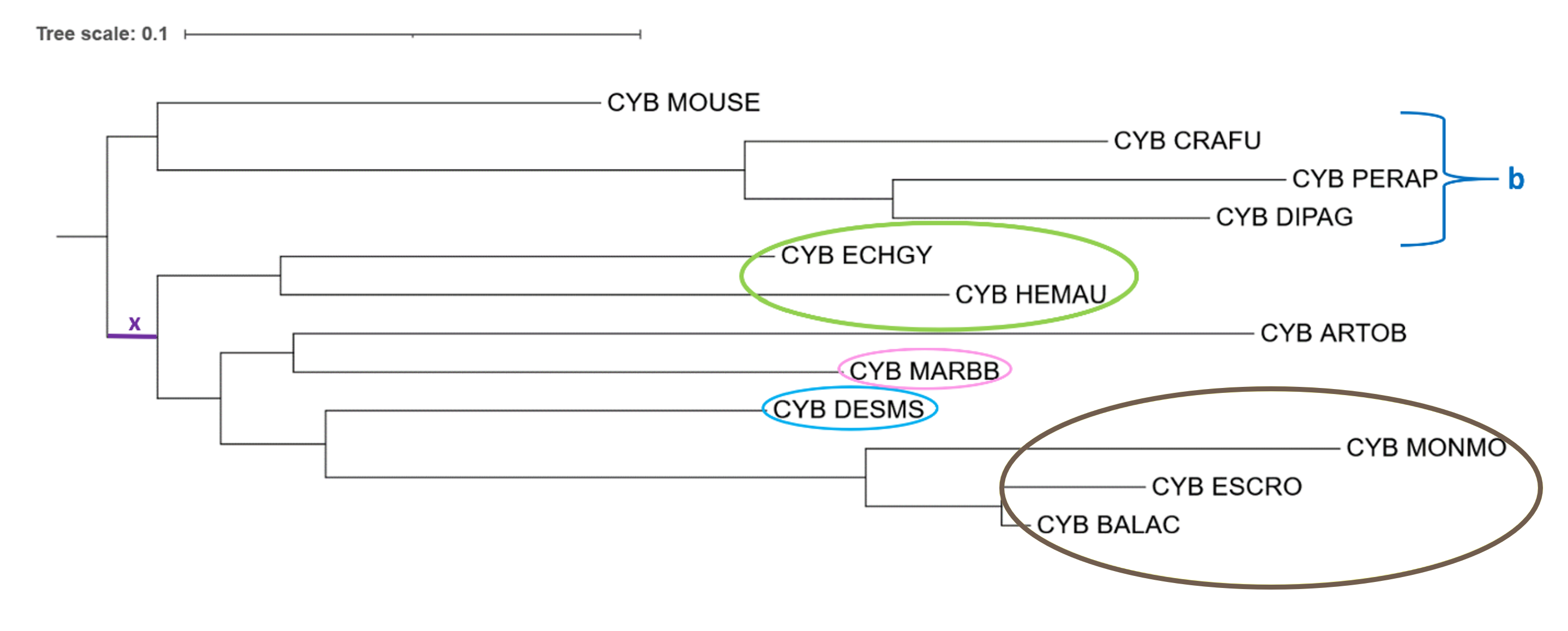
Дерево, построенное программой IQ-Tree сильно отличается по эволюционным расстояниям в сравнении с использованием программы FastME.
Отличия от дерева, основанного на NCBI Taxonomy: 1) Тоже ошибочно не реконструирована ветвь, включающая Eulipotyphla (Насекомоядных) – выделена темно-красным. Есть клада из HEMAU и ECHGY (выделена зеленым), но DESMS является сестринским видом не к этой кладе, а к кладе из 3х видов Cetacea (Китообразные) – выделена коричневым (включает виды MONMO (Нарвал), BALAC (Малый полосатик), ESCRO (Серый кит)). Если сравнивать с деревьями, построенными FastME, здесь положение DESMS ближе к правильному – этот вид не находится в составе вклады, включающей виды Rodentia, как в случает с FastME, а находится в составе клады из видов, отходящих от ветви x. 2) Тоже ошибочно не реконструирована ветвь, включающая Rodentia (Грызунов) - выделена оранжевым. Верно реконструирована клада b, а также расположение MOUSE, но MARBB (Байбак) находится в другой части дерева – составляет кладу с ARTOB (Темный фруктоядный листонос), которая в свою очередь является сестринской с кладой Cetacea+DESMS. Таким образом, при использовании программы IQ-Tree, расположение вида MARBB (Байбак) сильнее всего отличается от верного (на дереве NCBI Taxonomy).
Итог по всем реконструкциям:
- Можно рассмотреть 2 клады на исходном дереве NCBI Taxonomy: выделенная оранжевым и состоящая из 7ми оставшихся видов. В деревьях, построенных программой FastME, DESMS (Выхухоль) и MARBB (Байбак) находятся не в своей кладе; при использовании программы IQ-Tree DESMS (Выхухоль) находится в своей кладе. - На всех деревьях верно реконструированы клада b, клада из Cetacea (MONMO, BALAC, ESCRO) и клада из HEMAU и ECHGY, что составляет не очень большую часть дерева.