Филогенетические деревья, укоренение, сравнение деревьев, бутстрэп
1. Реконструкция дерева по нуклеотидным последовательностям.
Была проведена реконструкция дерева по последовательностям малой РНК митохондриальных рибосом (12S rRNA). Для 5 видов (MARBB, DIPAG, PERAP, CRAFU, ARTOB) не было последовательности митохондриального генома в ENA, поэтому далее я использовала геномы других родственных видов (самые близкие из тех, митохондриальные геномы которых есть в ENA). 1. Marmota bobak (MARBB): я взяла геном самого близкого родственника[1] - Marmota sibirica (Монгольский сурок) - MARSI. 2. Dipodomys agilis (DIPAG): находились короткие неаннотированные последовательности. Только с помощью поиска по семейству Dipodomyinae были найдены 2 представителя, я использовала геном более близкого родственника[2] - Dipodomys merriami (Кенгуровый прыгун Мерриама) - DIPME. 3. Perognathus amplus (PERAP): единственный найденный представитель рода - Perognathus longimembris, но последовательность неаннотирована. Другой единственный вид из того же семейства - Chaetodipus penicillatus (Пустынная карманная мышь) - CHAPN. 4. Cratogeomys fumosus (CRAFU): взят вид из самого родственного рода[3] - Geomys pinetis (Юго-восточный карманный суслик) - GEOPI. 5. Artibeus obscurus (ARTOB) – взят самый родственный вид[4] того же рода - Artibeus jamaicensis (Ямайский листонос) - ARTJA.
Чтобы вырезать последовательность 12S rRNA в отдельный файл была использована команда (пример для Mus musculus):
seqret embl:J01420.1[70:1024] MOUSE_12S_rRNA.fastaКоманда чтобы переименовать последовательности мнемониками организмов:
descseq -seq MOUSE_12S_rRNA.fasta -out MOUSE_12S_rRNA.fasta -name "MOUSE"Команда для выравнивания последовательностей:
muscle -align 12S_all.fasta -output 12s_ali.fastaКоманда для реконструкции дерева программой IQ-Tree:
iqtree -s 12s_ali.rform.phy
Сравнение деревьев
Еще раз приведу список видов, которые были заменены на родственные при реконструкции дерева по последовательностям 12S rRNA: MARBB -> MARSI DIPAG -> DIPME PERAP -> CHAPN CRAFU -> GEOPI ARTOB -> ARTJA Чтобы можно было сравнить деревья, каждую пару придется условно считать за один организм. 1) Отличия от дерева, построенного программой IQ-Tree по белковой последовательности цитохрома B (см. Рис. 1):
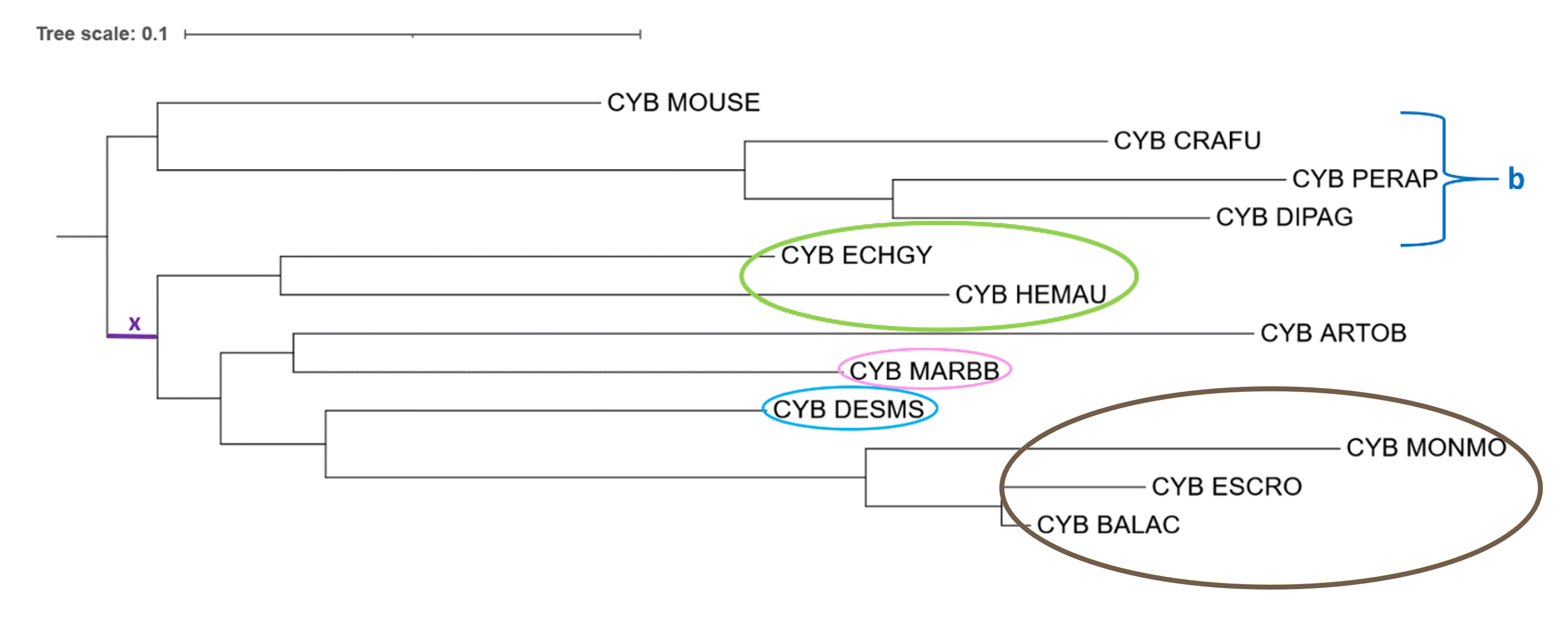
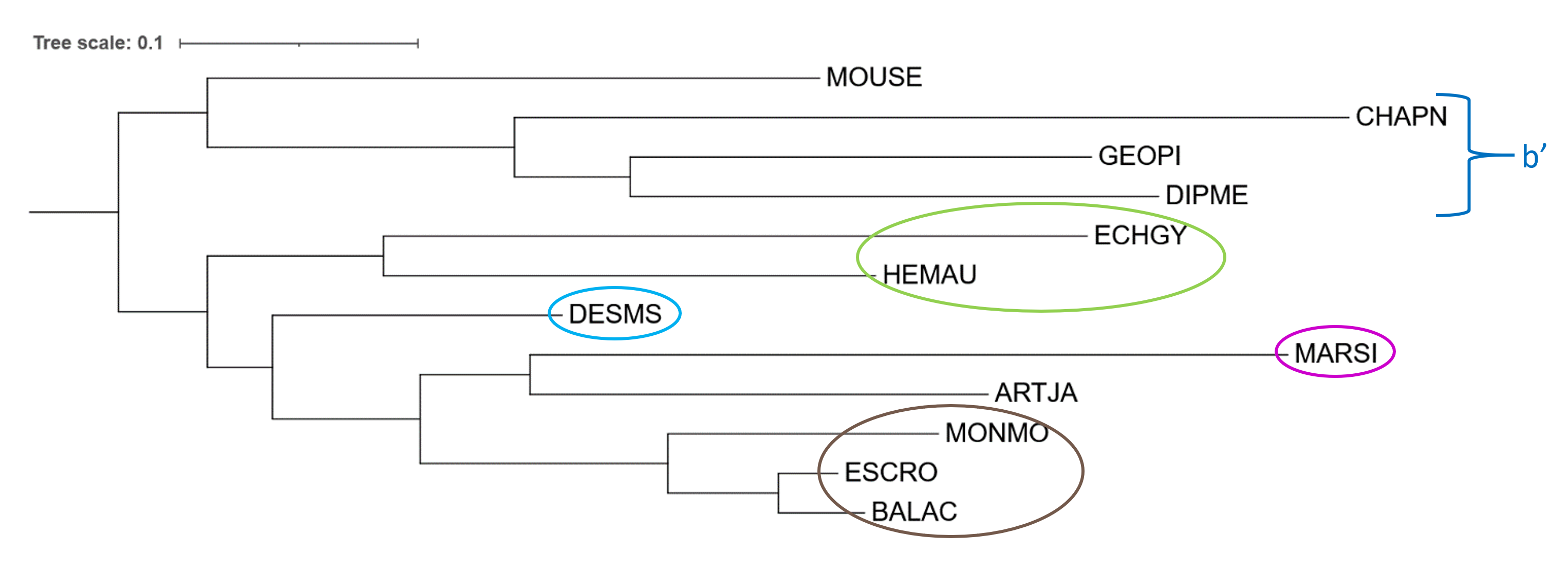
1. Сравним клады b и b’. Они состоят из одинаковых видов, но в случае cyt B PERAP и DIPAG, составляют кладу, сестринскую с CRAFU, а в случае 12S рРНК DIPAG (DIPME) и CRAFU(GEOPI), составляют кладу, сестринскую с PERAP (CHAPN). 2. На обоих деревьях есть клада, включающая MARBB (MARSI) и ARTOB (ARTJA), но ее расположение отличается: в случае cyt B она является сестринской к кладе (Cetacea (выделена коричневым)+DESMS), а в случае 12S рРНК она является сестринской к кладе Cetacea. 3. Эволюционные расстояния. Сходства: - одинаковый видовой состав клад b и b’. - MOUSE является сестринским видом к кладе b/b’. - к ветви x (отмечена фиолетовым на Рис. 1) принадлежат одинаковые виды. - есть одинаковые клады: выделенные зеленым (HEMAU+ECHGY) и коричневым (MONMO, BALAC, ESCRO), а также описанная выше клада MARBB (MARSI) + ARTOB (ARTJA).
2) Отличия от дерева, основанного на NCBI Taxonomy (см. Рис. 3):
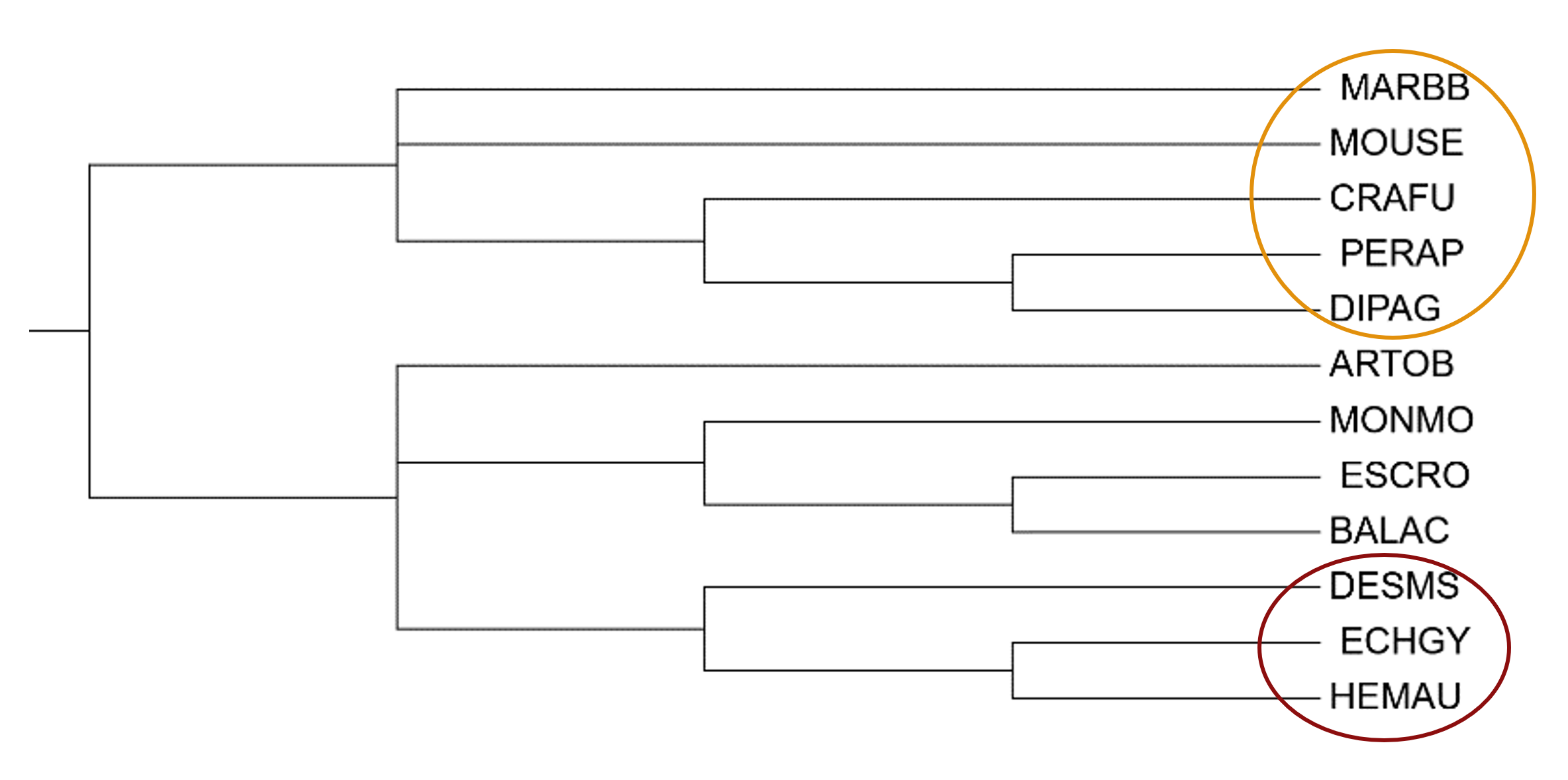
1. Можно, как в практикуме 2, рассмотреть 2 самые крупные клады на исходном дереве NCBI Taxonomy: выделенная оранжевым и состоящая из 7ми оставшихся видов. В полученном дереве только MARBB (MARSI) находится не в своей кладе. 2. Ошибочно не реконструирована ветвь, включающая Eulipotyphla (Насекомоядных) – выделена темно-красным. Есть клада из HEMAU и ECHGY (выделена зеленым), но DESMS является сестринским видом не к этой кладе, а к кладе из видов MONMO, BALAC, ESCRO, MARBB (MARSI), ARTOB (ARTJA). 3. Ошибочно не реконструирована ветвь, включающая Rodentia (Грызунов) - выделена оранжевым. В кладе b’ верный видовой состав, но PERAP (CHAPN) и DIPAG (DIPME) должны составлять кладу, сестринскую с CRAFU (GEOPI); то есть описаннвя выше клада b реконструирована верно. Расположение MOUSE в кладе Грызунов верное. MARBB (MARSI) находится в другой части дерева – составляет кладу с ARTOB (ARTJA), которая в свою очередь является сестринской с кладой Cetacea. Таким образом, расположение вида MARBB (MARSI) сильнее всего отличается от верного (на дереве NCBI Taxonomy).
3) Отличия от деревьев, построенных программой FastME по белковой последовательности цитохрома B (см. Рис. 4,5):
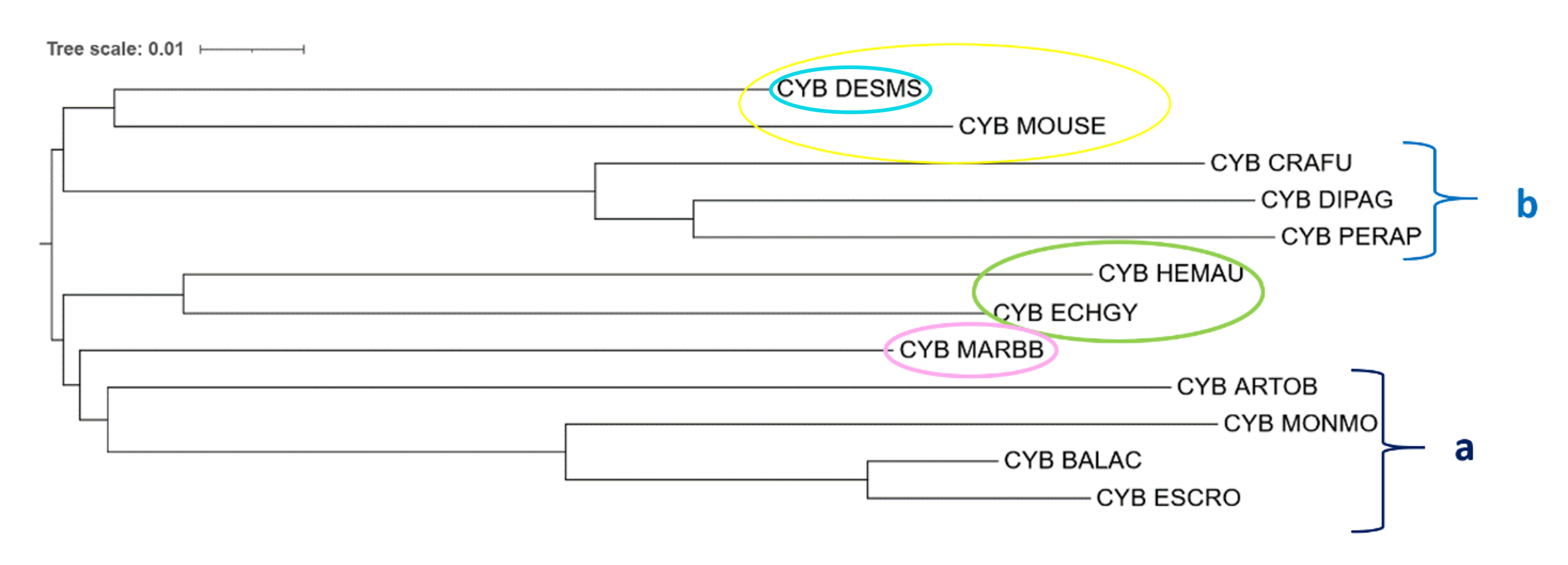
1. Описанные выше различия между кладами b и b’. 2. DESMS не сестринский вид к кладе b + MOUSE, как в случае модели MtREV или к MOUSE, как в случае модели p-distance; DESMS является сестринским видом к кладе из видов MONMO, BALAC, ESCRO, MARBB (MARSI), ARTOB (ARTJA), то есть находится совсем в другой части дерева. 3. MARBB (MARSI) не является сестринским видом к кладе HEMAU + ECHGY, как в случае модели MtREV или к кладе a, как в случае модели p-distance; MARBB (MARSI) образует кладу с ARTOB (ARTJA). 4. Эволюционные расстояния. Сходства: - одинаковый видовой состав клад b и b’. - есть одинаковые клады: выделенные зеленым (HEMAU+ECHGY) и коричневым (MONMO, BALAC, ESCRO).
Итог: Деревья, реконструированные с помощью программы IQ-Tree (по белковой последовательности цитохрома B и нуклеотидной последовательности 12s рРНК) больше похожи между собой, чем деревья, реконструированные по белковой последовательности цитохрома B программами FastMe и IQ-Tree. То есть интересно, что выбор программы больше повлиял на топологию дерева, чем выбор последовательности, по которой происходила реконструкция.
2. Укоренение во внешнюю группу.
Самый нижний таксон из тех, к которым принадлежат выбранные ранее животные - Boreoeutheria. В качестве организма, который может служить внешней группой был выбран Trichechus manatus (Обыкновенный ламантин) - TRIMA. Его таксономия (NCBI Taxonomy): Metazoa; Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Mammalia; Theria; Eutheria; Afrotheria; Sirenia; Trichechidae; Trichechus. Он вместе с ранее выбранными видами относится к Eutheria, но следующий нижележащий таксон для него - Afrotheria, а не Boreoeutheria. Для построения совместного дерева была использована последовательность цитохрома B, так как реконструированные по этой последовательности деревья были ближе по топологии к дереву NCBI Taxonomy. Для реконструкции дерева была использована программа IQ-Tree.
Результат:
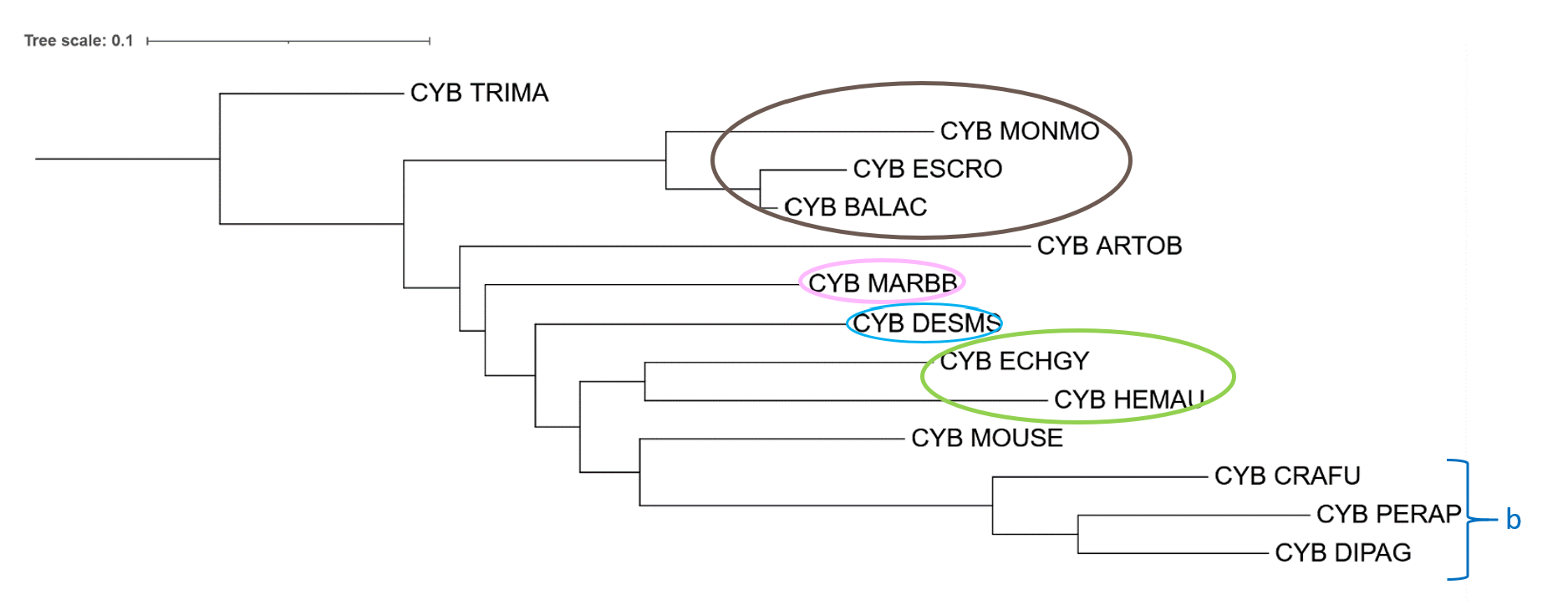

Сходство с деревом, реконструированном с помощью программы IQ-Tree без использования внешней группы (рис.1): есть клады, выделенные зеленым (HEMAU + ECHGY), коричневым ((MONMO, BALAC, ESCRO) и клада (b (CRAFU, DIPAG, PERAP) + MOUSE).
Сравнение с деревом, основанном на NCBI Taxonomy: Некоторые клады были реконструированы верно, хотя в целом топология дерева сильно отличается по расположению крупных клад: 1) Клада Cetacea - MONMO, BALAC, ESCRO (выделена коричневым) расположена совсем в другой части дерева (самая корневая), а не является сестринской к ARTOB и Eulipotyphla (Насекомоядных) – выделена темно-красным. 2) Ошибочно не реконструирована ветвь, включающая Eulipotyphla. Есть клада из HEMAU и ECHGY (выделена зеленым), но она неверно оказалась сестринской к кладе (b + MOUSE); DESMS является сестринским видом не к этой кладе, а к кладе (HEMAU + ECHGY, MOUSE, b). 3) Ошибочно не реконструирована ветвь, включающая Rodentia (Грызунов) - выделена оранжевым. Клада (b + MOUSE) верно реконструирована по видовому составу, хотя находится в другой части дерева, MARBB расположен неверно - является сестринским к кладе (DESMS, HEMAU + ECHGY, MOUSE + b). 4) Неверно расположен ARTOB (не является сестринским к кладам Eulipotyphla и Cetacea). Однако, можно заметить, что отсутствуют многие неверные клады (в сравнении с рис. 1): ARTOB + MARBB, DESMS + Cetacea (MONMO, BALAC, ESCRO), хотя все равно неверно расположены DESMS, ARTOB, MARBB (сильнее всего удален от своей клады), что описано выше.
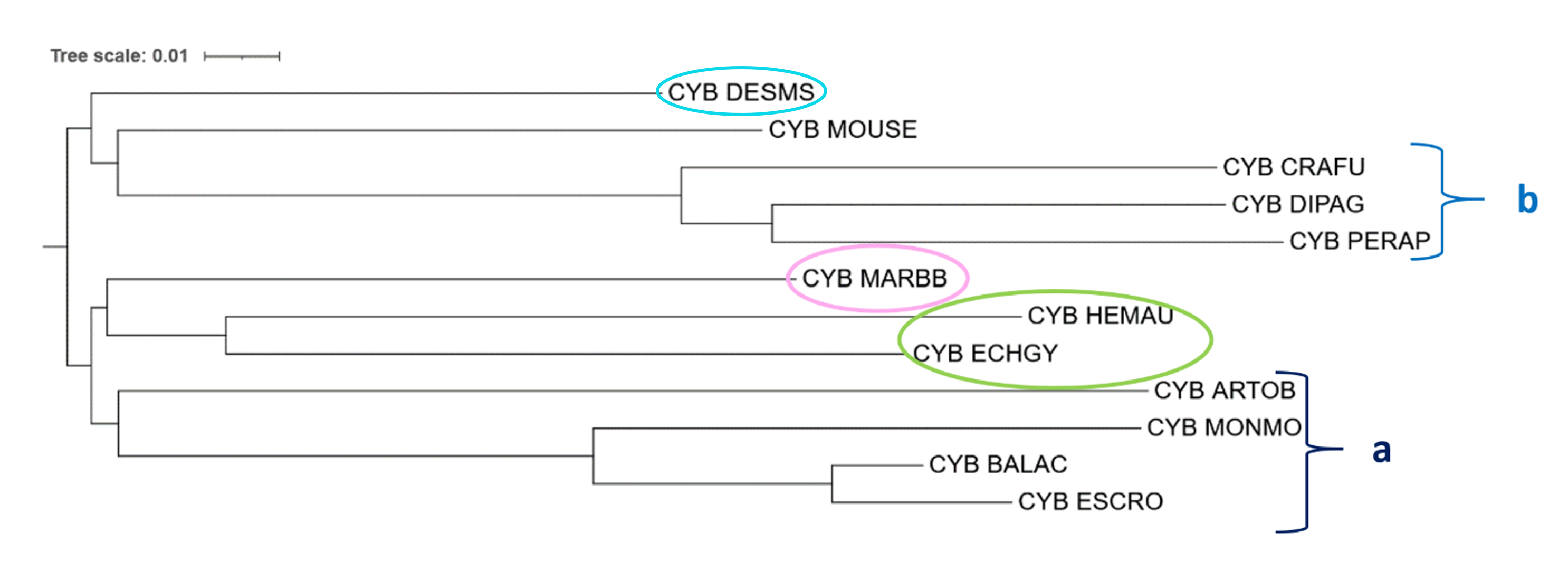
3. Бутстреп.
Была проведена реконструкция дерева по белковым последовательностям цитохрома B с помощью программы FastME (модель MtREV) с использованием 100 реплик бутстрепа. Команда:
fastme -pm -b 100 -i cyb.phy -o cyb-p.tre
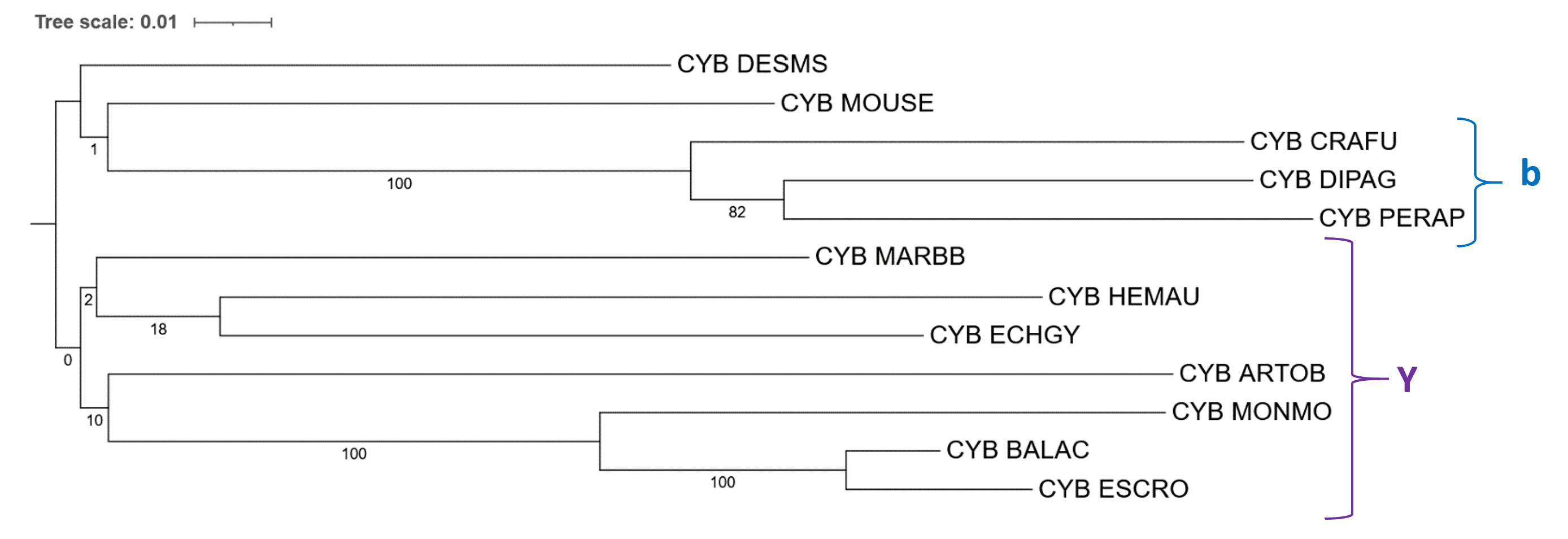

Сравнение полученного дерева по топологии с деревом, основанном на NCBI Taxonomy было приведено в практикуме 2. Описание дерева: - Поддержку 100 имеют 3 ветви, и они действительно верно реконструированы (это клады (BALAC, ESCRO), (MONMO, BALAC, ESCRO) и (CRAFU, DIPAG, PERAP)). - Ветвь, образующая кладу DIPAG + PERAP имеет поддержку 82, хотя она реконструирована верно. Из всех построенных деревьев она не была реконструирована только при использовании последовательности 12S рРНК. Рассмотрим ветви с достаточно низкой поддержкой: - Поддержку 0 имеет ветвь, образующая кладу Y; а поддержку 2 – образующая кладу (MARBB, HEMAU + ECHGY). Это можно объяснить тем, что MARBB находится совсем не в той части дерева, где должен быть по NCBI Taxonomy. - Поддержку 1 имеет ветвь, образующая кладу (b + MOUSE), что необычно, так как MOUSE и клада b действительно являются сестринскими по NCBI Taxonomy. Возможно, это связано с тем, что MARBB тоже должен быть в составе клады Грызунов (b + MOUSE + MARBB), выделенной оранжевым, а он находится внутри клады Y. - Ветвь, образующая кладу Cetacea (MONMO, BALAC, ESCRO) + ARTOB имеет поддержку 10, хотя ARTOB и Cetacea являются сестринскими. Возможно, такое маленькое число поддержки обусловлено тем, что к этой ветви должны еще принадлежать Насекомоядные (HEMAU, ECHGY, DESMS), но здесь HEMAU + ECHGY образуют кладу с MARBB, что описано выше. - Самое неожиданное, что ветвь, образующая кладу HEMAU + ECHGY имеет поддержку 18, хотя она является верной в соответствии с NCBI Taxonomy и была реконструирована на всех построенных деревьях не зависимо от программы или последовательности.
Источники:
1. Brandler O. V., Lyapunova E. A. Molecular phylogenies of the genus Marmota (Rodentia Sciuridae): comparative analysis //Ethology Ecology & Evolution. – 2009. – Т. 21. – №. 3-4. – С. 289-298. 2. Harder A. M. et al. High-quality reference genome for an arid-adapted mammal, the banner-tailed kangaroo rat (Dipodomys spectabilis) //Genome Biology and Evolution. – 2022. – Т. 14. – №. 1. – С. evac005. 3. Álvarez-Castañeda S. T., Segura-Trujillo C. A. Genus-level review of pocket gophers in the family Geomyidae //THERYA. – 2025. – Т. 16. – №. 1. – С. 41-75. 4. Larsen P. A. et al. Phylogenetics and phylogeography of the Artibeus jamaicensis complex based on cytochrome-b DNA sequences //Journal of Mammalogy. – 2007. – Т. 88. – №. 3. – С. 712-727.