Практические аспекты реконструкции филогении. Ортологи и паралоги.
1. Составление списка гомологичных белков, включающих паралоги.
Выбранные бактерии из отдела Pseudomonadota: - Burkholderia mallei (BURMA) - Neisseria meningitidis (NEIMA) - Pasteurella multocida (PASMU) - Polynucleobacter asymbioticus (POLAQ) - Roseobacter denitrificans (ROSDO) - Thiobacillus denitrificans (THIDA) - Yersinia pestis (YERPE) Сначала был проведен поиск в протеомах достоверных гомологов белка CLPX_ECOLI (Файл с аминокислотной последовательностью белка – clpx.fasta): Команда для создания файла с протеомами всех выбранных бактерий:
cat BURMA.fasta NEIMA.fasta PASMU.fasta POLAQ.fasta ROSDO.fasta THIDA.fasta YERPE.fasta > proteomes.fastaКоманда для индексации полученного файла:
makeblastdb -in proteomes.fasta -dbtype prot -out prot_dbКоманда для поиска гомологов белка CLPX_ECOLI в полученной локальной базе данных:
blastp -query clpx.fasta -db prot_db -evalue 0.001 -out blastp_out.txt
Получилось 27 находок.
Cписок находок из выдачи BLAST: sp|Q8ZC66|CLPX_YERPE ATP-dependent Clp protease ATP-binding subun... 805 0.0 sp|Q3SI99|CLPX_THIDA ATP-dependent Clp protease ATP-binding subun... 642 0.0 sp|Q62JK8|CLPX_BURMA ATP-dependent Clp protease ATP-binding subun... 617 0.0 sp|A4SXD7|CLPX_POLAQ ATP-dependent Clp protease ATP-binding subun... 613 0.0 sp|P57981|CLPX_PASMU ATP-dependent Clp protease ATP-binding subun... 612 0.0 sp|Q165G0|CLPX_ROSDO ATP-dependent Clp protease ATP-binding subun... 582 0.0 sp|Q9JTX8|CLPX_NEIMA ATP-dependent Clp protease ATP-binding subun... 557 0.0 sp|Q8ZJJ5|HSLU_YERPE ATP-dependent protease ATPase subunit HslU O... 95.1 2e-21 sp|Q16CY2|HSLU_ROSDO ATP-dependent protease ATPase subunit HslU O... 93.6 6e-21 sp|P57968|HSLU_PASMU ATP-dependent protease ATPase subunit HslU O... 93.2 8e-21 tr|Q3SFW1|Q3SFW1_THIDA ATP-dependent protease ATPase subunit HslU... 86.7 1e-18 sp|Q62F00|HSLU_BURMA ATP-dependent protease ATPase subunit HslU O... 82.4 3e-17 tr|A0A5P8YGZ0|A0A5P8YGZ0_YERPE ATP-dependent Clp protease ATP-bin... 51.2 4e-07 tr|A0A5P8YB42|A0A5P8YB42_YERPE ATP-dependent protease OS=Yersinia... 46.2 2e-05 tr|A0A5P8YCE6|A0A5P8YCE6_YERPE ATP-dependent zinc metalloprotease... 45.8 2e-05 sp|Q168A2|RUVB_ROSDO Holliday junction branch migration complex s... 45.4 2e-05 tr|A0A0H2WJ72|A0A0H2WJ72_BURMA ATP-dependent zinc metalloprotease... 43.9 7e-05 tr|Q3SJR4|Q3SJR4_THIDA ATP-dependent zinc metalloprotease FtsH OS... 43.5 1e-04 tr|A4SXL5|A4SXL5_POLAQ ATP-dependent zinc metalloprotease FtsH OS... 43.5 1e-04 tr|Q9CNJ2|Q9CNJ2_PASMU ATP-dependent zinc metalloprotease FtsH OS... 43.1 1e-04 sp|Q9JUB0|RUVB_NEIMA Holliday junction branch migration complex s... 42.7 1e-04 tr|Q3SJH1|Q3SJH1_THIDA ATP-dependent Clp protease, ATP-binding su... 42.7 2e-04 tr|Q9CKU5|Q9CKU5_PASMU ComM OS=Pasteurella multocida (strain Pm70... 41.6 4e-04 tr|Q16C81|Q16C81_ROSDO Chaperone protein ClpB OS=Roseobacter deni... 41.2 6e-04 tr|Q167Z2|Q167Z2_ROSDO ATP-dependent zinc metalloprotease FtsH OS... 41.2 6e-04 tr|A0A0U1RJ22|A0A0U1RJ22_NEIMA Replication-associated recombinati... 40.8 7e-04 tr|Q3SKL1|Q3SKL1_THIDA Chaperone protein ClpB OS=Thiobacillus den... 40.8 9e-04
2. Реконструкция и визуализация.
Далее Fasta файл с последовательностями находок blastp (названия – ID белков) был создан для выравнивания. Для выравнивания была использована программа Muscle. Команда для реконструкции дерева программой IQ-Tree:
iqtree -s ali.phyПолученный файл с формулой Newick. Дерево было укоренено в среднюю точку.
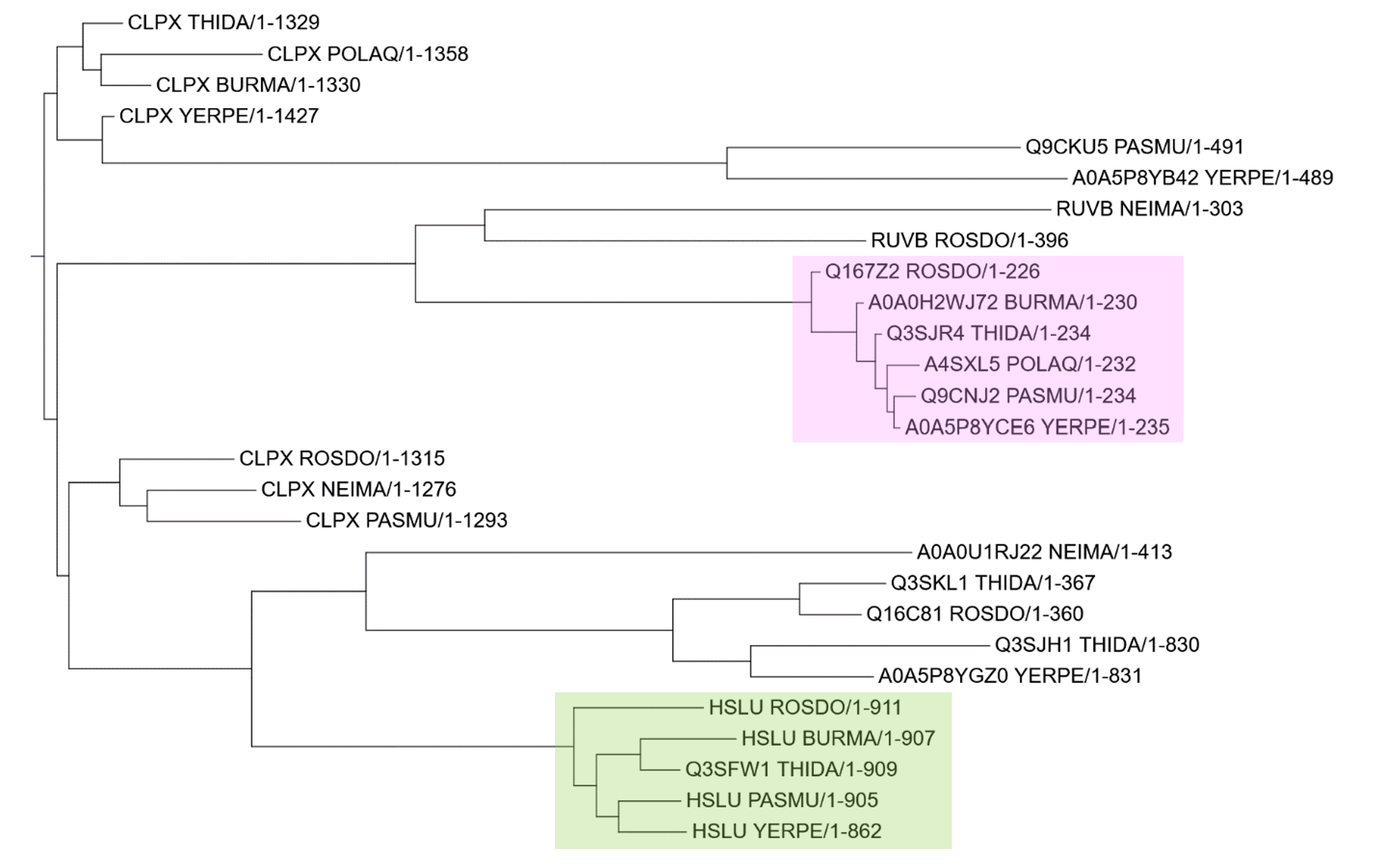
Три пары ортологов: HSLU_PASMU/1-905 и HSLU_YERPE/1-862 HSLU_BURMA/1-907 и Q3SFW1_THIDA/1-909 Q9CNJ2_PASMU/1-234 и A0A5P8YCE6_YERPE/1-235 Три пары паралогов: Q9CKU5_PASMU/1-491 и Q9CNJ2_PASMU/1-234 CLPX_NEIMA/1-1276 и RUVB_NEIMA/1-303 CLPX_ROSDO/1-1315 и HSLU_ROSDO/1-911
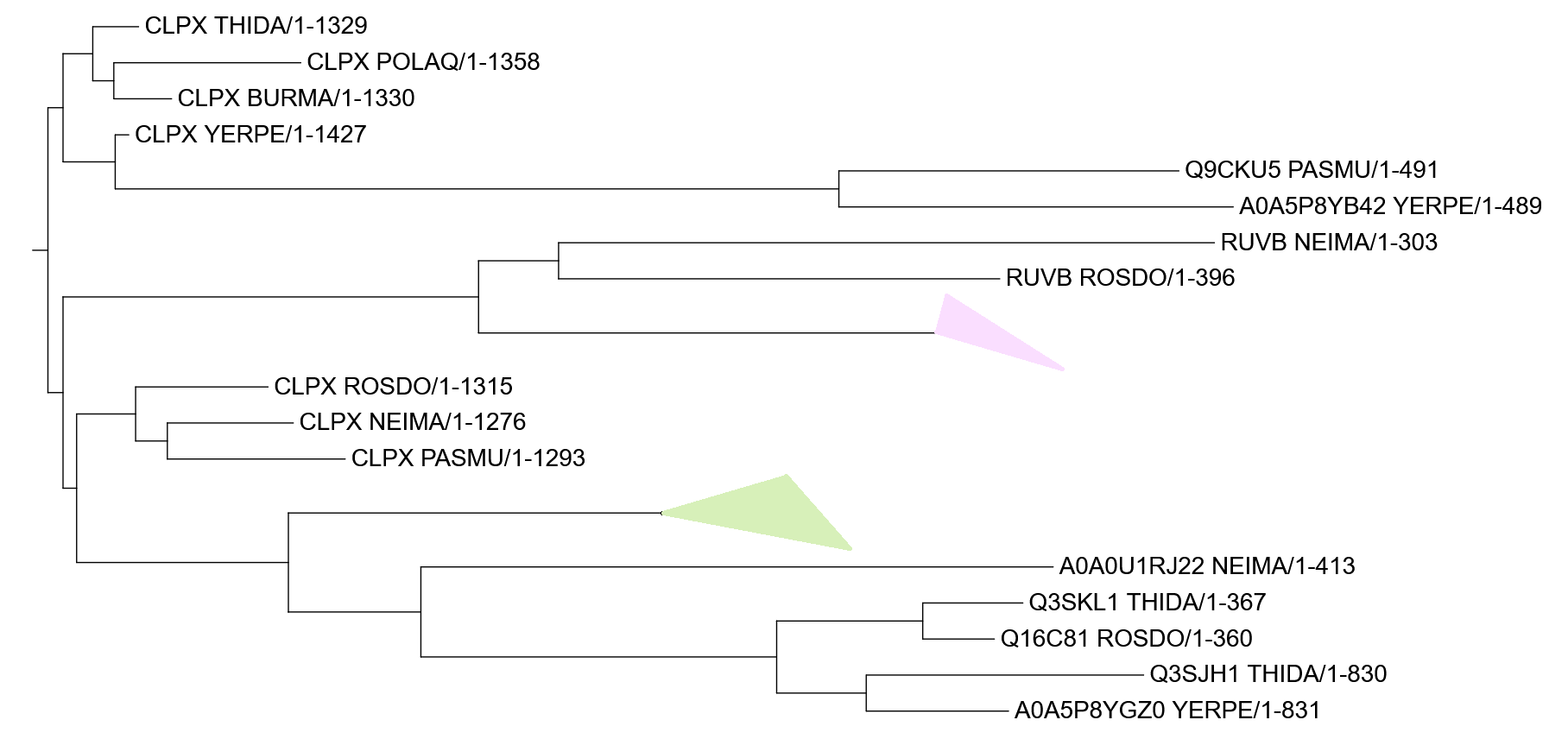
Рассмотрим ортологические группы. Группа, выделенная зеленым: Все белки представляют собой АТФ-зависимую субъединицу АТФ-зависимой протеазы HslU из разных бактерий. Эта субъединица обладает шапероновой активностью. Связывание АТФ и его последующий гидролиз с помощью HslU необходимы для разворачивания белковых субстратов, которые впоследствии гидролизуются с помощью hslV. Включает белки из 5 видов бактерий (все, кроме NEIMA и POLAQ). Реконструированная филогения белков соответствует филогении бактерий. Группа, выделенная розовым: Все белки представляют собой АТФ-зависимую цинковую металлопротеазу FtSH из разных бактерий. Этот фермент играет важную роль в контроле качества интегральных мембранных белков. Включает белки из 6 видов бактерий (все, кроме NEIMA). Филогения белков не совпадает с филогенией бактерий (см. Рис. 3, 4): не реконструирована клада POLAQ + BURMA; THIDA является сестринским видом не к этой кладе, а к кладе (POLAQ, PASMU + YERPE).