Базы данных GO, STRING, Reactome
Для анализа мне был предложен список из 21 гена человека: BCKDHB, ACAD8, BCKDHA, HIBADH, MCCC1, MCCC2, ACAT1, ACADSB, PPM1K, ECHS1, HSD17B10, BCKDK, ALDH6A1, AUH, DBT, IVD, DLD, SLC25A44, BCAT2, BCAT1, HIBCH. Я посмотрела какие белки кодируют эти гены – большинство оказались ферментами, работающими в митохондриях. Теперь нужно узнать, связаны ли эти белки между собой, в каких метаболических процессах участвуют.
1. STRING
Созданный граф (рис. 1) показывает, что белки биологически связаны; то есть, возможно, участвуют в одном метаболическом пути.
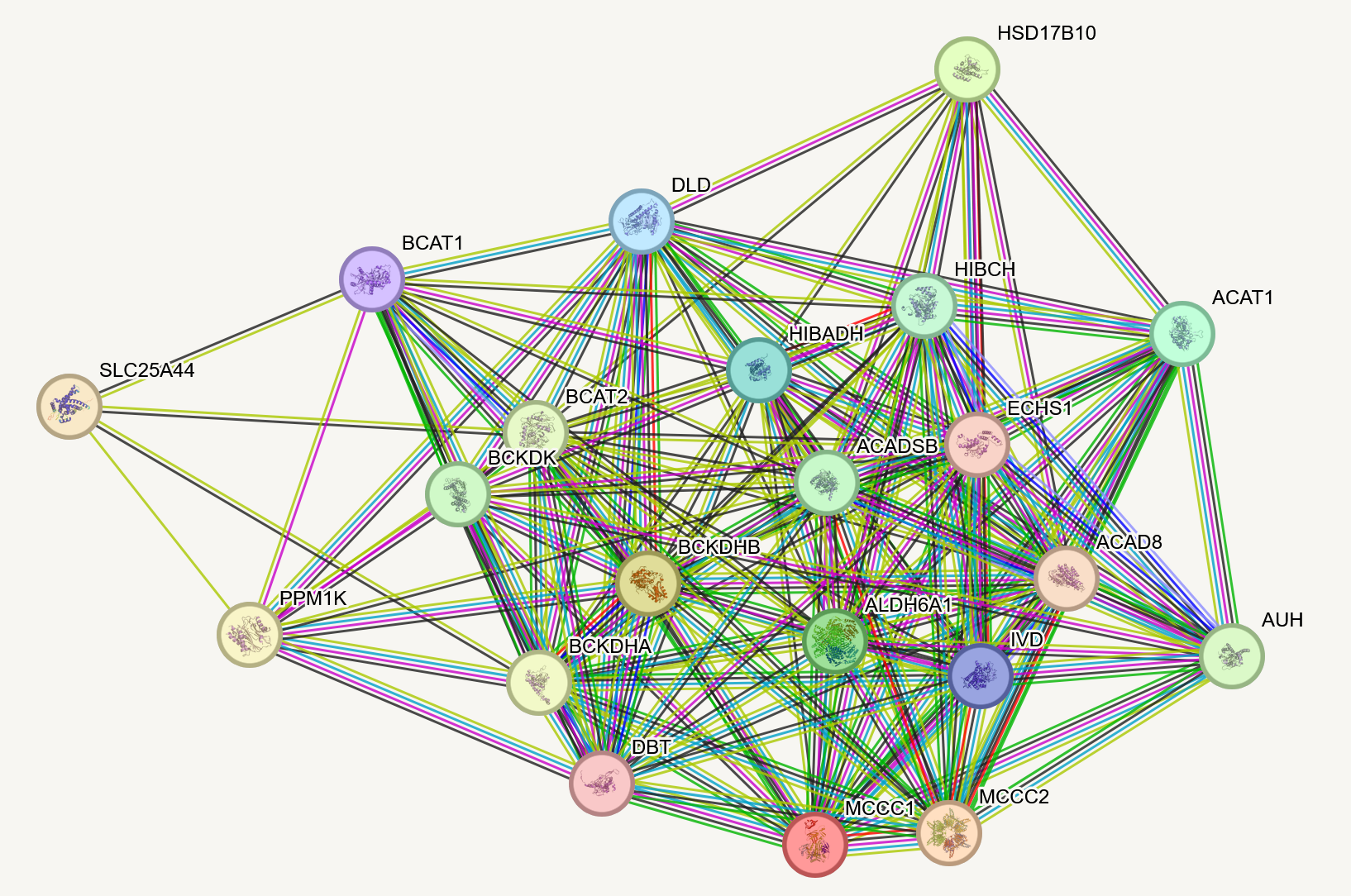
Белков достаточно много, поэтому была использована кластеризация (алгоритм k-means clustering). Если создавать 2 кластера (number of clusters: 2), белок SLC25A44 отделяется остальных, которые образуют кластер, что можно было предположить и из рисунка 1, так как этот белок имеет меньше всего взаимодействий. Если строить 3 кластера, они получались следующие: - Белок SLC25A44. - 12 белков, участвующих в катаболизме аминокислот с разветвленной цепью (это лейцин, изолейцин и валин) – 12 белков. - 8 белков, для которых помимо участия в катаболизме аминокислот с разветвленной цепью, указана также функция “деградация лейцина, изолейцина и валина”, что, по сути, одно и то же. Следовательно, не имеет смысла пытаться разделить белки на большее количество групп (кластеров). Какую же функцию выполняет белок, кодируемый геном SLC25A44? По данным из NCBI Gene и UniProt: SLC25A44 – белок переносчик растворенных веществ (Solute carrier family 25 member 44). Этот белок как раз транспортирует аминокислоты с разветвленной цепью и функционирует в митохондриях. Можно посмотреть подробнее в каких процессах участвует каждый белок с помощью окрашивания в Local Network Cluster (Рис. 2)
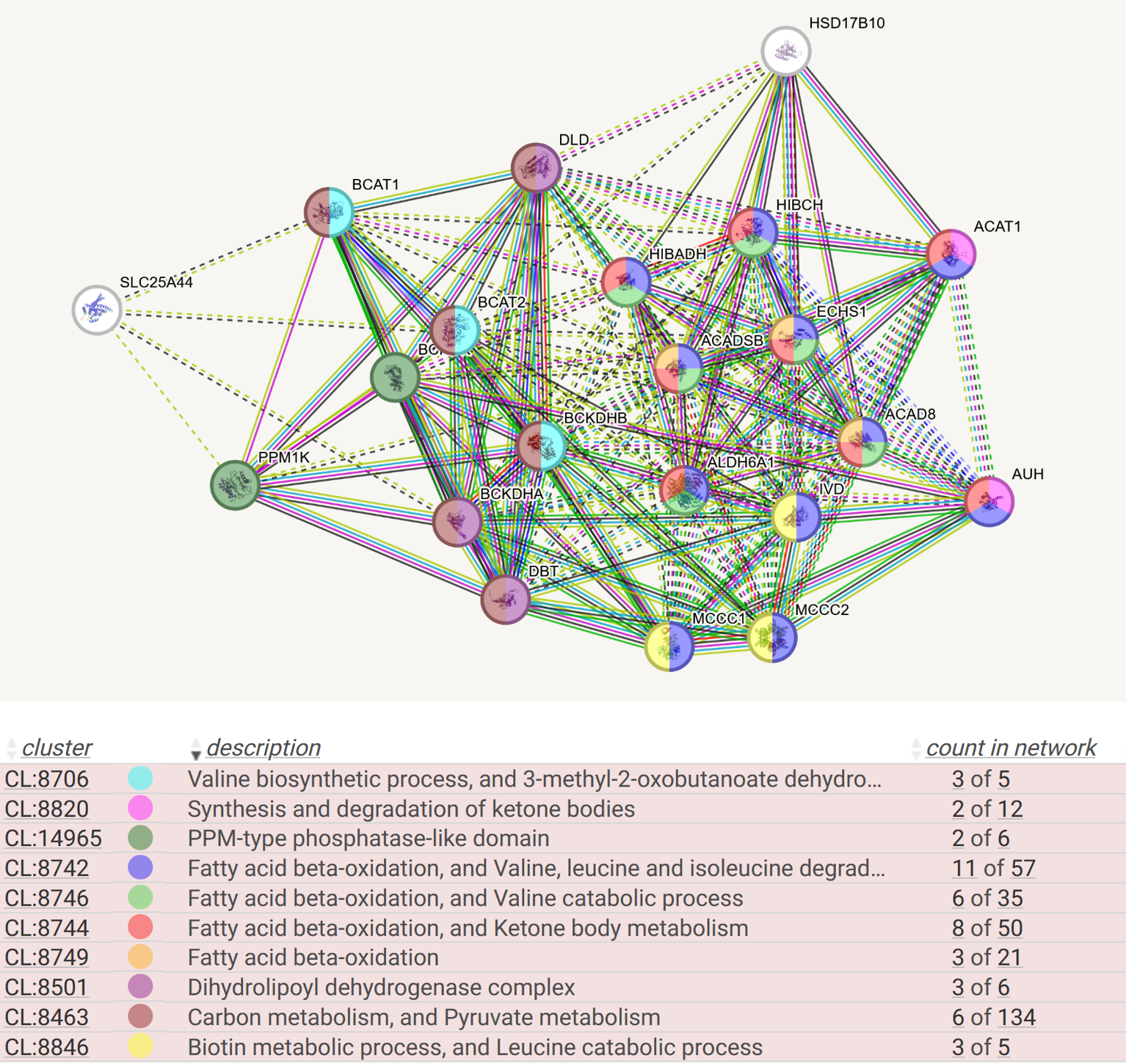
Из рисунка 2 видим, что помимо роли в метаболизме лейцина, изолейцина и валина, некоторые принимают участие и в других процессах тоже. Например: - синтез и разложение кетоновых тел (2 белка, розовый цвет на рисунке) - 2 белка (темно-зеленый) являются ферментами, которые вместе образуют функциональную пару, служащую метаболическим регуляторным узлом, который координирует аминокислоты с разветвленной цепью с метаболизмом глюкозы и липидов - бета-окисление жирных кислот (обозначены синим, светло- зеленым, красным, оранжевым, 8 белков) - метаболизм пирувата (коричневый, 6 белков) - метаболизм биотина (желтый, 3 белка) - дигидролипоилдегидрогеназа (фиолетовый, 3 белка) Заметно, что белок HSD17B10 не окрасился никаким цветом вместе с упомянутым SLC25A44, поэтому я решила посмотреть его функцию. Это 3-гидроксиацил-КоА дегидрогеназа 2-го типа, и не очень понятно, почему этот белок не окрасился, ведь он участвует и в метаболизме аминокислот с разветвленной цепью, и в бета-окислении жирных кислот.
Таким образом, с помощью STRING было выяснено, что все рассматриваемые белки помимо других разных процессов, участвуют в метаболизме аминокислот с разветвленной цепью: SLC25A44 переносит эти аминокислоты, а остальные 20 белков являются ферментами, катализирующими различные их превращения.
Использованные возможности STRING: Эта база данных содержала очень много нужной мне информации. Удобно, что можно объединять белки в кластеры, а также смотреть и на набор процессов, в которых участвует каждый белок, и на подробное описание белка при нажатии на узел графа.
2. Reactome
Я уже выяснила, в каком процессе участвуют исследуемые белки, и мне стало интересно посмотреть, как именно выглядит этот процесс. Reactome позволяет посмотреть схему нужного биологического пути. В выдаче, конечно, было указано, что белки участвуют в метаболизме аминокислот (Рис. 3), и там я нашла схему “ Branched-chain amino acid catabolism” – ссылка на схему в формате svg.
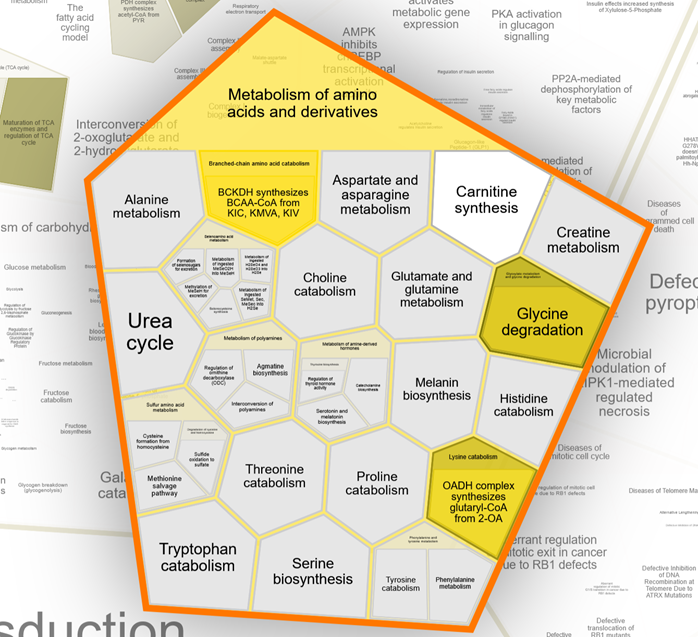
Некоторые белки из схемы:
1) На рисунке 4 виден белок SLC25A44, который транспортирует лейцин, изолейцин и валин из цитоплазмы клетки в матрикс митохондрии.
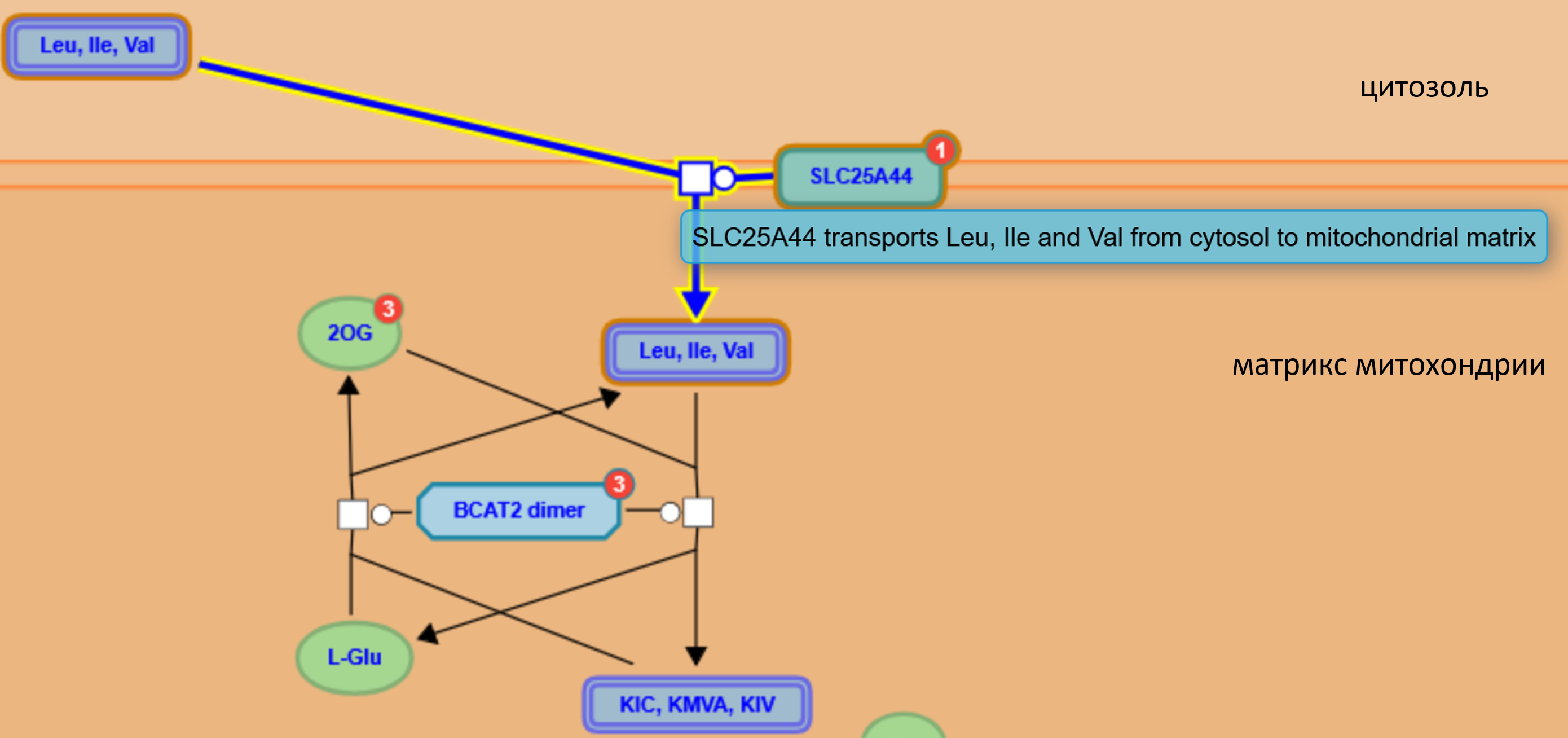
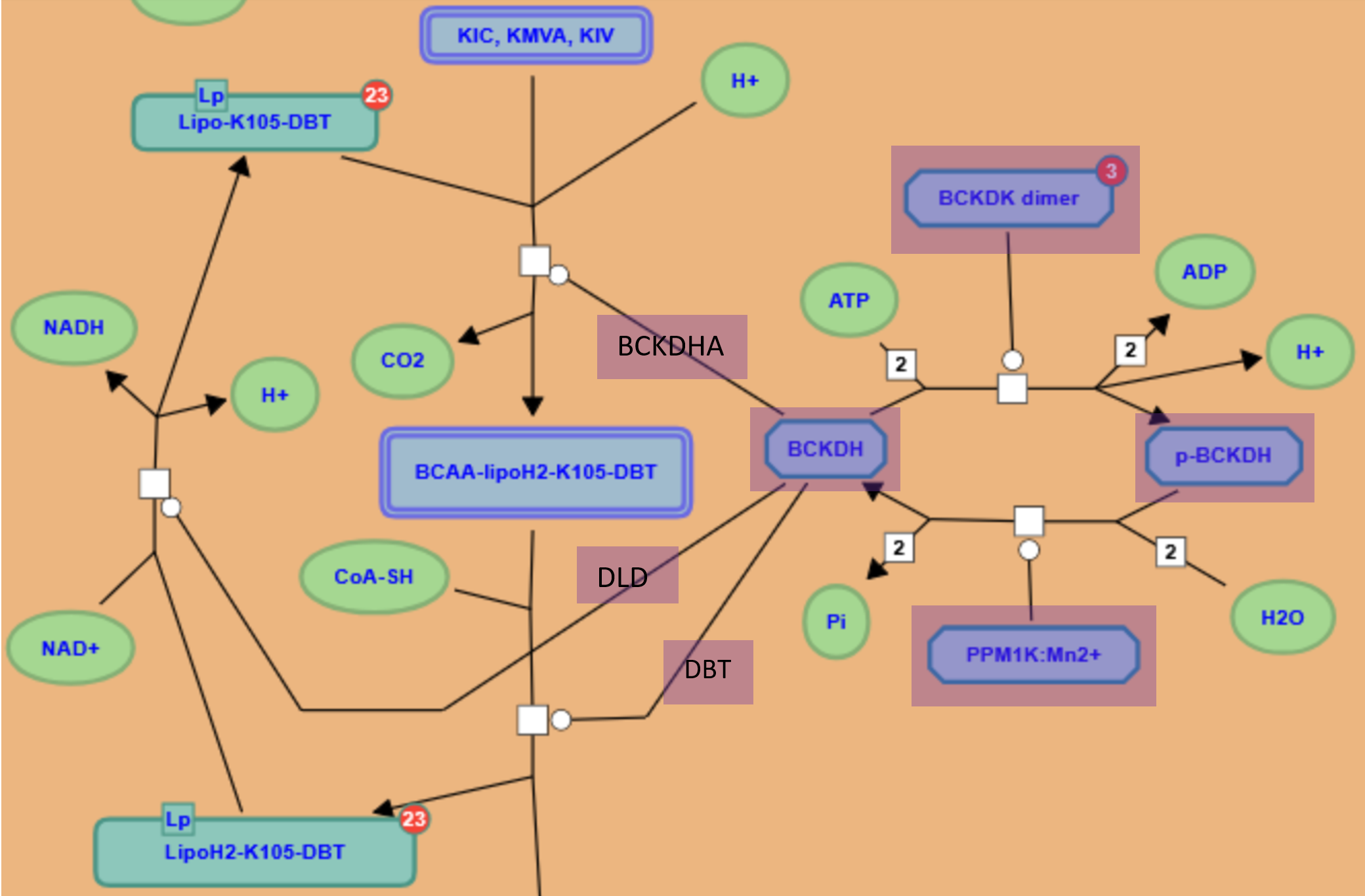
Использованные возможности Reactome: База данных помогла разобраться с функциями белков: со схемой проще понять, что означают различные термины, примеры которых приведены выше; можно для каждой стрелки посмотреть, что это за реакция. Также, удобно, что если на схеме не указаны расшифровки аббревиатур названий химических веществ, можно было в reactome content отдельно посмотреть про каждое вещество, его локализацию в клетке, участие в различных процессах и связь с заболеваниями.
3. Gene Ontology (GO)
Проверим, будут ли результаты анализа обогащения терминами GO соответствовать полученным ранее выводам. Для проведения анализа я использовала Enrichr, группа аннотаций GO Biological Process 2025. Использовался тест Фишера и поправка на множественное тестирование - False Discovery Rate. Количество терминов - 50, на рисунке 6 указаны 10 с самым низким p-value. ссылка на текстовую выдачу.

Получилось, что самое низкое значение p-value у для процесса катаболизма аминокислот с разветвленной цепью (что является частью их метаболизма, указанного как 2го по p-value процесса). Дальше идут процессы катаболизма лейцина и валина, что также относится к метаболизму аминокислот с разветвленной цепью. Были также найдены другие процессы, которые описывались в выдаче STRING (например, бета-окисление жирных кислот) – таких белков действительно было меньше, чем участвующих в метаболизме аминокислот с разветвленной цепью. Таким образом, результат анализа обогащения терминами GO соответствует данным, полученным с помощью STRING и Reactome.