Трансмембранные белки
1. Сравнение предсказаний трансмембранных участков в бета-листовом белке.
Я нашла тип Transmembrane, класс Beta-barrel transmembrane и выбрала семейство белков Gasdermin, белок Gasdermin B pore, структура показана на Рис. 1, 2. Название: Gasdermin B pore (газдермин B) Идентификатор PDB: 8et2 Идентификатор UniProt: GSDMB_HUMAN Организм, из которого был взят белок: Homo sapiens Локализация: клеточная мембрана
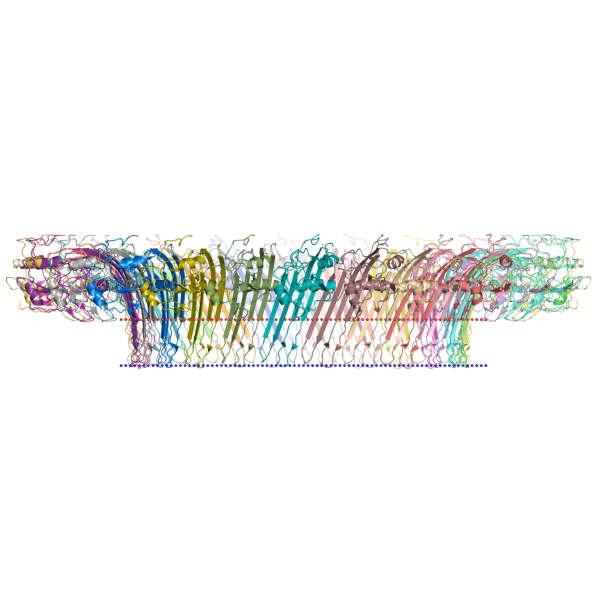
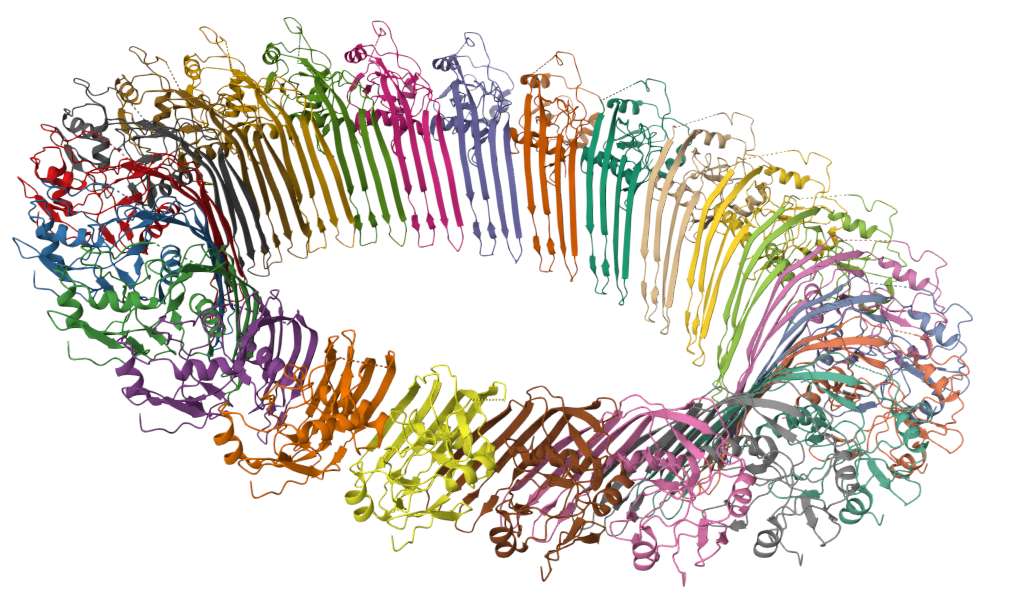
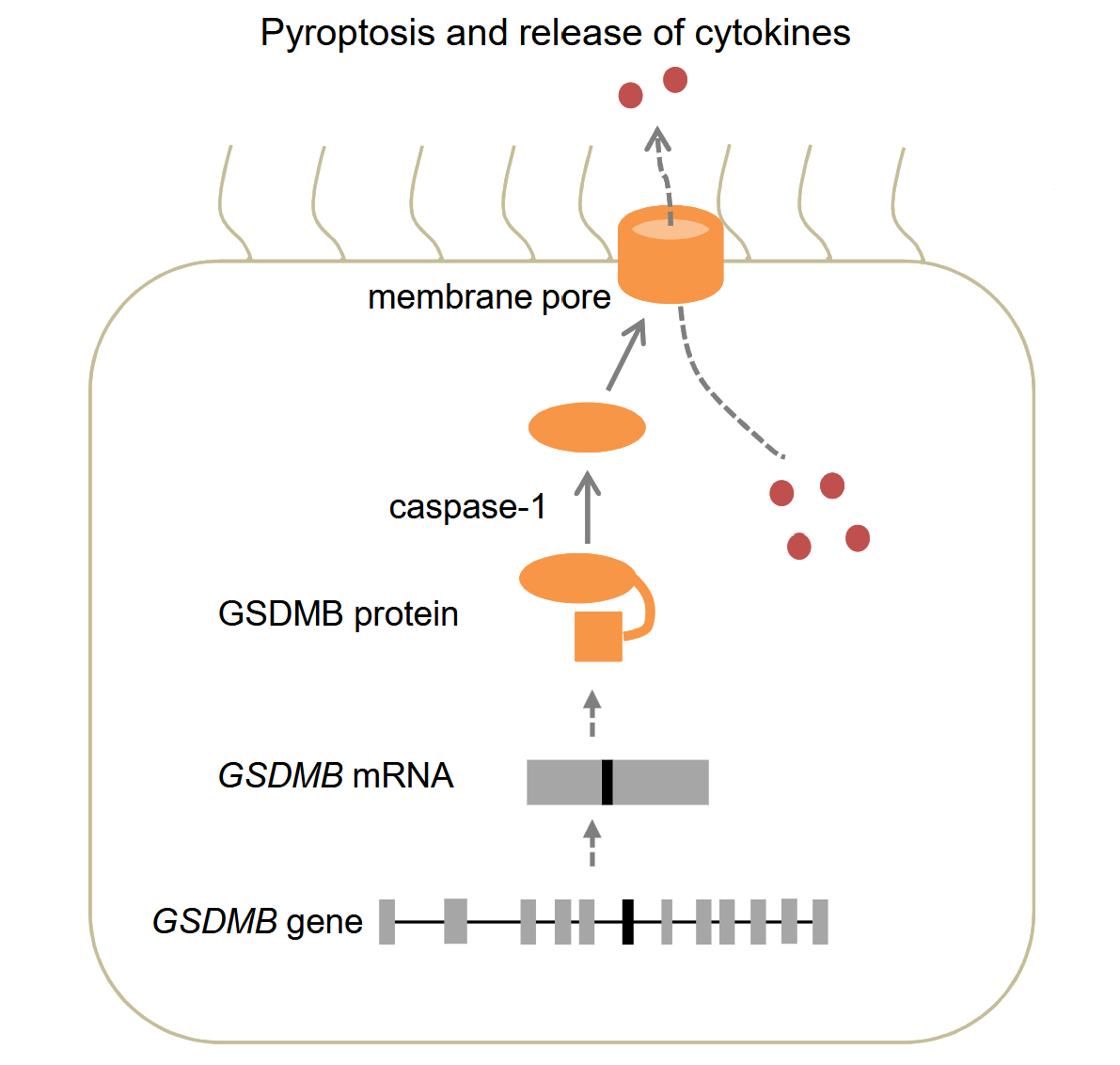
Этот белок экспрессируются у человека в клетках эпителия различных органов (по данным Human Protein Atlas – в основном в эпителии желудочно-кишечного тракта). Газдермин B может запускать механизм литической и провоспалительной гибели клеток, известный как пироптоз: при расщеплении высвобождающийся N-концевой домен связывается с мембранами и образует поры (в составе комплекса из 24 субъединиц), которые впоследствии приводят к лизису клеток (см. Рис.3). То есть после трансляции он локализован в цитоплазме, но здесь рассматривается именно его трансмембранная форма (N-концевой домен - Gasdermin B pore). Также действует как регулятор восстановления эпителиальных клеток: перемещается к плазматической мембране и способствует поддержанию и восстановлению эпителия, регулируя фосфорилирование различных белков.
Координаты трансмембранных участков белка газдермина B (24 субъединицы): A -TM segments: 1( 95- 98), 2( 105- 108), 3( 176- 185), 4( 186- 189) B - TM segments: 1( 95- 98), 2( 105- 109), 3( 176- 185), 4( 186- 189) C - TM segments: 1( 94- 98), 2( 105- 109), 3( 176- 185), 4( 186- 189) D - TM segments: 1( 94- 98), 2( 105- 109), 3( 176- 185), 4( 186- 189) E - TM segments: 1( 94- 98), 2( 105- 109), 3( 176- 185), 4( 186- 189) F - TM segments: 1( 95- 98), 2( 105- 108), 3( 176- 185), 4( 186- 189) G - TM segments: 1( 95- 98), 2( 105- 109), 3( 176- 185), 4( 186- 189) H - TM segments: 1( 95- 98), 2( 105- 108), 3( 176- 185), 4( 186- 189) I - TM segments: 1( 95- 98), 2( 105- 108), 3( 176- 185), 4( 186- 189) J - TM segments: 1( 95- 98), 2( 105- 109), 3( 176- 185), 4( 186- 189) K - TM segments: 1( 94- 98), 2( 105- 109), 3( 176- 185), 4( 186- 189) L - TM segments: 1( 95- 98), 2( 105- 108), 3( 176- 185), 4( 186- 189) M - TM segments: 1( 95- 98), 2( 105- 108), 3( 176- 185), 4( 186- 189) N - TM segments: 1( 95- 98), 2( 105- 108), 3( 176- 185), 4( 186- 189) O - TM segments: 1( 95- 98), 2( 105- 108), 3( 176- 185), 4( 186- 189) P - TM segments: 1( 95- 98), 2( 105- 109), 3( 176- 185), 4( 186- 189) Q - TM segments: 1( 95- 98), 2( 105- 108), 3( 176- 185), 4( 186- 189) R - TM segments: 1( 95- 98), 2( 105- 108), 3( 176- 185), 4( 186- 189) S - TM segments: 1( 95- 98), 2( 105- 108), 3( 176- 185), 4( 186- 189) T - TM segments: 1( 95- 98), 2( 105- 109), 3( 176- 185), 4( 186- 189) U - TM segments: 1( 94- 98), 2( 105- 109), 3( 176- 185), 4( 186- 189) V - TM segments: 1( 95- 98), 2( 105- 109), 3( 176- 185), 4( 186- 189) W - TM segments: 1( 95- 98), 2( 105- 108), 3( 176- 185), 4( 186- 189) X - TM segments: 1( 95- 98), 2( 105- 109), 3( 176- 185), 4( 186- 189)
Была получена последовательноcть белка из базы данных UniProt и запущен сервис DeepTMHMM. DeepTMHMM предсказал, что газдермин B локализован в цитоплазме клетки и имеет глобулярную форму (см. Рис.4). Этого можно было ожидать, так как он встраивается в мембрану только после модификаций каспазой (см. Рис. 3).
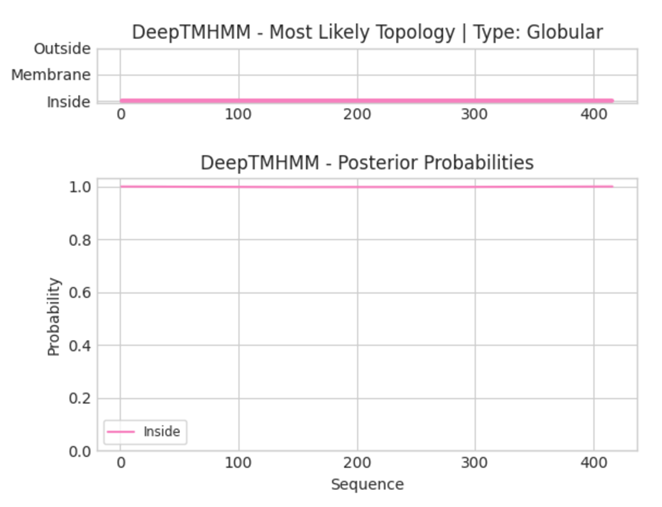
Я решила проверить, определится ли в DeepTMHMM N-концевой домен как трансмембранный, но в нескольких статьях, UniProt, Pfam у меня не получилось найти последовательность того фрагмента белка, который с большей вероятность является трансмембранным (отрезается каспазой). В статье [2] упоминается, что N-концевой домен встраивается в мембрану, и после внесения мутации L192D белок не проявлял пироптозную активность, следовательно, возможно, именно эту аминокислоту могут узнавать ферменты и в этом месте отрезать N-концевой домен, также в OPM последняя аминокислота в трансмембранных фрагментах -189я. Однако моделирование DeepTMHMM для короткого белка тоже показало только локализацию в цитоплазме. Получается, предсказание DeepTMHMM не совпало с данными OPM. Возможно, это также связано с большим количеством цепей у газдермина B и сложностью его структуры.
2. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке.
Выданный белок - 3n5k. Структура показана на рис.5. Название: Calcium ATPase, E2-Pi state, conformation 7 (Кальциевая АТФаза, состояние E2-Pi, конформация 7) Идентификатор PDB: 3n5k Идентификатор UniProt: AT2A1_RABIT Организм, из которого был взят белок: Oryctolagus cuniculus (Дикий кролик) Локализация: Саркоплазматический/эндоплазматический ретикулум
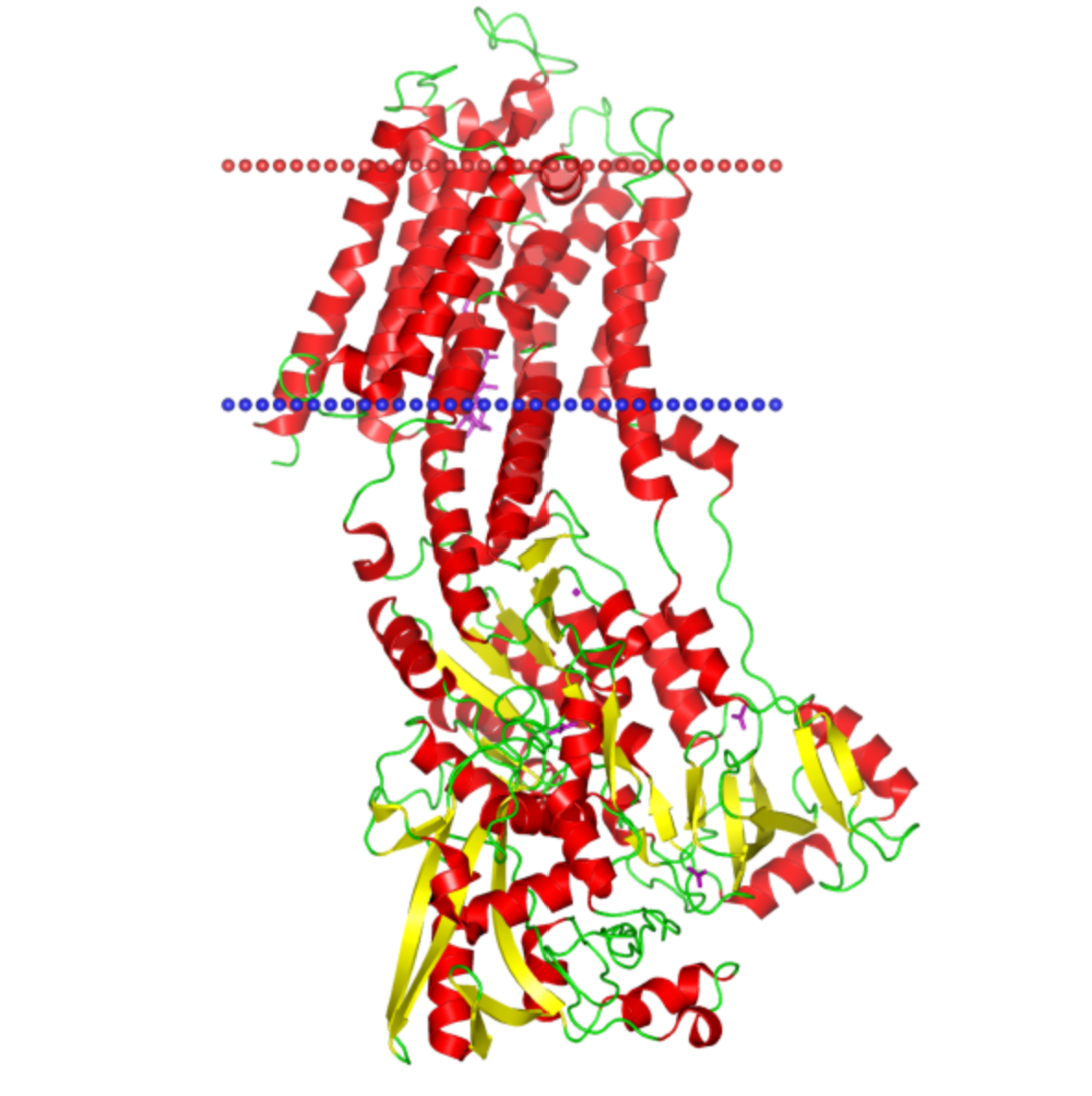
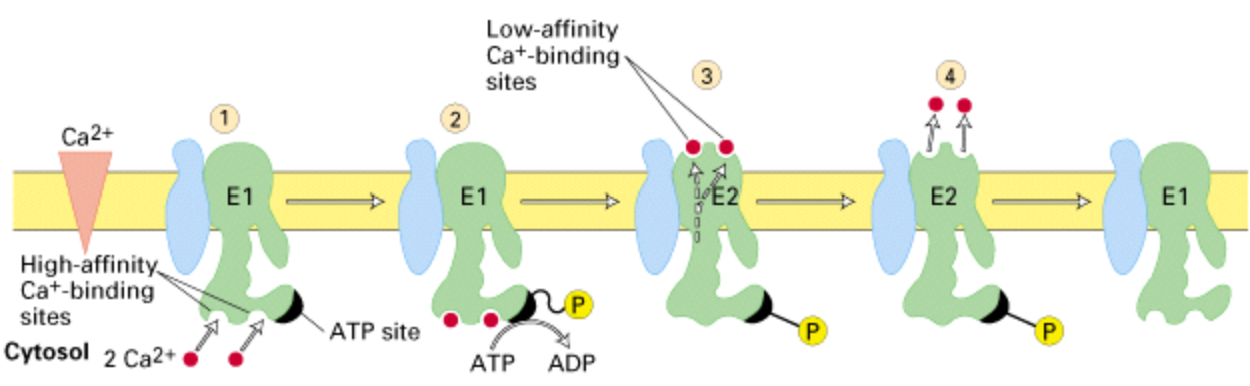
Этот белок является ключевым регулятором работы поперечно-полосатых мышц, действует как основная Са2+-АТФаза, ответственная за обратный захват цитозольного Са2+ саркоплазматическим ретикулумом. Катализирует гидролиз АТФ в сочетании с перемещением кальция из цитозоля в просвет саркоплазматического ретикулума. Са2+-АТФаза в своем цикле работы принимает различные конформации (См. Рис.6). Гидролиз АТФ и фосфорилирование белка вызывают конформационные изменения (переход из E1 в E2), которые обеспечивают перемещение Са2+ через мембрану. E2-Pi state, conformation 7 – конформация перед переходом к состоянию E1, здесь неорганический фосфат еще связан с ферментом, а определенные альфа-спирали закрывают канал, предотвращая обратный выход Са2+ в цитоплазму.
Координаты трансмембранных участков белка: TM segments: 1( 59- 78), 2( 85- 104), 3( 256- 280), 4( 291- 314), 5( 759- 781), 6( 789- 807), 7( 832- 853), 8( 896- 916), 9( 933- 950),10( 967- 987)
Была получена последовательноcть белка из базы данных UniProt и запущен сервис DeepTMHMM. Файл с результатом работы DeepTMHMM, также см. Рис.7.
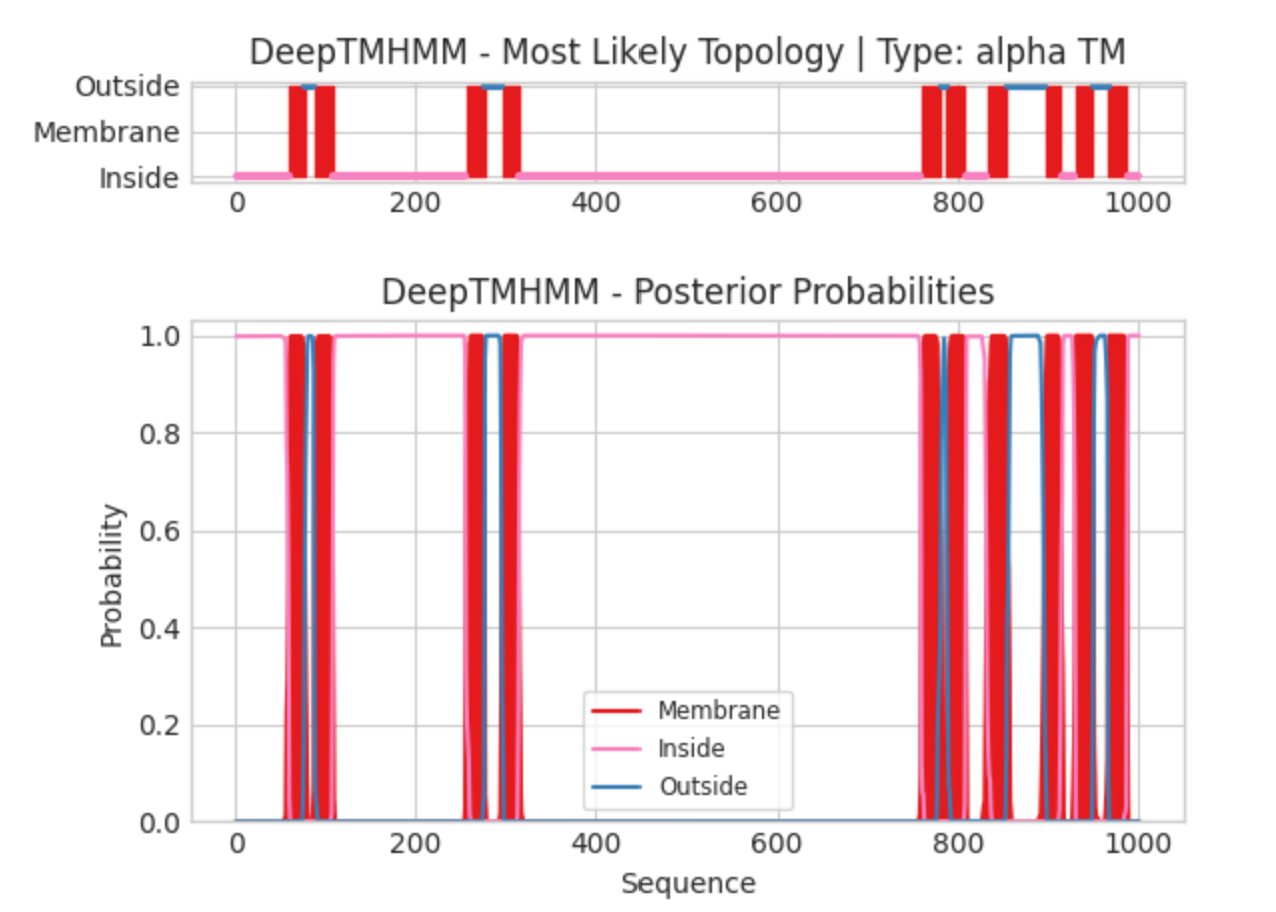
Координаты трансмембранных участков белка Calcium ATPase (предсказание DeepTMHMM): sp|P04191|AT2A1_RABIT TMhelix 60 77 sp|P04191|AT2A1_RABIT TMhelix 88 108 sp|P04191|AT2A1_RABIT TMhelix 256 276 sp|P04191|AT2A1_RABIT TMhelix 296 314 sp|P04191|AT2A1_RABIT TMhelix 760 781 sp|P04191|AT2A1_RABIT TMhelix 788 808 sp|P04191|AT2A1_RABIT TMhelix 833 854 sp|P04191|AT2A1_RABIT TMhelix 897 914 sp|P04191|AT2A1_RABIT TMhelix 931 950 sp|P04191|AT2A1_RABIT TMhelix 967 987
Сравнение с OPM: DeepTMHMM предсказал наличие 10 трансмембранных участков белка, что соответствует базе данных OPM. Их координаты слабо отличаются (на 1-4 позиции), последний участок полностью совпадает по координатам. Сильнее большинства отличаются координаты второго участка (85- 104 в OPM и 88-108 в DeepTMHMM). В PDB я посмотрела расположение этих аминокислот в структуре белка – вторая альфа-спираль начинается с 88 аминокислоты, однако по данным OPM аминокислота 85 находится внутри мембраны, но не входит в состав альфа спирали. Возможно, DeepTMHMM может лучше предсказать трансмембранный участок, если он в форме альфа-спирали, а не неупорядоченного фрагмента. Также, некоторые различия можно объяснить разной точностью определения структуры рентгеноструктурным анализом, результаты которого представлены в PDB (и OPM). Например, в еще одном относительно сильно отличающемся третьем фрагменте (256- 280 в OPM и 256-276 в DeepTMHMM) – точность предсказания 77-79 аминокислот - low coinfedence, что может быть связано с положением на границе мембраны и подвижностью белка.
Источники: 1. Panganiban R. A. et al. A functional splice variant associated with decreased asthma risk abolishes the ability of gasdermin B to induce epithelial cell pyroptosis //Journal of Allergy and Clinical Immunology. – 2018. – Т. 142. – №. 5. – С. 1469-1478. e2. 2. Ding J. et al. Pore-forming activity and structural autoinhibition of the gasdermin family //Nature. – 2016. – Т. 535. – №. 7610. – С. 111-116. 3. https://zoology.ubc.ca (сайт Department of Zoology at UBC)