
Чтение последовательностей по Сэнгеру.
1. Прочитать последовательность ДНК на основании данных, полученных из капиллярного секвенатора по Сэнгеру. Составить отчёт о проблемах при чтении хроматограмм.
Исходные файлы: Прямая последовательность , Обратная последовательность
Файлы были открыты в Chromas и для обратной была построена комплементарная последовательность (Edit>Reverse+Complement). Хроматограммы были выровнены с использованием поиска подслов (find). Для прямой цепи нечитаемый 5' конец нуклеотиды 1-52, 3'конец: 850-916, хотя проблемы начинаются с 774. Для обратной последовательности использовалась комплементарная, для 5' конца: 1-175, для 3'конца: 914-961. Качество хроматограмм довольно хорошее, но у прямой последовательности качество ухудшается к концу, а у обратной - к началу. Были удалены не читаемые 5'- и 3'- концы (Continuous edit). Затем последовательности были отредактированны, в результате были получены две fasta последовательности: Прямая последовательность fasta , Обратная последовательность fasta
С полученными последовательностями в программе Jalview было построено выравнивание, посмотреть проэкт по ссылке Выравнивание . В группы без окраски выделены проблемные нуклеотиды, которые в обратной последовательности вероятно образуют пятно.

Рис. 1 Выравнивание последовательностей (измененные нуклеотиды обозначены маленькими буквами).
Обоснование решений для 4х проблемных нуклеотидов.
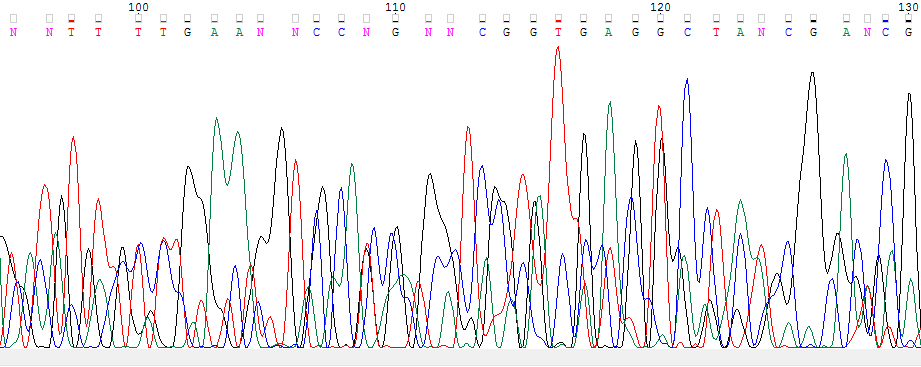
Ниже представлено два рисунка: на первом участок прямой последовательности с проблемным нуклеотидом, который выделен мышкой; на втором участок обратной последовательности, в которой выделен нуклеотид, соответствующий проблемному нуклеотиду из прямой последовательности. На первом рисунке видно,что сигнал проблемного нуклеотида N слаб и почти вровень с шумом, но на втором рисунке у соответствующего нуклеотида ясно виден пик, поэтому проблемный нуклеотид заменен на соответствующий из обратной последовательности.
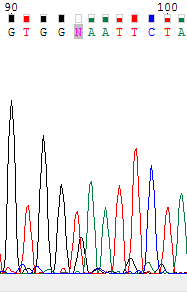
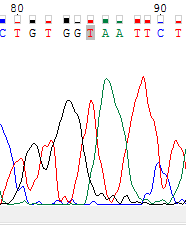
Ниже два рисунка: на первом участок прямой последовательности, на втором - обратной. На первом рисунке виден проблемный нуклеотид N, из-за высокого уровня шума. На втором рисунке в районе соответствующего нуклеотида виден размноженный пик у которого довольно низкий уровень шума. Поэтому проблемный нуклеотидбыл заменен на соответствующий из обратной последовательности.
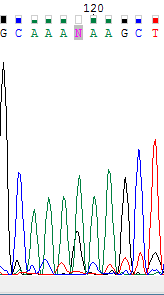
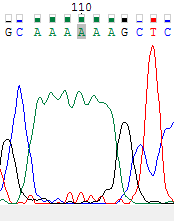
Ниже представлены два рисунка: на первом участок прямой последовательности, на втором обратной. На прямой последовательности у проблемного нуклеотида N, уровень шума довольно высок и не позволяет программе Chromas определить этот нуклеотид. А у обратной последовательности уровень шума у соответствующего нуклеотида мал, что позволяет мне заменить на него проблемный нуклеотидиз прямой последовательности.
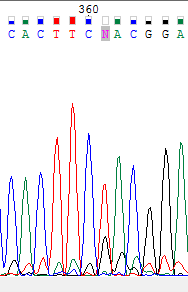
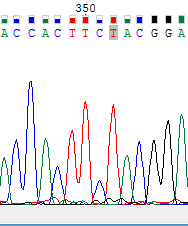
Ниже представлены два рисунка: на первом участок прямой последовательности, на втором обратной. Проблемный нуклеотид из прямой последовательности заменен на соответствующий нуклеотид из обратной по той же причине, что и на предыдущих рисунках, из-за того что у прямой цепи уровень шума высокий, а у обратной низкий.
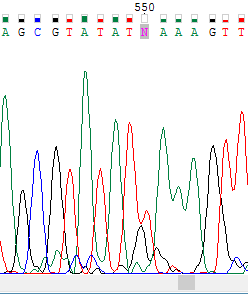
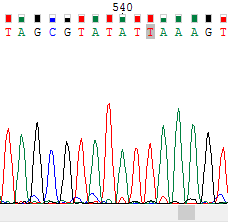
2. Приведите пример не читаемого фрагмента хроматограммы.
Плохо читаемая хроматограмма была взята из папки bad на диске P: kamp3_18SIII_R_F04_WSBS-Seq-1-08-15.ab1
Ниже представлен не читаемой хроматограммы, на которой уровень шума сопоставим с уровнем сигнала, и из-за этого её нельзя читать. Вероятно эта ДНК чем-то загрязнена.
Ссылки:
© Кузнецова Ксения, 2015