Нуклеотидный BLAST.
1. Определить таксономию и функцию прочтенной нуклеотидной последовательности из практикума 6.
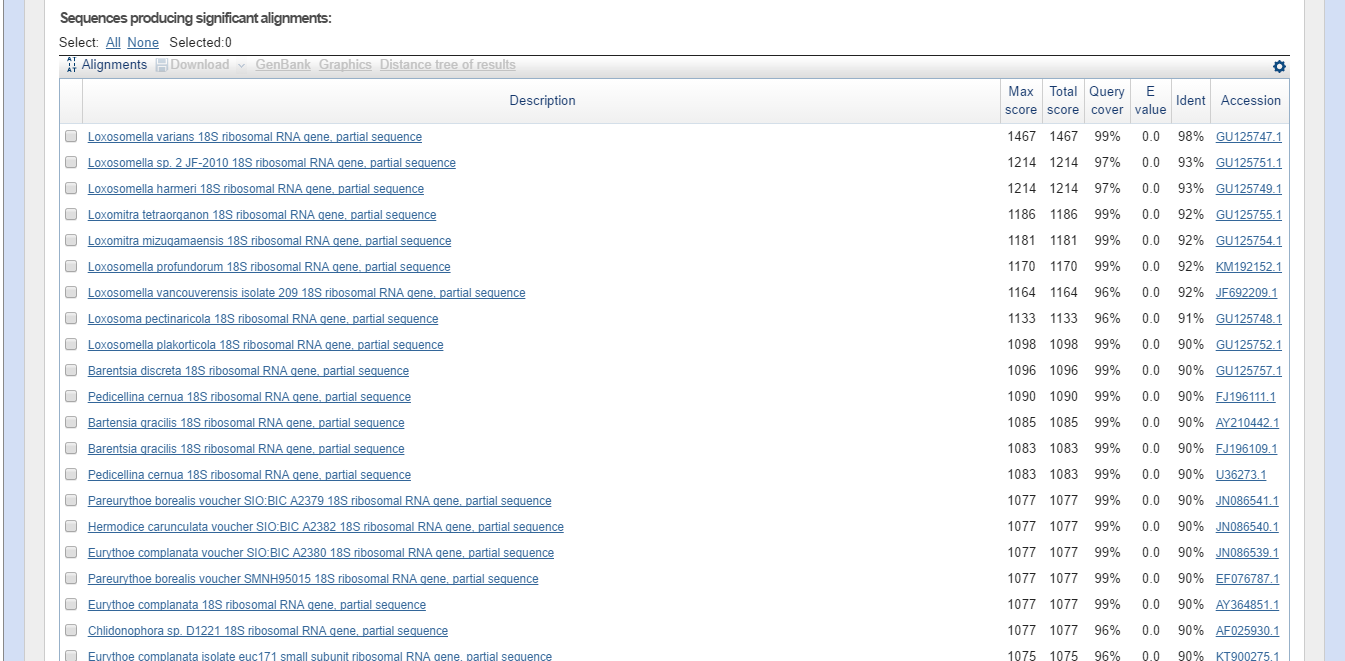
Прямая и обратная последовательности из практикума 6 были объединены в единую последовательность. Эта последовательность была введена в программу Standard Nucleotide BLAST (megablast) с произвольными параметрами. Выдача представлена на рисунке ниже.

По выдаче программы видно, что есть 9 находок указывающих на одно семейство Loxosomatidae и 18S рибосомальный РНК ген. В программе Jalview было построено выравнивание 9 лучших находок и исходной последовательности, его можно увидеть на рисунке снизу:

Из выравнивания видно, что исходная последовательность довольно сильно схожа с последовательностью вида Loxosomella varians. Таксономия: Eukaryota; Metazoa; Entoprocta; Loxosomatidae; Loxosomella varians.
2. Сравнение списков находок нуклеотидной последовательности 3-я разными алгоритмами BLAST.
Поиск по семейству Loxosomatidae дал слишком мало находок, поэтому при всех дальнейших запросах поиск велся по типу Entoprocta.
| Algoritm | Database | Max target sequences | Expect threshold | Word size | Max matches in a query range | Match/Mismatch Scores | Gap Costs |
| Highly similar sequences (megablast) | Nucleotide collection (nr/nt) | 1000 | 10 | 28 | 0 | 1,-2 | Linear |
| Optimize for More dissimilar sequences (discontiguous megablast) | Nucleotide collection (nr/nt) | 1000 | 10 | 11 | 0 | 2,-3 | Existence: 5 Extension: 3 |
| Somewhat similar sequences (blastn) | Nucleotide collection (nr/nt) | 1000 | 10 | 11 | 0 | 2,-3 | Existence: 5 Extension: 3 |
| Algoritm | Число находок | Находки найденные этим алгоритмом и не найденные другим |
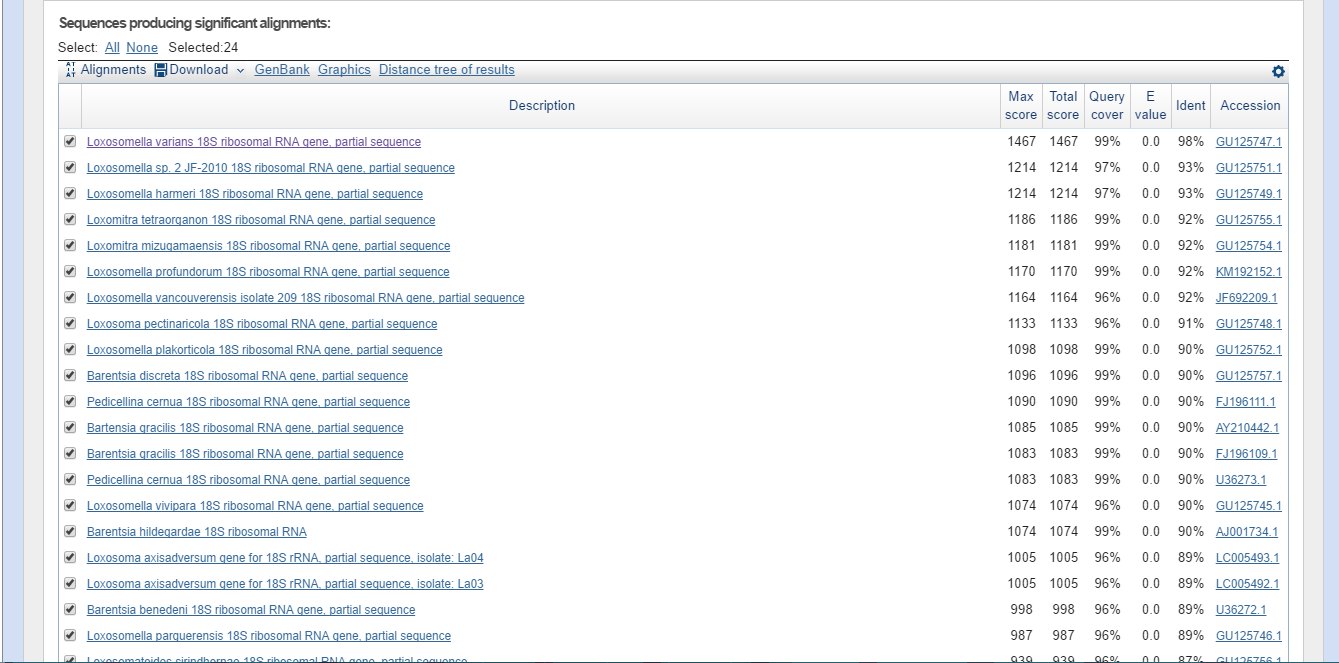
| megablast | 24 | C discontiguous megablast находки одинаковые, но в разном порядке (разные max score и total score и query cover у одних и тех же находок); совпадают 1, 7, 10, 11, 16, 24. blastn нашел больше находок. |
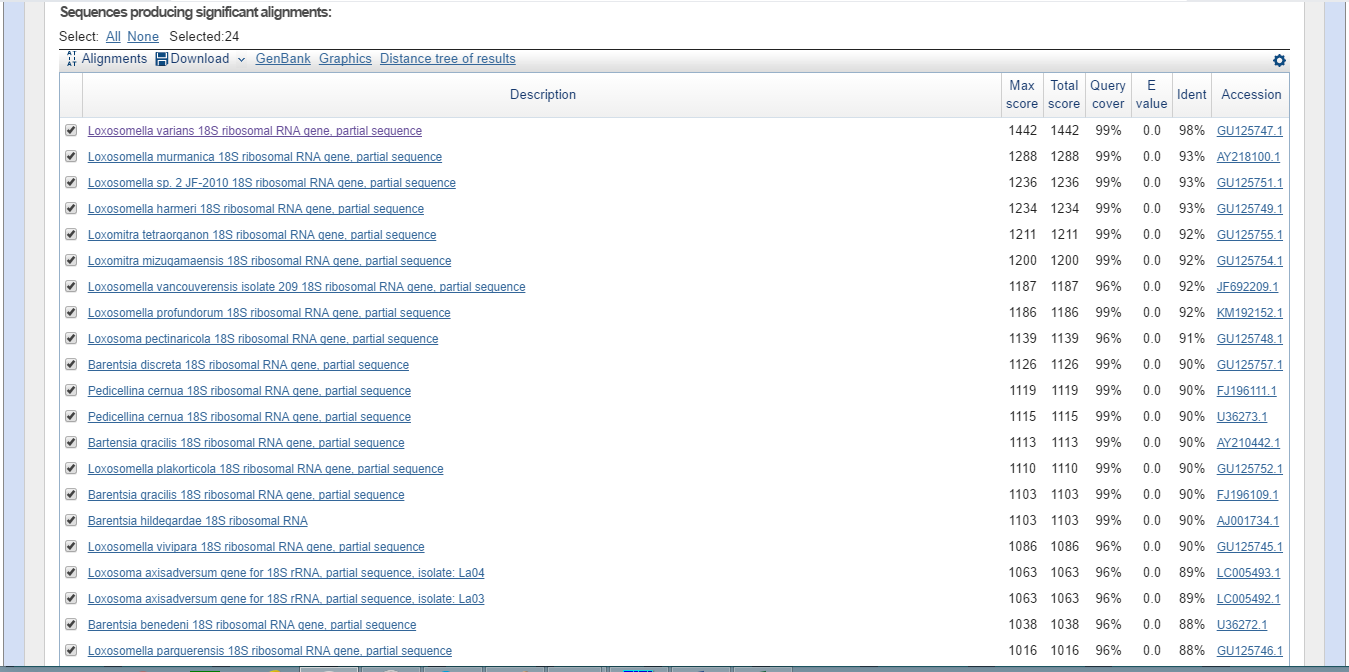
| discontiguous megablast | 24 | C megablast находки одинаковые, но в разном порядке (разные max score и total score и query cover у одних и тех же находок). С blastn совпадает по порядку находок, но у blastn больше находок. |
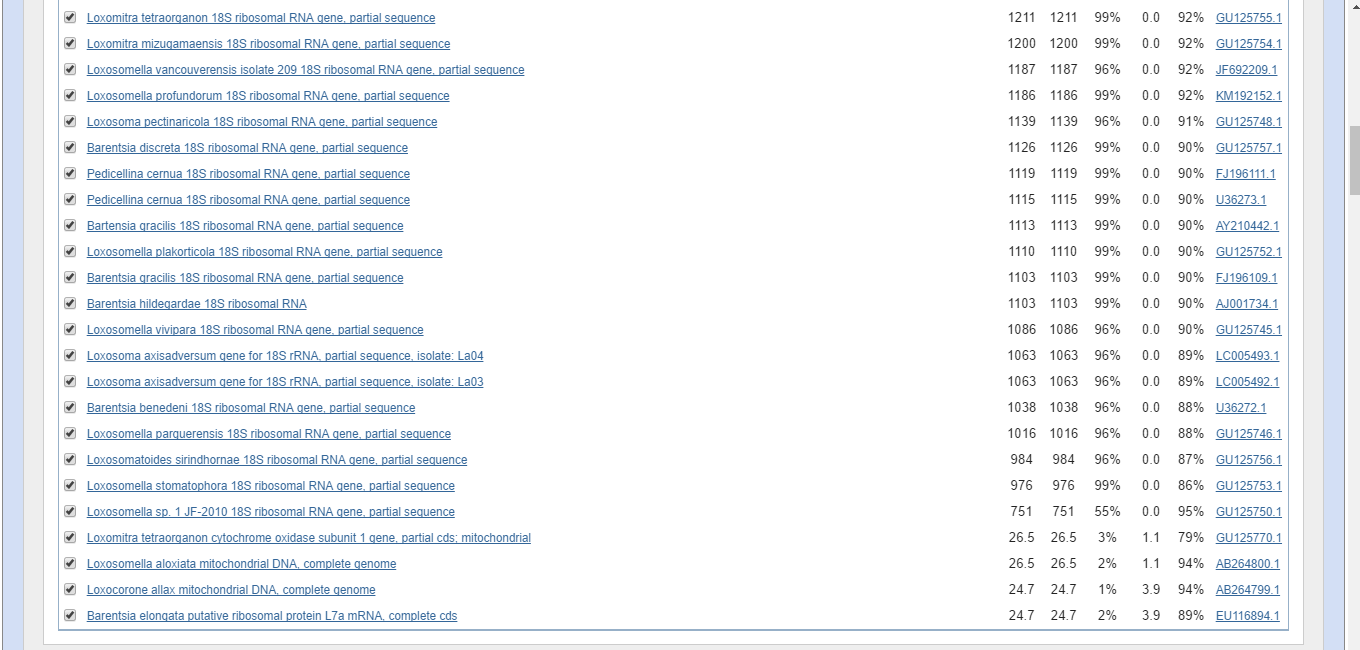
| blastn | 28 | Loxomitra tetraorganon cytochrome oxidase subunit 1 gene, partial cds; mitochondrial Loxosomella aloxiata mitochondrial DNA, complete genome Loxocorone allax mitochondrial DNA, complete genome Barentsia elongata putative ribosomal protein L7a mRNA, complete cds Эти находки с низкими max score, total score, query cover и высоким E-value. |
Находки megablast:
Находки discontiguous megablast:
Находки blastn (последние 4 найдены только этим алгоритмом):
Из полученных данных можно сделать вывод, что blastn и discontiguous megablast работают одинаково, с единственным отличием, что blastn находит немного больше находок. Blastn и megablast находят одни и те же находки, но отличные по параметрам max score, total score и query cover.
3. Проверить наличие гомологов трех белков в геноме организма Amoboaphelidium protococarum с помощью локального BLAST.
Для проверки были взяты белки HSP7C_HUMAN, TERT_HUMAN, CISY_HUMAN и геном организма Amoboaphelidium protococarum.
Сначала был создан банк данных для данного организма командой makeblastdb -in X5.fasta -dbtype nucl. Затем для каждого из белков был запущен алгоритм tblastn, который проверяет наличие гомологов данного белка в организме, командой tblastn -query xxx.fasta -db X5.fasta > xxx.out (вместо xxx подставляется название белка).
Проверка белка HSP7C_HUMAN. HSP7C_HUMAN - консервативный шаперон HSP70, белок теплового шока; имеется у большинства организмов из всех царств. Действует, как репрессор активации транскрипции. Компонент PRP19-CDC5L комплекса, который является неотъемлимой частью сплайсосомы и необходим для активации пре-мРНК сплайсинга. Ингибирует транскрипционный коактиватор CITED1 Smad-mediated транскрипции. Связывает бактериальный липополисахарид и является посредником ЛПС-индуцированной воспалительной реакции, включая секрецию ТНФ моноцитами.
Результатом проверки является файл HSP7C_HUMAN.out. Находок всего 16.

Лучшая находка - scaffold 199.
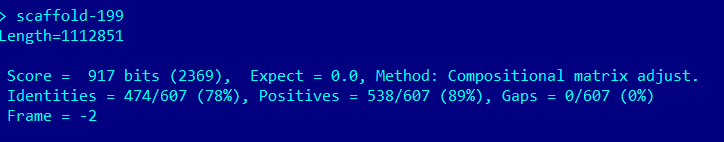
Лучшая находка имеет хороший E-value и остальные параметры, на основе этого я считаю, что эту находку можно назвать гомологом, и скорее всего она имеет схожие функции.
Проверка белка TERT_HUMAN. TERT_HUMAN - теломераза, восстанавливающая длину хромосомы при репликации; имеется у большинства (но не всех) эукариот. Активна в прогениторных клетках и раковых, в соматических имеет низкую активность. Играет важную роль в процессе старения и апоптоза. Катализирует РНК зависимые расширения 3'хромосомальной термини с 6-нуклеотидными теломерными повторами белка.
Результатом проверки является файл TERT_HUMAN.out. Находок всего 3.
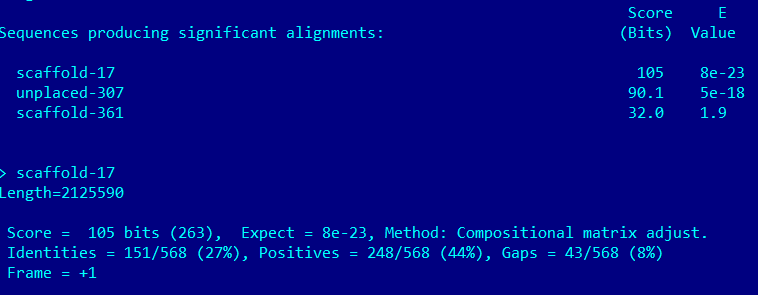
Лучшая находка - scaffold-17. Параметры сходства лучшей находки довольго низкие, что не позволяет нам говорить о сходных функциях, а также совпадения расположены равномерно по всей последовательности, из-за чего вероятнее всего о гомологии отдельных доменов мы говорить не можем.
Проверка белка CISY_HUMAN. CISY_HUMAN - митохондриальная цитратсинтаза. Каталитическая активность: Acetyl-CoA + H2O + oxaloacetate = citrate + CoA. Белок участвует в subpathway, который синтезирует изоцитрат из оксалоцитрата. Этот subpathway является частью цикла трикарбоновых кислот, который является частью углеводного обмена.
Результатом проверки является файл CISY_HUMAN.out. Находок всего 4.
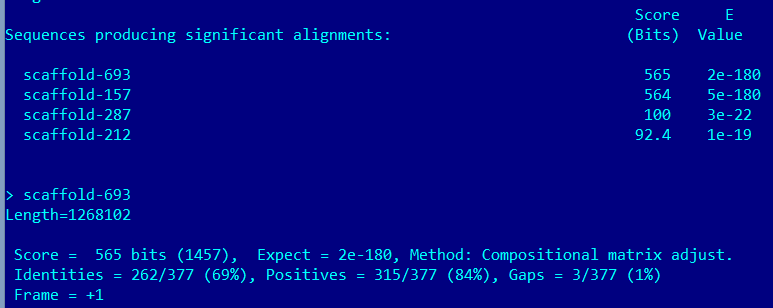
Лучшая находка - scaffold-693. Параметры сходства лучшей находки относительно хорошие, но не такие хорошие как у первого белка. На основе этого можно говорить о гомологии и, вероятно, сохранении функций.
4. Нахождение гена, закодированного в одном скэффолде Amoeboaphelidium.
Чтобы выбрать контиг, подходящей длины, была использованна команда infoseq пакета EMBOSS: infoseq X5.fasta -only -name -length. Был выбран контиг unplaced-1034 с длиной 25619 п.н., которой хватит для того, чтобы поместился ген. Для получения последовательности контига использовалась команда seqret: seqret X5.fasta:unplaced-1034 -out unplaced1034.out. Файл с последовательностью контига unplaced1034.out.
Последовательность контига была запущена в BLAST по алгоритму blastn по организму Amoeboaphelidium пристандартных параметрах. Выдача BLAST:
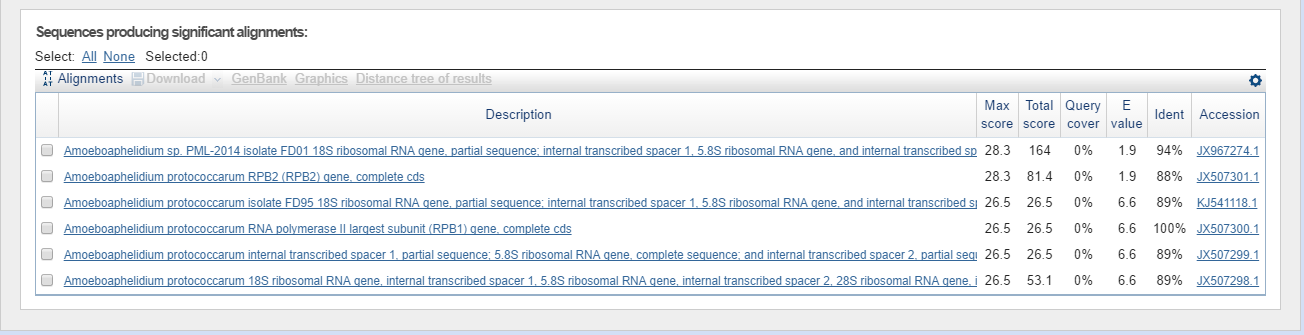
Находок всего 6, причем покрытие у всех 0% хотя Ident почти 100% и query cover большой. Параметры очень плохие, но вынужденно предполагаем, что в контиге закодираванны гены, которые закодированны в находках. В лучшей находке: частичная последовательность рибосомального гена 18S ПМЛ-2014 изолята FD01; полная последовательность внутреннего транскрибируемого сплайсера 1 5.8S рибосомального РНК гена и внутренноего транскрибируемого сплайсера 2; частичная последовательность 28S рибосомального РНК гена.
Ссылки:
© Кузнецова Ксения, 2015