Реконструкция эволюции доменной архитектуры
Задание 1. Построить выравнивание представителей домена Pfam белков с разной доменной архитектурой.
Для белка выданного мне в первом семестре не нашлось подходящего под требования семейства на сайте Pfam. Поэтому я поискала по своему организму Ruminiclostridium thermocellum. В результате выбрала семейство Amine oxidase (copper-containing) (AOC).
Аминоксидазы (содержащие медь). Это - семейство ферментов амин оксидазы, которое включает в себя первичные-аминоксидазы и диамин оксидазы; эти ферменты катализируют окисление широкого спектра биогенных аминов, в том числе многих нейромедиаторов, гистамина и ксенобиотиков аминов. Они действуют как дисульфид-связывающие гомодимеры. Они катализируют окисление первичных аминов до альдегидов с последующим выделением аммиака и перекиси водорода, которой необходим ион меди и topaquinone как кофактор.
| AC | ID | Функция | Ссылка |
| PF07833 | Cu_amine_oxidN1 | катализируют окислительное дезаминирование первичных аминов до соответствующих альдегидов | Copper amine oxidase N-terminal domain |
Cписок разных доменных архитектур с выбранным доменом и указанием числа последовательностей.
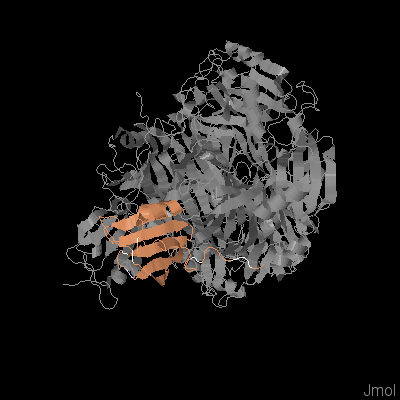
Также было построено выравнивание для выбранного семейства доменов. Ссылка на выравнивание в fasta . Ссылка на проект выравнивания. Кроме того, ниже представлена 3D структура одного из доменов (AMO_ECOLI; 1OAC).

Выбор архитектур для белка.
Для выбранного домена существует 432 архитектуры. Я выбрала архитектуру Cu_amine_oxidN1, AMIN, Amidase_3, содержащую 86 последовательностей. Она представлена на рисунке слева. Затем я выбрала архитектуру Cu_amine_oxidN1, Glyco_hydro_18, содержащую 118 последовательностей. Её рисунок расположен ниже справа.

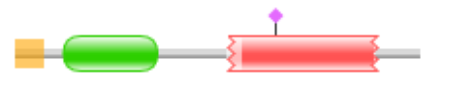
На рисунках оранжевым обозначен sig_p, зеленым наш домен - Cu_amine_oxidN1 (PF07833), красным на левом - Glyco_hydro_18 (PF00704), фиолетовым ромбиком - предсказанный Pfam сайт, голубым на левом - low_complexity, синим на левом - AMIN (PF11741), желтым - Amidase_3 (PF01520).
Была получена таблица с информацией об архитектуре всех последовательностей, содержащих выбранный домен с помощью скрипта swisspfam-to-xls.py, который отбирает последовательности с указанным доменом и составляет таблицу для Excel.
Команда: python swisspfam-to-xls.py -z /srv/databases/pfam/swisspfam.gz -p PF07833 -o PF07833.xls
Затем был составлен список последовательностей с указанием доменной архитектуры, используя сводную таблицу. В полученный Excel файл был добавлен лист с информацией о таксономии, который был получен скачиванием полных записей всех последовательностей с Uniprot и запуском скрипта uniprot-to-taxonomy, который преобразует информацию о таксономии из Uniprot в Excel. Кроме того в файл были добавлены длины для каждой последовательности.
Команда: python uniprot-to-taxonomy.py -i all.txt -o taxonomy.xls
Выбор таксона и подтаксонов.
В качестве таксона я выбрала Firmicutes, а в качестве подтаксонов Bacilli и Clostridia.
Затем я выбрала по 20 представителей каждого выбранного подтаксона и каждой выбранной архитектуры. Для них были придуманы удобные ID. Таблица.
Только отобранные мной последовательности из выбранных архитектур были оставлены в выравнивании с помощью скрипта filter-alignment.py.
Команда: python filter-alignment.py -i alin.fasta -m ids.txt -o part.fasta -a "/"
Полученное выравнивание было отредактировано в Jalwiev. Последовательности переименованы в удобные названия, где b обозначает подтаксон Bacilli, 1 - первую выбранную архитектуру Cu_amine_oxidN1, AMIN, Amidase_3, с - подтаксон Clostridia, 2 - 2ю Cu_amine_oxidN1, Glyco_hydro_18 архитектуру. Были удалены пустые колонки, последовательности объединены в две группы по архитектурам и раскрашены ClustalX. Также были удалены некоторые последовательности недостаточно хорошо выровненые и N-, C- концевые участки. Ссылка на проект.
Выравнивание правильное, так как нету фрагментов, нету последовательностей, резко отличающихся от выравнивания в целом, нет очевидных ошибок в выравнивании.
Задание 2: построение филогенетического дерева домена
В программе MEGA методом Neighbour Joining было построено филогенетическое дерево. Скобочная формула дерева.
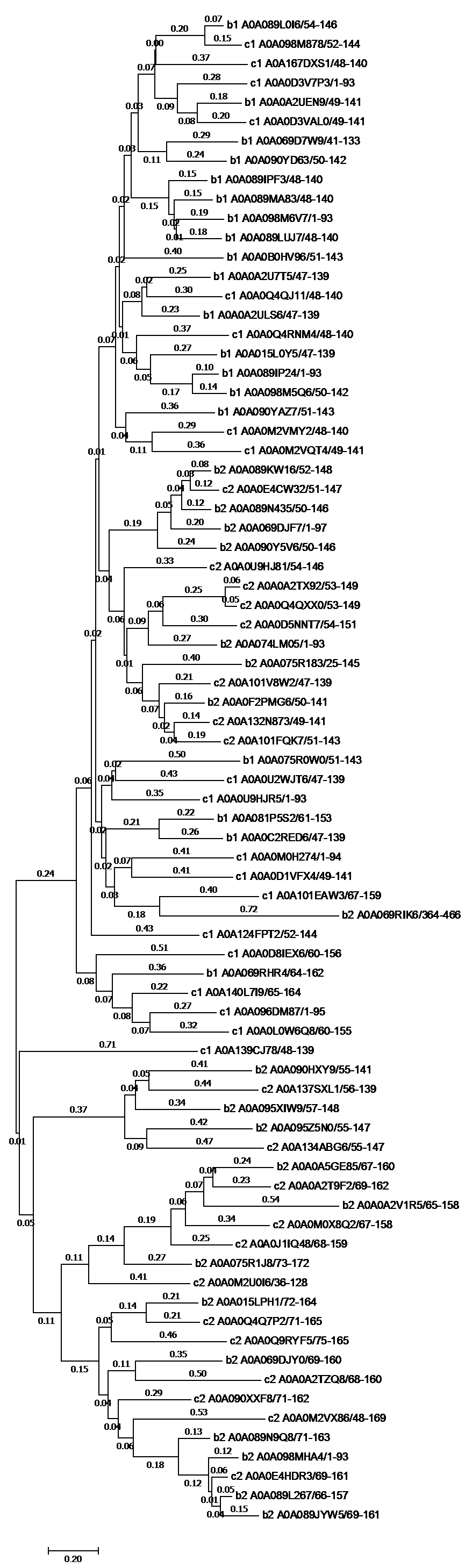

Слева дерево построенное в MEGA, справа дерево раскрашенное с помощью ITOL. Бирюзовым раскрашены ветви с последовательностями из подтаксона Bacilli и Cu_amine_oxidN1, AMIN, Amidase_3 архитектуры. Салатовым - из подтаксона Clostridia и Cu_amine_oxidN1, AMIN, Amidase_3 архитектуры. Красным - из подтаксона Bacilli и Cu_amine_oxidN1, Glyco_hydro_18 архитектуры. Розовым - из подтаксона Clostridia и Cu_amine_oxidN1, Glyco_hydro_18 архитектуры. Из раскрашенного дерева четко видны клады доменных архитектур, не считая нескольких попавших "не туда". Вероятно у предка была сначала 2я архитектура, затем возможно возникла 1я. 2я архитектура по-видимому возникала несколько раз на разных ветвях.
Ссылки:
© Кузнецова Ксения, 2015