Задание 4
1. Построение дерева по нуклеотидным последовательностям
Последовательности 16S рибосомальной РНК каждой из бактерий,которые я использовала в предыдущих практикумах, были скачены из из базы полных геномов NCBI. Были использованы: Enterococcus_faecalis_62_uid159663/; Streptococcus_pneumoniae_670_6B_uid52533/; Streptococcus_pyogenes_M1_GAS_uid57845/; Lactococcus_lactis_cremoris_KW2_uid219629/; Lactobacillus_delbrueckii_bulgaricus_ATCC_11842_uid58647/; Lactobacillus_acidophilus_30SC_uid63605/; Finegoldia_magna_ATCC_29328_uid58867/.
Последовательности были выровнены программой Clustal with Defaults, потому что при выравнивании Muscle выдавалась ошибка. Ссылка на выравние fasta
Дерево было построено в Mega методом Maximum likelihood. Полученное дерево изображено ниже.
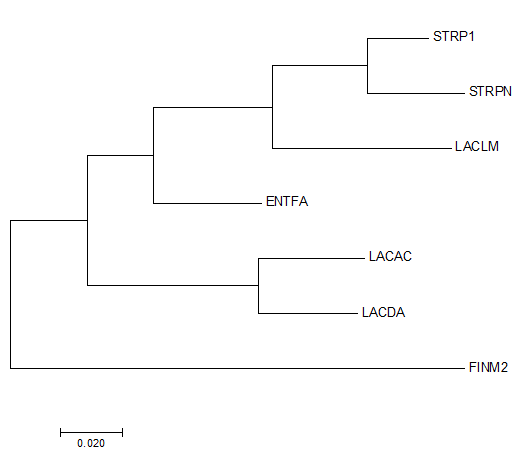
Дерево полностью совпадает с правильным. В случае с белками дерево тоже совпадало с правильным.
2. Построение и анализ дерева, содержащего паралоги
С помощью программы BLAST на сайте NCBI я нашла гомологи белка CLPX_BACSU в бактериях используемых мной ранее. Белки с хорошим E-value были выровнены и по ним построено дерерво программой Mega методом Neighbor-Joining. Ссылка на fasta файл с выравниванием.
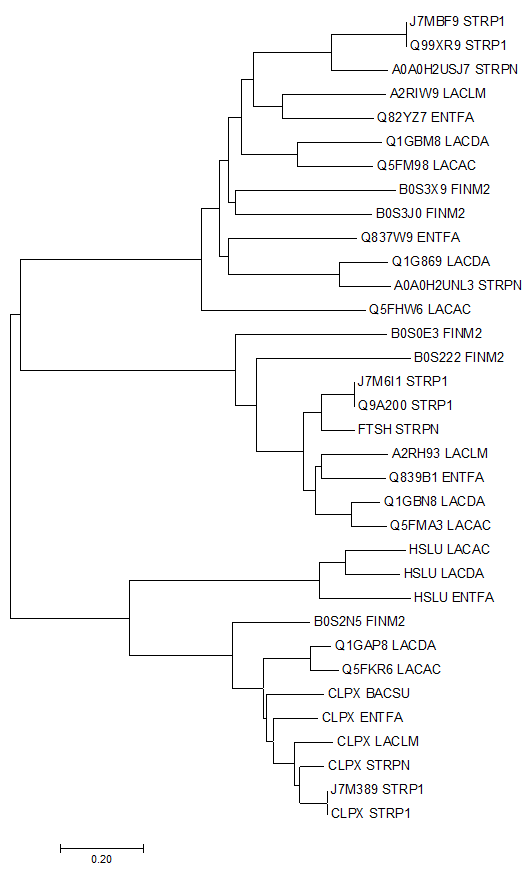
Паралогами будем называть два гомологичных белка из одного организма. Примерами паралогов на моем дереве являются: CLPX_STRP1 и J7M389_STRP1; J7M6I1_STRP1 и Q9A200_STRP1.
Ортологами будем называть два гомологичных белка, если они:а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования. Примерами ортологов на моем дереве являются: B0S222_FINM2 и J7M6I1_STRP1; HSLU_LACAC и HSLU_LACDA
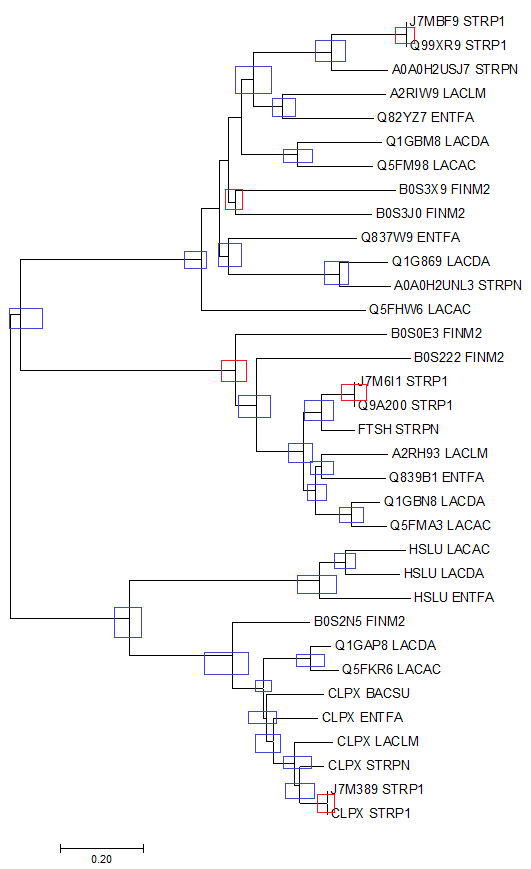
На рисунке ниже красным обозначена дуплекация гена, а синим видообразование.
Ссылки:
© Кузнецова Ксения, 2015