Геномное окружение. База данных GO
1. Получение информации о КОГе, к которому относится ваш белок
В 1м семестре мне был выдан белок с идентификатором YP_005686898.1, сейчас он переименован на WP_003514395.1 бактерии Ruminiclostridium thermocellum DSM 1313. В моем белке 870 аминокислотных остатков.
Используя сервис CDD и последовательность моего белка, я нашла хиты которые относят мой белок к тому или иному КОГу.
| ID | E-value | Interval | Название | Функциональная группа |
| COG4639 | 3.19e-49 | 7-175 | Predicted kinase Предсказаная киназа | R: General function prediction only Предсказание общей функции только |
| COG0645 | 1.40e-06 | 10-122 | Predicted kinase | R: General function prediction only |
2. Визуализация геномного окружения
Для COG4639 я получила изображение геномного окружения с помощью STRING.
Для начала я запустила STRING по организму Clostridium thermocellum. Параметры по умолчанию: Минимальный требуемый score взаимодействий = medium confidence 0,400; Максимальное количество взаимодействующих по первой линии белков = не более чем 10 взаимодействий; по второй линии взаимодействий нет.
Изображение полученое STRING
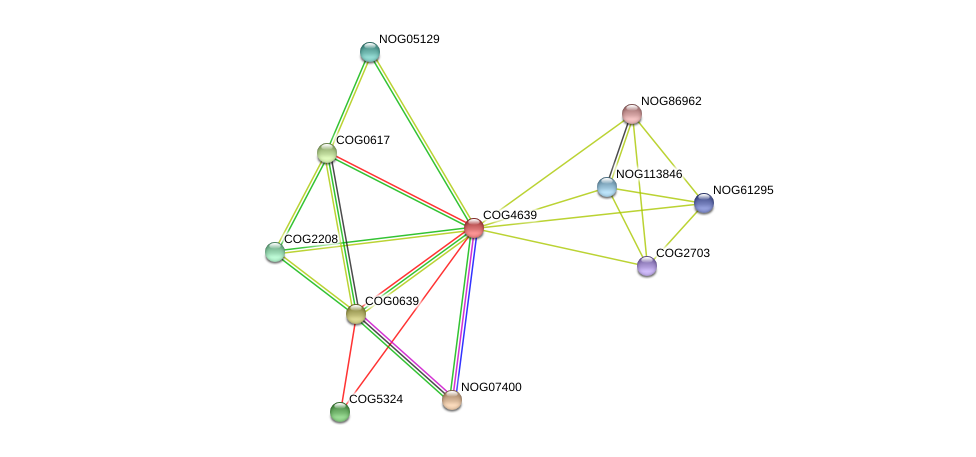
Каждый узел представляет собой все белки произведеные одним белок-кодирующим геномным локусом. Маленькие узлы - белки неизвестной 3D структуры. Большие узлы - некоторая 3D структура известна или предсказана. Ребра представляют собой белок-белковые взаимодействия (обязательно конкретные и содержательные). Известные взаимодействия: цветом морской волны обозначены полученные из курируемых баз данных, фиолетовым - экспериментально определенные. Предсказанные взаимодействия: зеленым - соседние в геноме, красным - слияние генов, синим - встречающиеся совместно гены. Другие: грязно салатовым - совместно упоминавшиеся в базе данных, черным - ко-экспрессию, голубым - гомологичность белков.
На рисунке видно всего лишь одно достоверно известное взаимодействие (фиолетовым). Хотя довольно много предсказанных взаимодействий, почти все взаимодействия совместно упоминаются в базе данных. Однако из рисунка ниже видно, что большинство белков еще не изучено (не наблюдаемая ортологичная группа). Из рисунка соседства генов видны два повторяющихся паттерна.
На рисунке ниже представлены найденые КОГи и их взаимодействия:
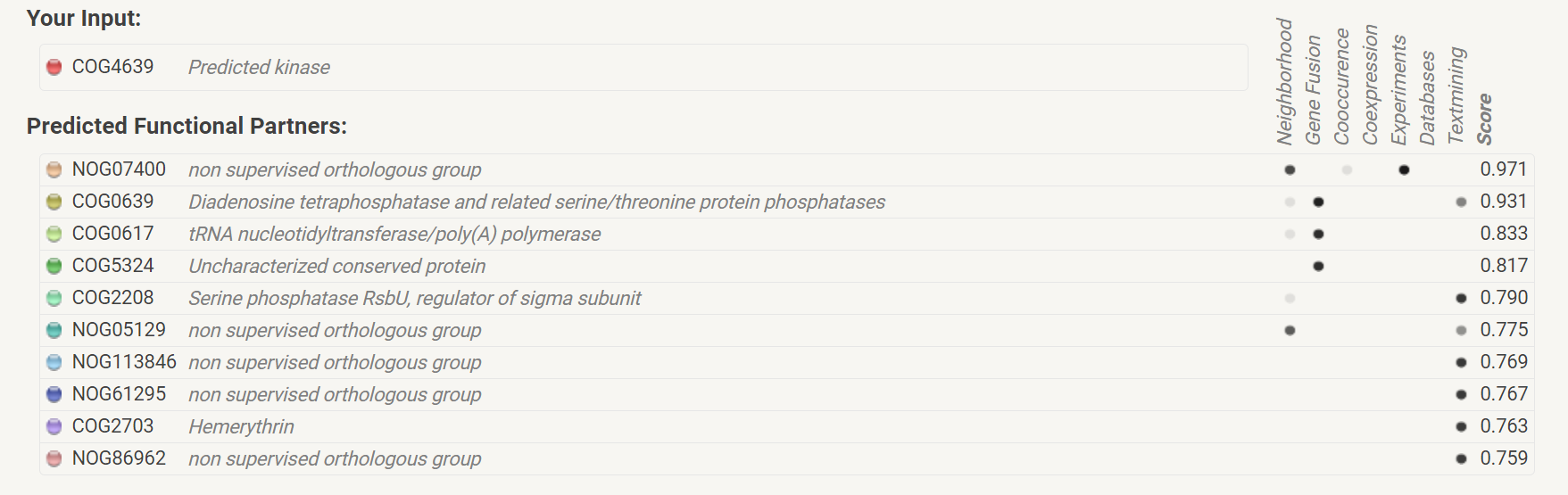
На рисунке ниже слева соседство генов, справа совместная встречаемость генов.
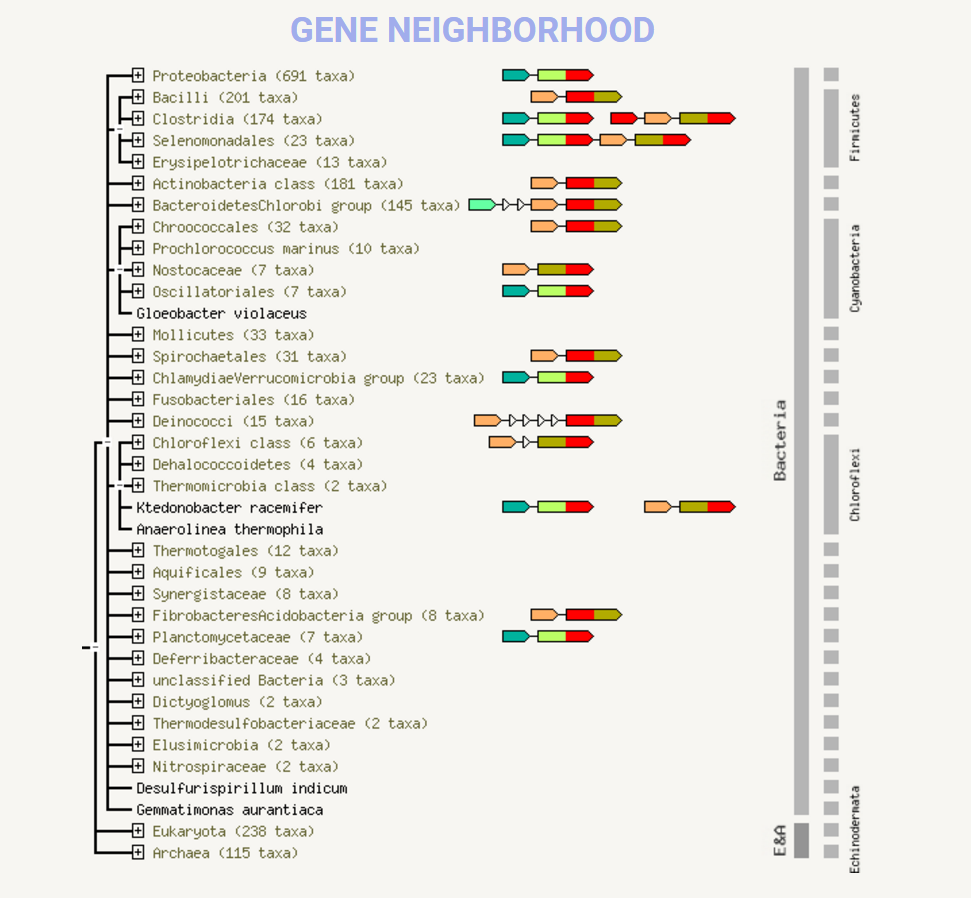

На рисунке слева вертикальная черта означает, что разные части гена могут иметь разную эволюционную историю. Первый встречающийся паттерн (цвет морской волны-салатовый) образован NOG05129 и COG0617. Второй паттерн (оранжевый-болотный) образован NOG07400 и COG0639. КОГи NOG05129 и NOG07400 не наблюдаемая ортологичная группа, поэтому нельзя говорить о каких либо функциях.
3. Отнесение белка Cthe-2768 (WP_003514395.1) из Ruminiclostridium thermocellum DSM 1313 к терминам GO
Используя инструмент AmiGo, поиском BLAST я обнаружила белок похожий на мой. Наиболее похож на мой белок белок PRPE_BACAN (название Bis(5'-nucleosyl)-tetraphosphatase PrpE [asymmetrical]) из организма Bacillus anthracis с P value 9.0e-48. Найденный белок не является тем же самым.Организмы моего белка и найденного относятся к одному отделу Firmicutes. Мой белок киназа, а найденный гидролаза. Они выполняют обратные функции.
Ниже можно увидеть выравнивание BLAST этих белков. E-value = 9.0e-48.

| Аспект | Идентификатор GO | Название термина | Перевод названия термина | Код типа достоверности |
| Биологический процесс (biological_process) | GO:0008150 | biological_process | Биологический процесс | ND |
| Код типа достоверности | Расшифровка кода типа достоверности | Объяснение |
| ND | No biological Data available (ND) evidence code | Используется для анотаций, в которых информация о молекулярных функциях, биологических процессах или клеточных компонентов гена или продукта гена не доступна. То есть аннотатор в базе данных не нашел никакой внушающей доверия информации или конкретных данных для аннотации. ND код указывает на то, что данный аспект находится в процессе исследования и не используется для получения аннотации. Это значит, что генный продукт аннотированный в GO не подходит для того, чтобы связать конкретный GO термин с конкретным генным продуктом. Таким образом в дополнение к негативной аннотации должна быть положительная аннотация, если все таки ничего не известно о роли генного продукта в данном аспекте, то положительная аннотация вносится в корневой узел аспекта, используя код ND. |
Я с помощью инструмента AmiGO нашла белок наиболее похожий на мой и в таблице 1 отнесла к терминам GO. Но к сожалению судить о функциях белка по такому плохому коду достоверности не представляется возможным без доскональной проверки данных и экспериментальных подтверждений.
Ссылки:
© Кузнецова Ксения, 2015