Поиск сигналов. Теория
1. Определить биологическую роль транскрипционного фактора (SaeR) в бактерии Staphylococcus epidermidis ATCC 12228.
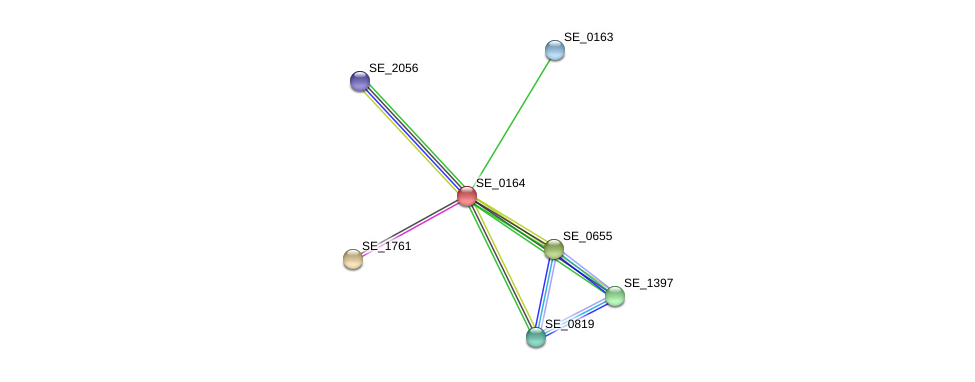
С помощью программы MEME я нашла единственный мотив связывания. Ниже приведены его параметры, logo и PWM.
E-value: 3.5e-137 Site Count: 45 Width: 16

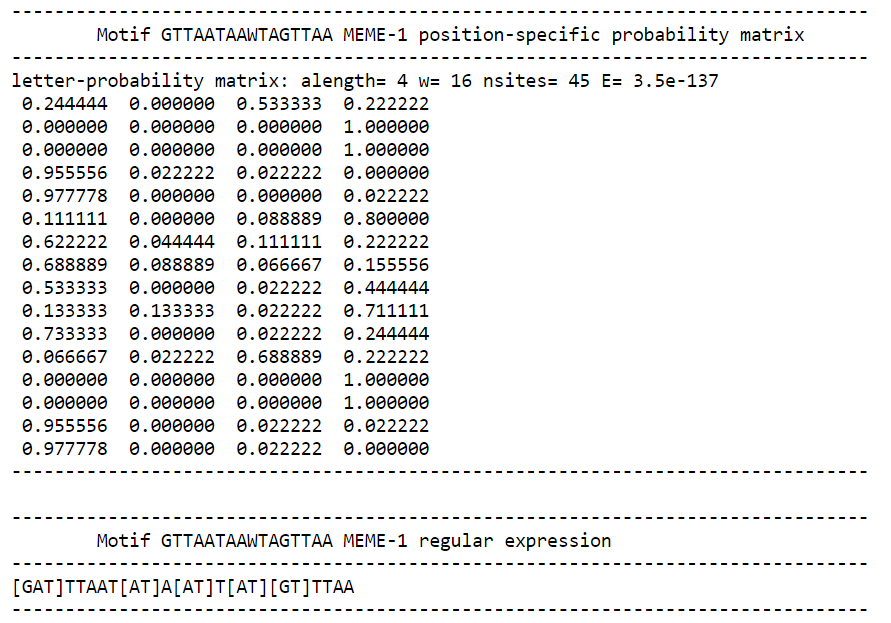
С помощью сервиса tomtom я нашла похожий мотив. Его имя EXPREG_00000800 (ArcA_E.coli). Параметры:
p-value = 7.03e-06
E-value = 5.90e-04
q-value = 1.14e-03
length=21
| 0.469651 | 0.0454785 | 9.22856e-05 | 0.484779 |
| 0.15195 | 0.0454785 | 0.0757357 | 0.726837 |
| 0.0157932 | 9.22856e-05 | 0.938065 | 0.0460504 |
| 0.0309213 | 0.0303494 | 9.22856e-05 | 0.938637 |
| 0.212464 | 0.0152214 | 0.0152214 | 0.757094 |
| 0.893251 | 9.22856e-05 | 0.0606066 | 0.0460504 |
| 0.605808 | 0.227021 | 0.0152214 | 0.15195 |
| 0.394007 | 0.151378 | 0.105993 | 0.348622 |
| 0.424264 | 0.0303494 | 0.227021 | 0.318365 |
| 0.560422 | 0.166507 | 0.0454785 | 0.227593 |
| 0.605808 | 0.0908638 | 0.0303494 | 0.272979 |
| 0.409136 | 0.0303494 | 0.196764 | 0.36375 |
| 0.0763075 | 0.196764 | 0.0606066 | 0.666323 |
| 0.0914356 | 9.22856e-05 | 0.877551 | 0.0309213 |
| 0.0460504 | 9.22856e-05 | 9.22856e-05 | 0.953765 |
| 0.167079 | 9.22856e-05 | 0.121121 | 0.711708 |
| 0.787351 | 0.0303494 | 0.0908638 | 0.0914356 |
| 0.590679 | 0.272407 | 0.0152214 | 0.121693 |
| 0.484779 | 0.13625 | 0.24215 | 0.136822 |
| 0.424264 | 0.0908638 | 0.181635 | 0.303236 |
| 0.499908 | 0.13625 | 0.166507 | 0.197336 |
На рисунке ниже выравнивание мотива найденного MEME (он снизу) и похожего мотива найденного tomtom (сверху).
Главным образом эти мотивы различаются длинной и PWM матрицей.
Затем с помощью программы FIMO я нашла этот мотив в геноме бактерии Staphylococcus epidermidis ATCC 12228.
Ссылка на таблицу всех находок.
| motif_alt_id|sequence_name | start | stop | strand | p-value | Координаты в геноме |
| NP_763718.1|SE0163 | 139 | 154 | + | 9.87e-09 | 150959-150974 |
| NP_763719.1|uhpT | 84 | 99 | - | 9.87e-09 | 151636-151651 |
| NP_764036.1|SE0481 | 327 | 342 | + | 8.41e-08 | |
| NP_764037.1|SE0482 | 64 | 79 | - | 8.41e-08 | |
| NP_763886.1|SE0331 | 310 | 325 | - | 1.79e-07 | |
| NP_763780.1|SE0225 | 75 | 90 | + | 1.02e-06 |
Мотив нужно искать в upstream region, потому что связывание ТФ происходит на участке расположенном до гена, т. е. upstream. Извлеченны координаты двух лучших находок complement(150820..151314); 151552..152931.
Найденные гены не входят в один метаболический путь KEGG. К тому же белок SE0163 является гипотетическим. Однако две лучшие находки входят в консервативное геномное окружение БД STRING. Ген uhpT переименовался в SE0164.

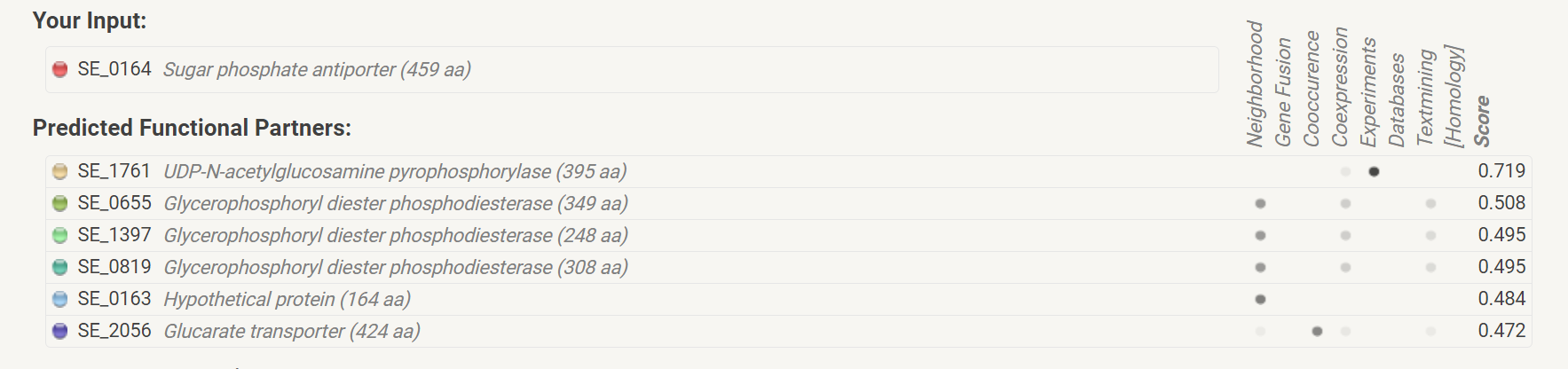
Ген uhpT является антипортером сахарной фосфотазы (Sugar phosphate antiporter).
Задание 2. Проверьте, может ли метилирование повлиять на связывание вашего ТФ со своим сайтом?
С помощью fuzznuc из пакета EMBOSS были найдены все сайты, пересекающиеся с предсказанными сайтами связывания ТФ SE0163 и uhpT, в бактерии Staphylococcus epidermidis ATCC 12228. Для этого использовался файл с известными последовательностями сайтов метилирования sites.fasta. Также были извлечены последовательности в fasta мотивов SE0163 и uhpT увеличенные на 50 нуклеотидов, чтобы не пропустить пересечение. Файлы SE0163 и uhpT.
Команда: fuzznuc -sequence SE0163_2.fasta -pattern @sites.txt -outfile out.out
Команда: fuzznuc -sequence uhpT_2.fasta -pattern @sites.txt -outfile out_2.out
Для SE0163 было найдено 126 последовательностей. Для uhpT было найдено 120 последовательностей. Однако среди находок имеются однобуквенные последовательности, которые не несут смысла, как мне кажется.
Затем я проверила есть ли в геноме бактерии Staphylococcus epidermidis ATCC 12228 ДНК метилтрансферазы с той же специфичностью с помощью REBASE. М генов в этом геноме 0.
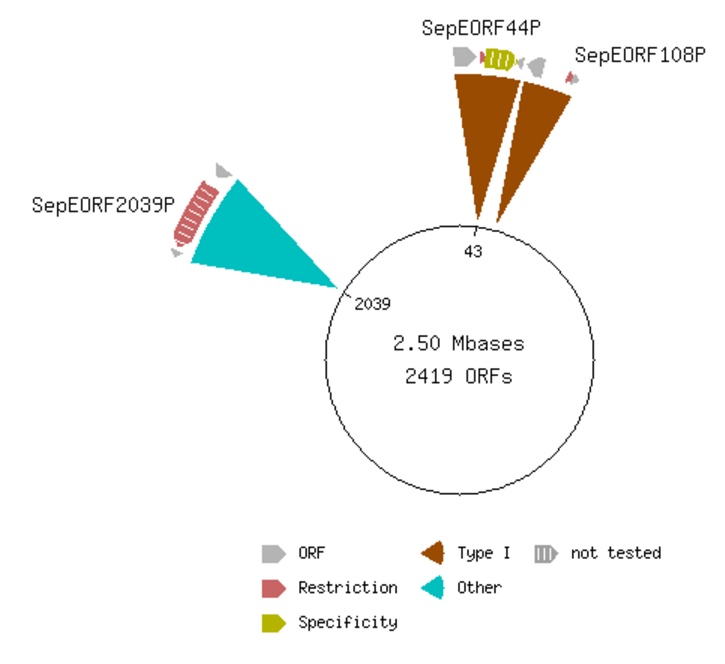
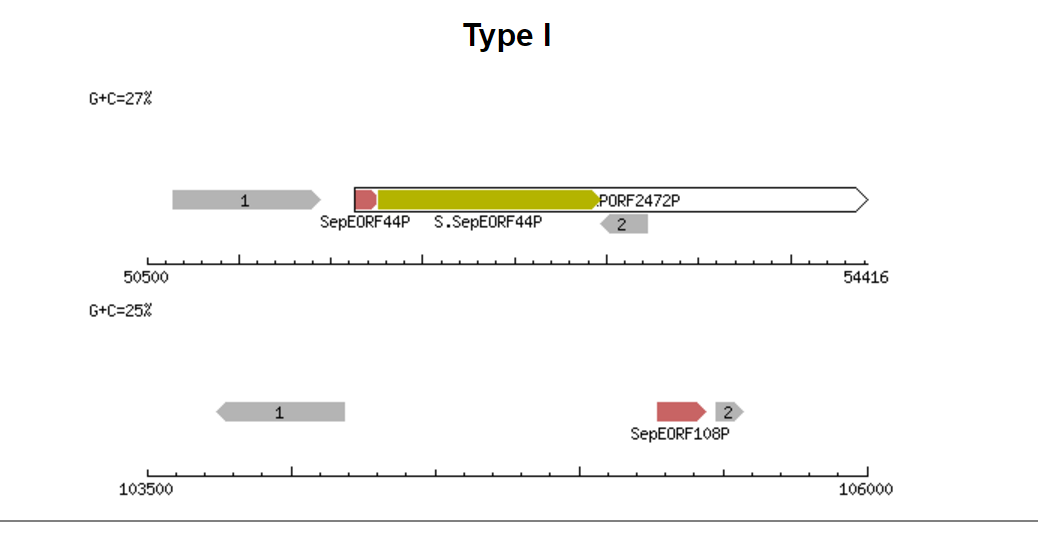
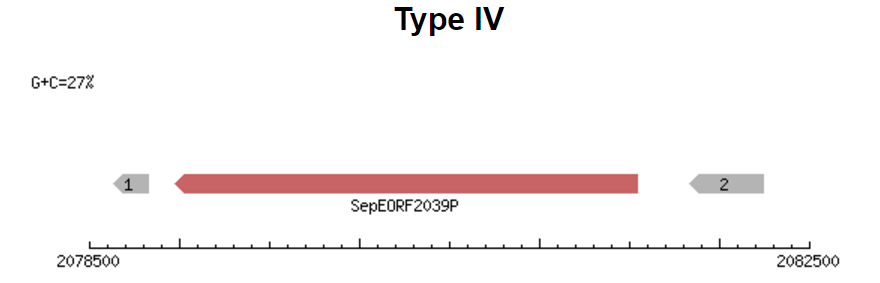
Видно, что метилтрансфераз нет. Ясно, что метилирование в моем случае не влияет на связывание ТФ со своим сайтом.
Ссылки:
© Кузнецова Ксения, 2015