|
||||
|
ЕС код фермента белка 6PGD_ECOLIНа странице белка 6PGD_ECOLI БД UniProt был найден код EC=1.1.1.44По данным базы IUBMB код был расшифрован:
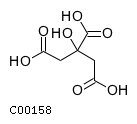
красным отмечен катализатор, синим - продукты реакции, зеленым - реагенты. Метаболические пути фермента из белка 6PGD_ECOLIВ записи о белке 6PGD_ECOLI в БД UniProt было найдено название локуса гена: b2029.В результате поиска гена по названию локуса в базе KEGG среди генов Escherichia coli K12 была найдена запись eco:b2029. В поле Pathways этой записи содержатся 3 идентификатора метаболических путей, ассоциированных с этим геном: ID eco00030 Pentose phosphate pathway Пентозофосфатный путь посмотреть карту пути Красным отмечен фермент 6-фосфоглюконат дегидрогеназы ID eco00480 Glutathione metabolism Метаболизм глутатиона ID eco01100 Metabolic pathways Метаболические пути Структурные формулы химических соединенийВ базе данных химических соединений KEGG LIGAND был проведен поиск двух веществ:Цитрат / Citrate ID C00158 Структура: Поиск метаболического пути между двумя веществамиПоиск осуществлялся в БД KEGG Pathway между веществами цитрат и глиоксилат (см. предыдущее задание). Было найдено 2 карты, в которых одновременно присутствовали оба соединения. Одна из них - Metabolic pathways, общая карта. Другая - Glyoxylate and dicarboxylate metabolism, карта для более специфических путей, она и была выбрана.Выбранная цепочка ферментативных реакций: путь: метаболические пути глиоксилата и дикарбоксилата цепочка: цитрат → глиоксилатПуть от цитрата к глиоксилату через промежуточные соединения цис-акониат и изоцитрат (3 стадии катализа) был отмечен на карте: посмотреть * красным отмечено первое соединение цепочки, желтым - промежуточные, зеленым - последнее соединение цепочки Сравнение метаболических путей у разных организмовКарта, полученная в предыдущем задании, была переведена в режим разных организмов. В некоторых из этих организмов ферменты, необходимые в реакции, оказались незакодированы.
в Escherichia coli K-12 MG1655 все ферменты цепочки присутствуют в Archaeoglobus fulgidus 1 фермент (EC 2.3.3.9) отсутствует (возможно, еще не обнаружен) в Arabidopsis thaliana все ферменты цепочки присутствуют в Homo sapiens 1 фермент (EC 2.3.3.9) отсутствует (возможно, еще не обнаружен)Таким образом, альтернативный путь может оказаться более распространенным, так как потенциально во всех 4 организмах путь возможен, в то время как первый путь точно не возможен в Archaeoglobus fulgidus. Сравнение ферментов из далеких организмовДля сравнения был взят фермент с ЕС=1.11.1.6. Этот фермент, согласно расшифровке IUBMB, является оксиредуктазой, использующей пероксид, как акцептор, а конкретно - каталазой: она разлагает образующуюся в процессе биологического окисления перекись водорода на воду и молекулярный кислород. В SRS был создан запрос для поиска фермента среди белков человека и археи Archaeoglobus fulgidus.Текст запроса (опция маски для ECNumber была снята, чтобы не были найдены такие EC, как 1.11.1.60 и проч): (([uniprot-ID:*_human*] | [uniprot-ID:*_arcfu*]) & [uniprot-ECNumber:1.11.1.6])Было 4 находки, среди которых 3 принадлежат человеку, а одна - архее: CATA_HUMAN D3DR07_HUMAN B4DWK8_HUMAN KATG_ARCFUПри этом при переходе в UniRef100 осталось только 3 разные записи (D3DR07_HUMAN и CATA_HUMAN соответствовали одной записи UniRef). В режиме SW_InterProMatches были сравнены доменные организации найденных белков. Для белков человека в Pfam описаны 2 домена: PF00199 Catalase/каталаза. PF06628 Catalase-related immune-responsive/домен, относящийся к каталазе, отвечающий на иммунный сигнал. Для белка археи в Pfam описан дублированный домен: PF00141 Peroxidase/пероксидаза. Пероксидазы и каталазы являются родственными ферментами (пероксидаза имеет EC=1.11.1.7 и участвует в реакциях окисления доноров электронов с восстановлением пероксидов), каталаза может выступать в роли пероксидазы. В некоторых микроорганизмах, в том числе в рассматриваемой архее, каталаза выполняет дополнительную роль пероксидазы и носит название каталазы-пероксидазы. Для дальнейшего анализа были выбраны белки CATA_HUMAN и KATG_ARCFU Поиск ортологов: Ген, соответствующий белку CATA_HUMAN, - CAT, id в KEGG - hsa:847. Ген, соответствующий белку KATG_ARCFU, - katG, в KEGG - afu:AF2233 Для гена белка CATA_HUMAN лучшим ортологом из архей стал Mbur_0523, каталаза, UniProt ID находки - Q12YH1_METBU, организм Methanococcoides burtonii (strain DSM 6242). Процент идентичности 0.567, перекрытие последовательностей 494. Для гена белка KATG_ARCFU лучшим ортологом среди эукариот стал AO090010000722, каталаза-пероксидаза, UniProt ID находки - KATG_ASPOR, организм Aspergillus oryzae. Процент идентичности 0.574, перекрытие последовательностей 734. Таким образом, гомолог человеческого белка в археях имеет ферментативную функцию каталазы (как и белок человека), а гомолог белка археи в эукариотах имеет ферментативную функцию каталазы-пероксидазы (как и белок археи, причем организм, в котором нашелся гомолог, принадлежит к царству грибы). Археи и человек находятся на огромном эволюционном расстоянии друг от друга. Однако они обладают ферментами, выполняющими сходные функции: восстановление пероксидов. Но несмотря на сохранение функции ферментов, ближайшие гомологи человеческого фермента в археях и фермента архей в млекопитающих находятся (хоть и с хорошим процентом идентичности: более 50%) в организмах, удаленных от Archaeoglobus fulgidus и человека соответственно. Таким образом, в процессе эволюции функция фермента сохранялась, последовательность менялась сильнее. |
© Яшина 2009
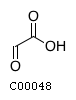
 Глиоксилат / Glyoxylate
ID C00048
Структура:
Глиоксилат / Glyoxylate
ID C00048
Структура: