Выравнивание последовательностей может дать нам много информации о гомологии совмещаемых последовательностей. При построении множественных выравниваний последовательности белков располагаются друг над другом, а для совмещения последовательностей вставляются гэпы - формальные символы, не обозначающие делеции. Работа с множественными выравниваниями осуществляется при помощи Jalview.
Чтобы выполнить первое задание, я установил консервативность 70% и выше, раскраску Clustalx. Не было найдено достаточно больших блоков, на которых наблюдалась бы гомология. Но, тем не менее, было найдено 2 участка выравнивания с высоким процентом консервативных аминокислотных. Эти участки показаны на рис.1. Эти участки нельзя объединить в кластер, потому что данные блоки разделены недостаточно консерватинвыми учатками.
Выравнивание, с которым была проведена работа, можно скачать здесь
Проект в формате JVP с выравниванием данных последовательностей можно скачать здесь.
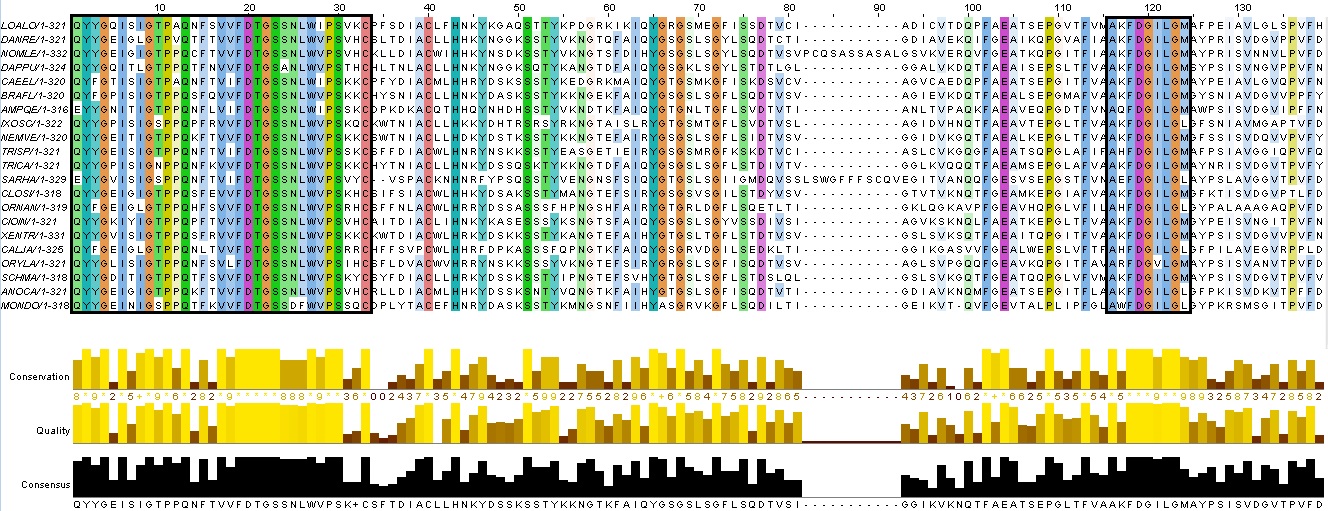 Рис. 1. Общий вид проекта в Jalview, вертикальные блоки выравнивания показаны черной рамкой, им соответствуют координаты 1-33, 116-124
Рис. 1. Общий вид проекта в Jalview, вертикальные блоки выравнивания показаны черной рамкой, им соответствуют координаты 1-33, 116-124
Для выполнения последующих заданий я выбрал первый блок. Всего в нем 33 колонки.
Абсолютно консервативных позиций (одинаковая аминокислота в каждой позиции) - 16 (48.5%).
Абсолютно функционально консервативных позиций (аминокислоты одной группы в каждой позиции)25 (75.8%).
Консервативная на 70% колонка - колонка, в которой в 70% выравниваемых последовательностей и более стоит одна и та же аминокислота - 28 (84.8%).
Функционально консервативных на 70% - 27 (81.8%).
Между блоками 82 позииций. 11 - гэпы (13.4%). Две отдельно гомологичные - 1-66 (2, 3 строки).
Пункт Б первого задания заключался в поиске двух гомологичных на каком-либо участке последовательностей, при отсутствии данных о гомологичности остальных последовательностей. Графические параметры остались как в прежней работе. Требуемый фрагмент выравнивания показаны на рис. 2, ему соответствуют кординаты последовательности 266-293, легко увидеть, что выделенные последовательности гомологичны на данном участке, при том, что совпадения среди остальных последовательностей, имеют скорее случайных характер. Скачать проект в формате JVP можно тут здесь
 Рис. 2 Две гомологичные на участке последовательности.
Рис. 2 Две гомологичные на участке последовательности.
Для выполнения четвертого задания нужно было посторить выравнивание дополнительной последовательности (ее можно скачать здесь), относительно одного из блоков из первого задания. Фрагмент этого выравнивания показан на рис.3. Исходя из этого множественного выравнивания, можно сказать, что добавленная последовательность гомологична всем остальным на представленном участке (1-30).
Проект в формате JVP с выравниванием данной последовательности можно скачать здесь.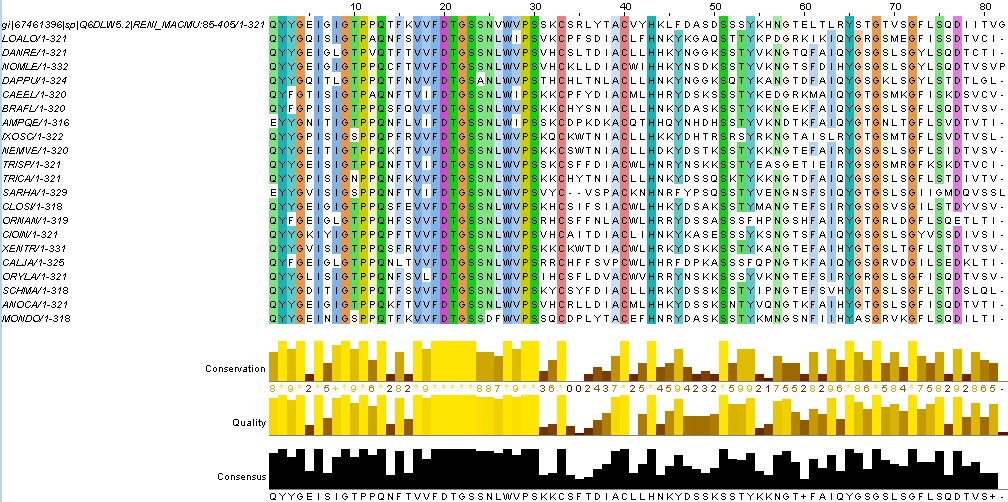 Рис. 3 Выравнивание дополнительной последовательности с первым блоком из задания 1
Рис. 3 Выравнивание дополнительной последовательности с первым блоком из задания 1
Пятое задание заключалось в получении косенсусной последовательности и постоения LOGO-изображения одного из блоков. Последовательность была получена с помощью инструментов Jalview и скачать ее можно здесь. Для построения LOGO использовались фрагменты последовательностей, соответствующие 2-му (правому) вертикальному блоку выравниваний. И увидеть его можно на рис. 3.
 Рис. 4. LOGO-изображение левого вертикального блока из пункта а первого задания.
Рис. 4. LOGO-изображение левого вертикального блока из пункта а первого задания.
Для выполнения последнего задания мы посмотриим, как выглядит выравнивание заведомо негомологичных белков. Для этого несколько аминокислотных последовательностей были выравнены при помощи MUSCLE. Полученный в итоге файл можно скачать здесь. Этот файл подается на вход редактору JalView. На рассматриваемых участках мало совпадающих аминокислот, а значит белки негомологичны.
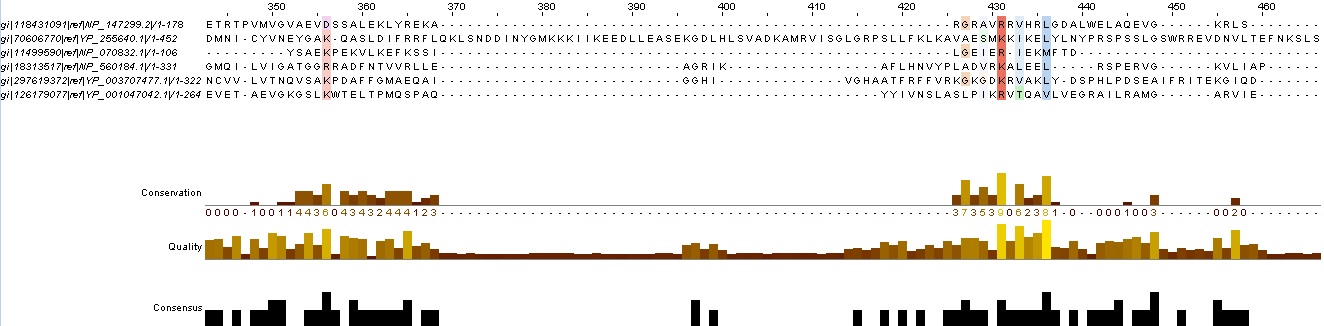 Рис. 5. Два блока выравнивания заведомо негомологичных белков.
Рис. 5. Два блока выравнивания заведомо негомологичных белков.
Проект этой работы с выравниванием заведомо негомологичных белков можно скачать здесь
Дата последнего изменения - 16 марта 23:54