Был проведен поиск гомологов последовательности в банке Uniprot/SwissProt с помощью программы protein blast (blasp). Процесс поиска представлен на рисунке 1, первый результат - исходный белок.
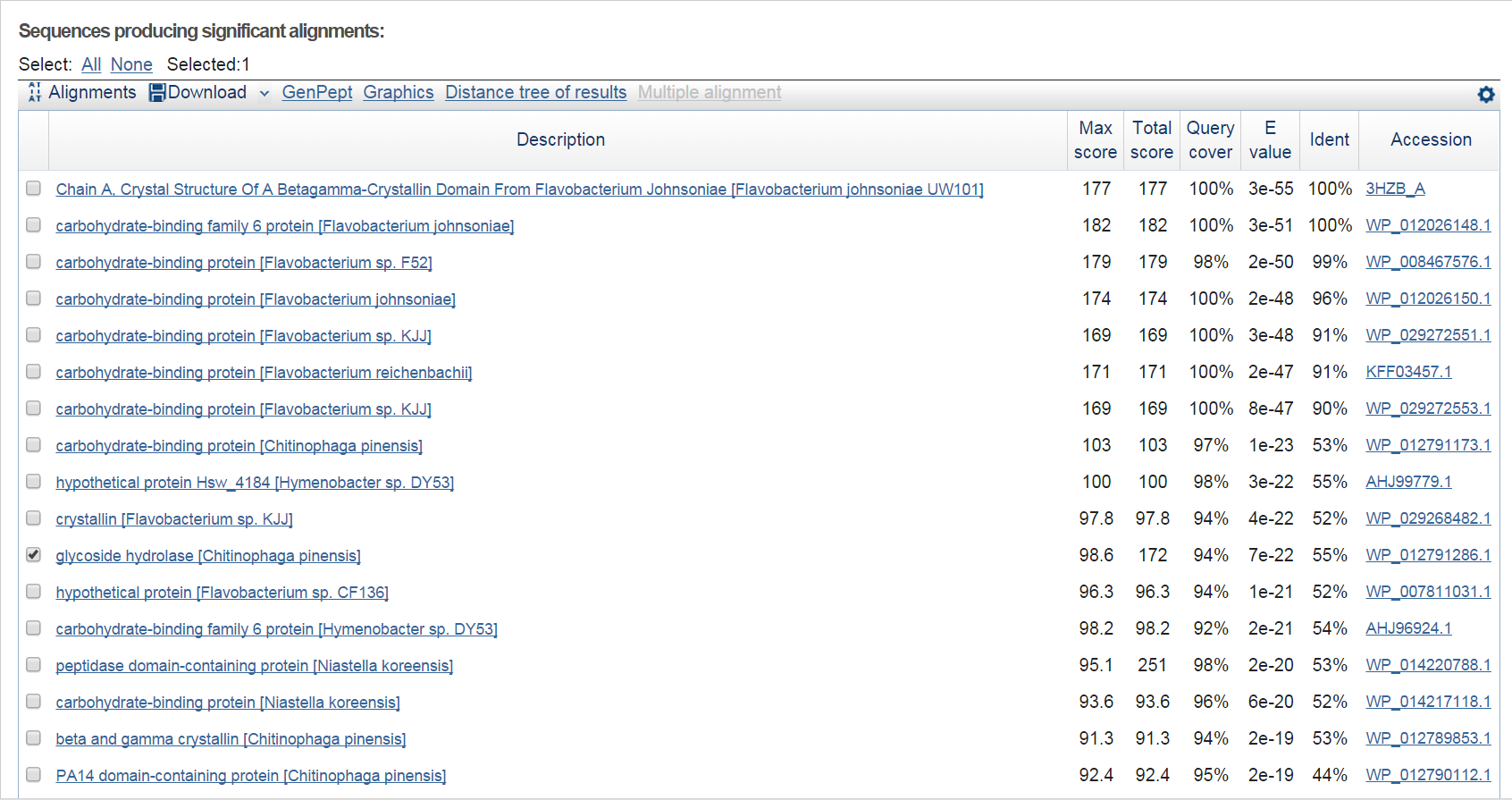
Рис. 1. Результаты работы Protein BLAST
- Поле Description - краткое описание белка.
- Max score - максимальный вес выравнивания.
- Total score - общий вес, он может отличаться от максимального, если в последовательностях выравнялось несколько участков.
- Query cover - покрытие последовательности, т.е какая часть последовательности покрыто выравниванием.
- E-value - величина, характеризующая математическое ожидание того, что в случайно м банке аминокислотных последовательностей такой же длины и такого же аминокислотно состава найдется последовательноость, дающая такой или более вес. Соответсвенно, чем она меньше, тем больше мы можем доверять выравниванию.
- Ident - процент совпавших позиций в двух последовательностях.
- Accession - код белка(AC) в базе данных.
Таблица 1. Характеристика выбранной мною находки и лучшей находки.
| Description | Max score | Total score | Query cover | E value | Ident | Accession |
| glycoside hydrolase [Chitinophaga pinensis] | 98.6 | 172 | 94% | 7e-22 | 55% | WP_012791286.1 |
| Chain A, Crystal Structure Of A Betagamma-Crystallin Domain From Flavobacterium Johnsoniae [Flavobacterium johnsoniae UW101] | 177 | 177 | 100% | 3e-55 | 100% | 3HZB_A |
Мною была построена карта локального сходства моей последовательности и находки. Для этого использовалась функция BLAST - "align two or more sequences". Карта локального сходства представлена на рис.2
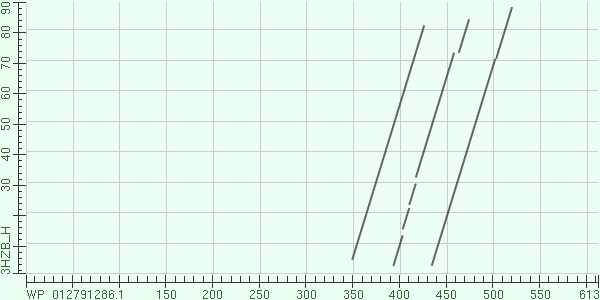
Рис. 2. Карта локального сходства
Из карты локального сходства можно предположить, что в glycoside hydrolase cодержится три участка, комплементарных практически всему моему белку, при этом интересно, что эти участки перекрываются между собой. Возможно мы видим результат двух дупликаций изначально одиночного участка.
Далее мною было проверено, какая часть находок принадлежит эукариотам. Выдача BLAST представлена на рис.3. Находок с e-value ≤ 0.001 оказалаось всего три.
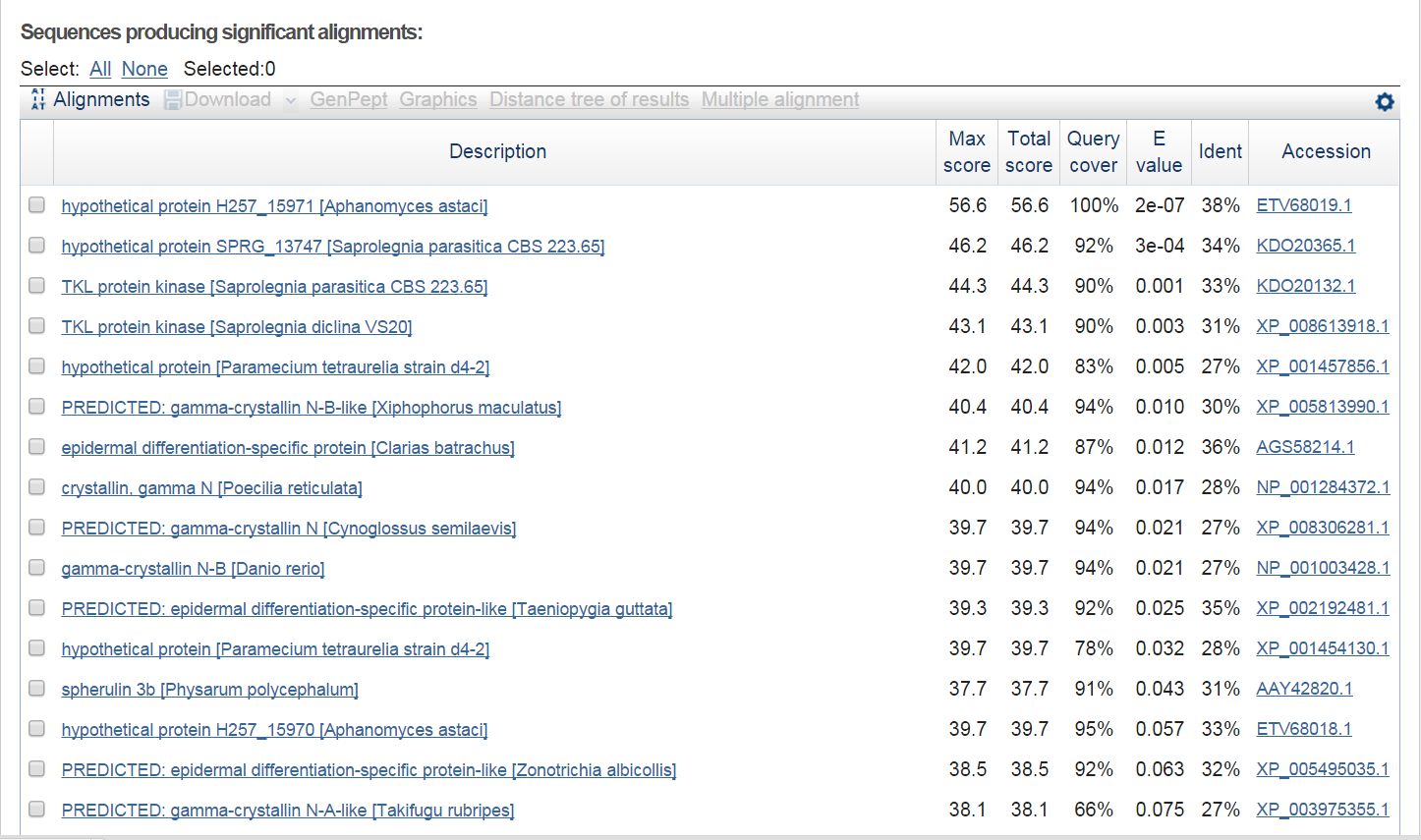
Рис. 3. Выдача BLAST с фильтром по эукариотам.
После этого мною были скачены последовательности находок с E-value ≤ 0.00001 и длиной, близкой к длине последовательности моего белка и произведено множественное выравнивание. Скачать файл в формате .fasta можно по этой ссылке. Процент функционально консервативных и консервативных колонок – 0%Дата последнего изменения - 16 марта 22:15