Целью этого задания был поиск белка, похожего на выданный мне в библиотеке PDB, и сравнение структур по средствам Jmol. Поиск проводился с помощью сервера PDBeFold. Всего похожих найдено было 86 белков. 8 из них полностью совпадали с рассматриваемым белком, и соответствовали восьми цепям данного белка, поэтому других белков - 78 штук. Сравнение проведено с белком, имеющим идентификатор 3SO0. Это белок из бактерии Clostridium beijerinckii. Эти две бактерии относятся к царству бактерий, но к разным типам. Clostridium beijerinckii - Firmicutes, a Flavobacterium johnsoniae - Bacteroidetes. Из названия очевидно, что данные два белка у этих бактерий имеют сходную функцию. На рис. 1 приведено частичное сравнение структур. Как видно из рисунка цепи практически совпадают. Цепь 3HZB:A - темно-голубая, 3SO0:H - светло-голубая. Подробнее о сравнении этих белков рассказано в таблице 1. Геометрическое ядро было выделено при помощи следующих команд:
select (68-82:A or 70-84:B) and (within(1.75, *A.CA)) and backbone and *B.CA
select (68-82:A or 70-84:B) and (within(1.75, *B.CA)) and backbone and *A.CA
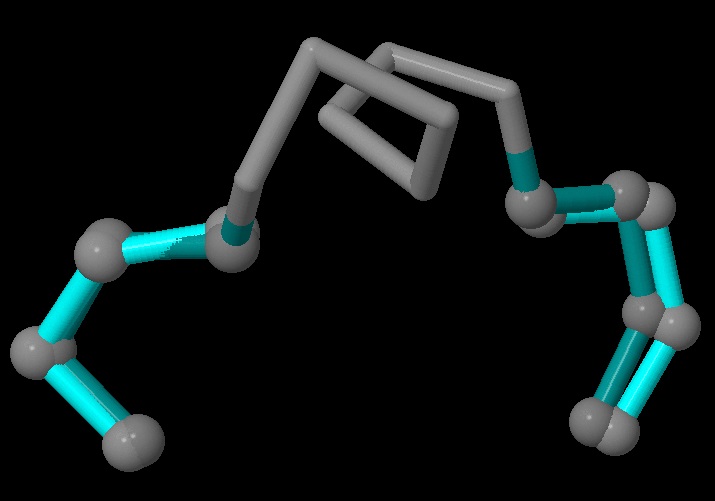 Рисунок 1. Сравнение структур белков 3HZB:A и 3SO0:H с помощью программы Jmol. Геометрическое ядро показано в шариковой модели.
Рисунок 1. Сравнение структур белков 3HZB:A и 3SO0:H с помощью программы Jmol. Геометрическое ядро показано в шариковой модели.
Таблица 1. Сравнение белков 1TJP и 2EKC, с помощью PDBeFold.
| ## | RMSD | Nalign | Ng | %seq | Title |
| 36 | 0.932 | 72 | 2 | 36.1 | CRYSTAL STRUCTURE OF A MUTANT T41S OF A BETAGAMMA-CRYSTALLIN DOMAIN FROM CLOSTRIDIUM BEIJERINCKII |
Дата последнего изменения - 16 марта 22:15