Учебный сайт Валяевой Анны
Предсказание парных выравниваний
В этом практикуме были использованы последовательности из файла aln_12.fasta. Целиком проект этого практикума можно скачать здесь.
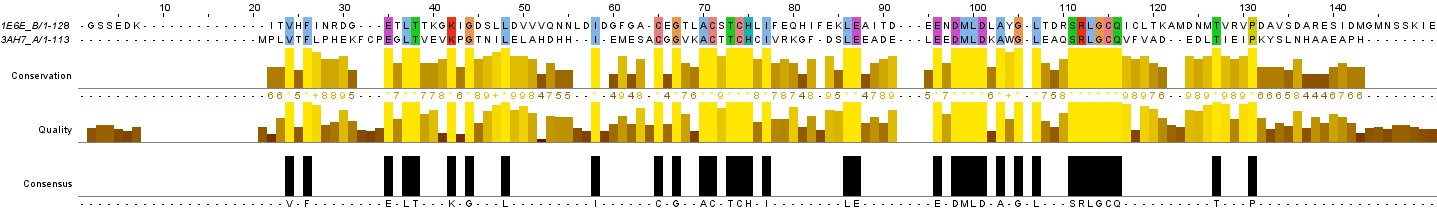
Из множественного выравнивания, изображенного на рисунке 1, удалив лишние последовательности (кроме последних двух), я получила парное выравнивание - рисунок 2.

Рис. 1. Исходное множественное выравнивание. Изображение получено с помощью программы Jalview.
Рис. 2. Парное выравнивание последних двух последовательностей из множественного. Изображение получено с помощью программы Jalview.
Глобальное выравнивание
С помощью программы needle пакета EMBOSS на kodomo из двух исходных последовательностей было посторено парное выравнивание (gap opening penalty 10.0, gap extension penalty 0.5) в формате needle и fasta:
>1E6E_B_1-128
GSSEDKITVHFINRD---GETLTTKGKIGDSLLDVVVQNNLDIDGFGACEGTLACSTCHL
IFEQHIFEKLEAITDEENDMLDLAYGLTDRSRLGCQICLTKAMDNMTVRVPD-AVSDARE
SIDMGMNSSKIE
>3AH7_A_1-113
-----MPLVTFLPHEKFCPEGLTVEVKPGTNILELAHDHHIEME--SACGGVKACTTCHC
IVRKG-FDSLEEADELEEDMLDKAWGLEAQSRLGCQVFV--ADEDLTIEIPKYSLNHAAE
APH---------
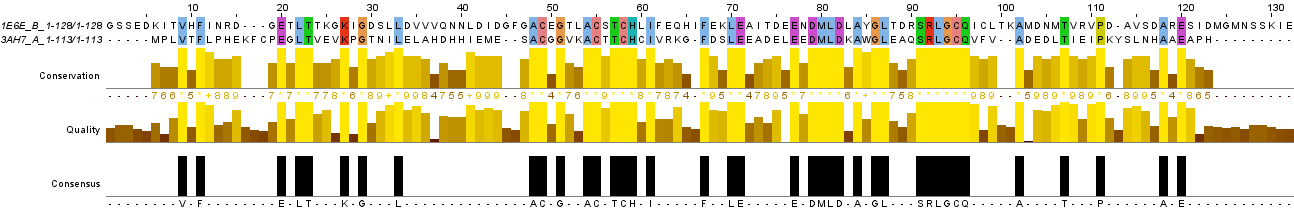
На рисунке 3 изображено это выравнивание в Jalview.
Рис. 3. Глобальное выравнивание. Изображение получено с помощью программы Jalview.
Наилучшее локальное выравнивание
Наилучшее локальное выравнивание (gap opening penalty 10.0, gap extension penalty 0.5) было получено с помощью программы water пакета EMBOSS на kodomo в формате water и fasta:
>1E6E_B_1-128
ETLTTKGKIGDSLLDVVVQNNLDIDGFGACEGTLACSTCHLIFEQHIFEKLEAITDEEND
MLDLAYGLTDRSRLGCQICLTKAMDNMTVRVP
>3AH7_A_1-113
EGLTVEVKPGTNILELAHDHHIEME--SACGGVKACTTCHCIVRKG-FDSLEEADELEED
MLDKAWGLEAQSRLGCQVFV--ADEDLTIEIP
На рисунке 4 изображено это выравнивание в Jalview.
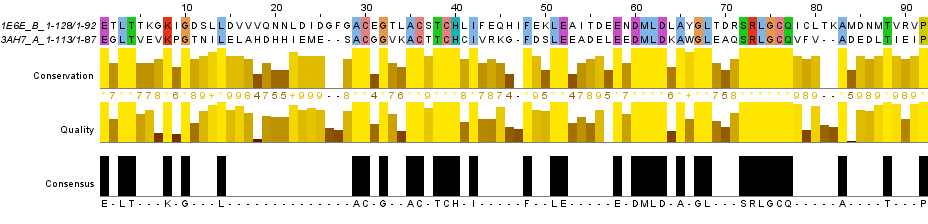
Рис. 4. Наилучшее локальное выравнивание. Изображение получено с помощью программы Jalview.
Парное выравнивание последовательностей двух заведомо негомологичных белков
Для построения парного выравнивания двух заведомо негомологичных белков я использовала последовательность своего белка (RefSeq: NP_070832.1, hypothetical protein) и последовательность белка другого студента (RefSeq: YP_002797966.1, Rhodanese). Были использованы программы needle (результат в формате needle и fasta) и water (результат в формате water и fasta) на сервере kodomo. Построенные парные выравнивания изображены на рисунках 5 и 6.

Рис. 5. Глобальное выравнивание последовательностей двух заведомо негомологичных белков. Изображение получено с помощью программы Jalview.
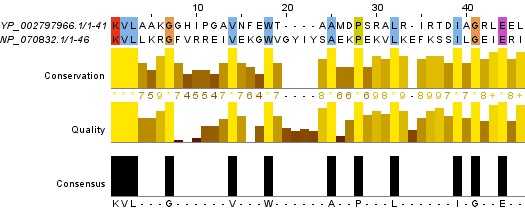
Рис. 6. Наилучшее локальное выравнивание последовательностей двух заведомо негомологичных белков. Изображение получено с помощью программы Jalview.
Параметры выравниваний
В таблице 1 представлены параметры выравниваний, полученных в ходе этого практикума (рисунки 2-6).
Таблица 1. Параметры выравниваний.
| Выравнивание | Длина | Число и процент совпадений | Число и процент сходных остатков (Similarity) | Число и процент гэпов | Число открытий гэпов |
| Парное выравнивание последних двух последовательностей, полученное из множественного | 151 | 35; 23,2% | 53; 35,1% | 43; 28,5% | 11 |
| Глобальное выравнивание последних двух последовательностей, полученное из множественного | 132 | 39; 29,5% | 69; 52,3% | 23; 17,4% | 7 |
| Наилучшее локальное выравнивание последних двух последовательностей, полученное из множественного | 92 | 35; 38,0% | 59; 64,1% | 5; 5,4% | 3 |
| Глобальное выравнивание последовательностей двух заведомо негомологичных белков | 283 | 27; 9,5% | 46; 16,3% | 189; 66,8% | 9 |
| Наилучшее локальное выравнивание последовательностей двух заведомо негомологичных белков | 46 | 12; 26,1% | 20; 43,5% | 5; 10,9% | 2 |
Как видно из таблицы 1, первое выравнивание не очень достоверно, поскольку в нем много гэпов, появившихся из-за того, что выравнивание первоначально было построено как множественное. Заметны значительные различия между глобальным и локальным выравниваниями негомологичных белков. При этом локальное выравнивание заведомо негомологичных белков нельзя назвать достоверным, поскольку длина выравнивания мала.
Участок различия парных выравниваний 1 и 2
Чтобы сравнить два парных выравнивания, я добавила к парному выравниванию 1 (рисунок 2) парное выравнивание 2 (рисунок 3). Один из участков несовпадения (позиции 56-64) в окружении совпадающих участков изображен на рисунке 7. Расхождение двух парных выравниваний возникло, потому что первое выравнивание было взято из множественного и было выровнено относительно других последовательностей, а второе было изначально построено как парное. Целиком выравнивание можно посмотреть в проекте.
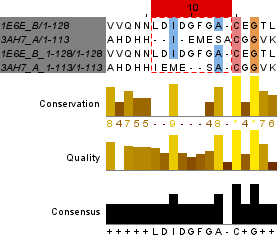
Рис. 7. Участок различия парных выравниваний 1 и 2 (позиции 51-69). Изображение получено с помощью программы Jalview.
Проверка правильности выравниваний
Знаком "+" я отметила те колонки выравниваний 1 и 2, в которых, по моему мнению, выравнивание является достоверным. С помощью программ SupCheck и RasMol было построено совмещение пространственных структур двух белков, колонки участков совмещения отмечены буквой "S". Ошибка 1го рода: C_alpha атомы пары остатков в колонке выравнивания, отмеченной "+", в структуре, очевидно, не совмещаются (Сомнения трактуйте в свою пользу). Ошибка 2го рода: C_alpha атомы двух остатков, очевидно, хорошо совмещаются, но соответствующие буквы либо не находятся в одной колонке, либо в одной колонке, но колонка не отмечена "+". Было выявлено 9 ошибок 1го рода и 3 ошибки 2го рода. Результат этого задания изображен на рисунке 8.

Рис. 8. Сопоставление парных выравниваний и пространственной структуры последовательностей. Изображение получено с помощью программы Jalview.
Дата последнего обновления: 27.05.14
©Валяева Анна