Учебный сайт Валяевой Анны
Построение дерева по нуклеотидным последовательностям
Для выполнения задания я скачала последовательности РНК малой субъединицы рибосомы (16S rRNA) бактерий, которые я использовала в предыдущих практикумах: в базе полных геномов NCBI в файлах с расширением .frn лежат последовательности аннотированных РНК. Затем было проведено выравнивание этих последовательностей с помощью сервера Muscle. После этого по выравненным последовательностям в программе MEGA методом Neighbor-Joining было построено дерево, изображенное на рисунке 1.
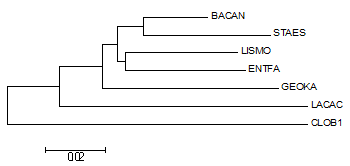
Рис. 1. Филогенетическое дерево выбранных бактерий по генам 16S рРНК. Изображение получено с помощью программы MEGA.
Получившееся дерево значительно отличается от деревьев из предыдущих практикумов и "правильного" дерева, приведенного на рисунке 2. Ни одна из нетривиальных ветвей, построенного по 16S rRNA, не совпадает с ветвями "правильного" дерева.
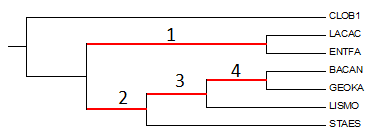
Рис. 2. "Правильное" дерево выбранных бактерий. Изображение получено с помощью программы MEGA.
Реконструкция дерева по нуклеотидным последовательностям оказалась менее качественной, чем реконструкция по белкам. Это, вероятно, связано с тем, что выравнивания нуклеотидных последовательностей менее достоверны.
Построение и анализ дерева, содержащего паралоги
С помощью программы blastp был проведен поиск гомологов белка CLPX_BACSU в протеомах рассматриваемых бактерий. Полученные последовательности белков-гомологов были выравненены на сервере Muscle. Дерево гомологов было построенно в программе MEGA методом Maximum likelihood, оно изображено на рисунке 3.
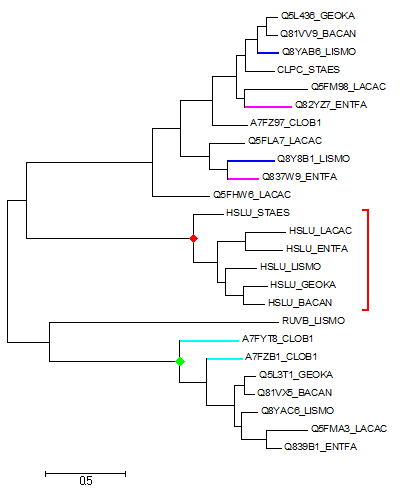
Рис. 3. Дерево гомологов белка CLPX_BACSU. Изображение получено с помощью программы MEGA.
Красная скобочка охватывает ветвь белков-ортологов, голубые, синие и фиолетовые ветви ведут к парам паралогов. Красный ромбик означает разделение путей эволюции белков в результате видообразования, зеленый - дупликация гена.
Дата последнего обновления: 13.03.15
©Валяева Анна