Мини-обзор генома и протеома бактерии Vibrio panuliri
Купцова Дарья
Факультет биоинженерии и биоинформатики, МГУ, Москва, Россия
Введение
Vibrio panuliri — вид морской бактерии из семейства Vibrionaceae. Она была выделена из яиц лангуста (Panulirus penicillatus), собранных в Андаманском море, Индийский океан, в 2014 году [1]. Бактерия мало изучена, но уже сейчас его относят к группе риска 1 в соответствии с немецкой классификацией прокариот по группам риска, TRBA 466 (группа 1: биологический агент, который с наибольшей вероятностью вызовет заболевание человека) (S1).
Классификация бактерии (S2):
Суперцарство: Bacteria
Царство: Pseudomonadati
Тип: Pseudomonadota
Класс: Gammaproteobacteria
Порядок: Vibrionales
Семейство: Vibrionaceae
Род: Vibrio
Вид: Vibrio panuliri
Материалы и методы
Из NCBI были загружены: нуклеотидная последовательность генома, нуклеотидная последовательность CDS генов, нуклеотидная последовательность РНК генов, таблица характеристик генома. Ссылка на сайт, с которого были загружены файлы представлены в разделе «Список литературы» (S3).
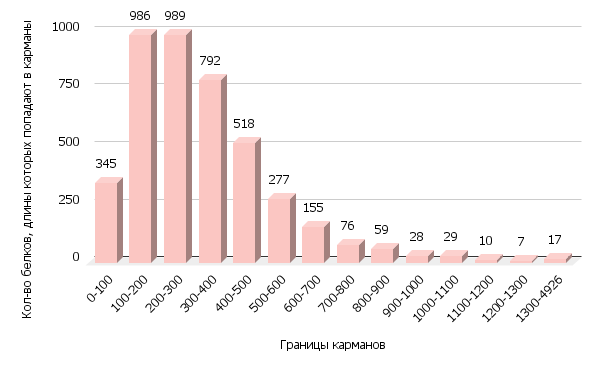
С помощью функции Google sheets (СЧЁТЕСЛИМН) построена гистограмма длин белков (S4 и Рисунок 1).
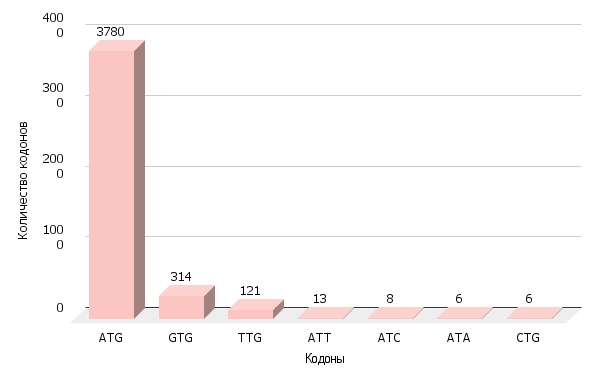
С помощью команд bash (grep, cut, sort, uniq) была извлечена таблица со всеми старт-кодонами бактерии и таблица старт-кодонов псевдогенов. Далее с помощью функций Google sheets (ЕСЛИОШИБКА, ВПР, -) построена диаграмма количества старт-кодонов (S5 и Рисунок 2).
С помощью программы Python 3.11 (использованные команды: input, with, for, if/else, list, set, sort, append, print, split, методы для словарей, списков, строк) получен файл с перечислением последовательностей тРНК, их количеством и аминокислотами, которые переносят последовательности (S6). Далее с помощью Google sheets были построены таблицы соответствующие данным файла (S7, Таблица 1, Таблица 2, Таблица 3, Таблица 4, Таблица 5).
Результаты
Длина белков, закодированных в геноме Vibrio panuliri
Как видно из гистограммы (Рисунок 1), преимущественно днина белков составляет до 900 аминокислот. При большем количестве аминокислот в белке, количество соответствующих белков сокращается. В диапазон более 1300 и до максимальной длины белка (4926) попадают лишь 17 белков (один белок на каждые 213 значений). Пик значений приходится на диапазон 100-300.
Старт-кодоны
Как видно из диаграммы (Рисунок 2) большинством старт-кодонов в геноме Vibrio panuliri является кодом ATG, соответствующий аминокислоте метионин, который является наиболее распространенным старт-кодом. Другие кодоны (GTG и TTG) являются альтернативными старт-кодонами. Находясь в середине кодирующей последовательности они кодирую соответствующие аминокислоты, но находясь в начале альтернативные кодоны соответствуют стартовому метионину. Это происходит, потому что альтернативный старт-кодоны частично комплементарны антикодону инициирующей аминоацил-тРНК (CAT). Остальные старт-кодоны (ATT, ATC, ATA и CTG) являются «слабыми» старт-кодонами и выполняют свою стартовую функцию в комбинации с «сильными» последовательностями и элементами, способствующими трансляции.
Аминокислоты, переносимые тРНК
Как видно из таблицы 1 всего последовательностей тРНК 111, различных лишь 45, из чего можно сделать вывод о неравном количестве различных последовательностей, это также описано в таблице 4. Наиболее частые аминокислоты, переносимые тРНК — Leu и Gly, наименее частые — His и Trp (Таблица 2).
Изучая таблицу 3 можно заметить что аминокислота Asp переносятся 5-ю последовательностями, но все они одинаковые, хотя аминокислота Asp может быть закодирована двумя различными триплетами (GAU, GAC). Из этого наблюдения можно сделать вывод о том, что при трансляции у бактерии Vibrio panuliri имеет место wobble-взаимодействие (основание на 5'’-конце антикодона не ограничивается пространственно двумя другими основаниями, что позволяет развивать водородные связи с другими основаниями, присутствующими на 3'’-конце кодона, то есть одна тРНК может соединятся с несколькими иРНК). Схожий вывод можно сделать об аминокислотах Ala, Asn, Cys, Gln, Lys исходя из данных представленных в таблице 3.
Далее информация описанная в таблицах 4 и 5, возможно ошибочная, однако в процессе изучения последовательностей переносящих аминокислоты Glu и Phe, было замечено незначительное расхождение в последовательностях (выделено в таблицах). Из этого расхождения можно предположить, что в этих последовательностях произошли мутации, а именно в одной из четырёх последовательностей тРНК, переносящих аминокислоту Glu, произошла одна инсерция (добавился один нуклеотид) и в одной из трех последовательностей тРНК, переносящих аминокислоту Phe, произошла одна трансверсия(замена пиримидинового основания на пуриновое основание) и одна транзиция (замена пуринового основания на другое пуриновое основание). Однако эти мутация предположительно не повлияли на антикодон. Для подтверждения предположения необходимо найти в последовательности сам антикодон, что на данных момент не представляется возможным.
| Количество аминокислот | Количество различных последовательностей | Всего последовательностей |
|---|---|---|
| 20 | 45 | 111 |
| Аминокислота | Количество последовательностей, переносящих аминокислоту | Аминокислота | Количество последовательностей, переносящих аминокислоту |
|---|---|---|---|
| Ala | 8 | Leu | 13 |
| Arg | 9 | Lys | 3 |
| Asn | 4 | Met | 8 |
| Asp | 5 | Phe | 3 |
| Cys | 4 | Pro | 4 |
| Gln | 5 | Ser | 7 |
| Glu | 4 | Thr | 6 |
| Gly | 10 | Trp | 2 |
| His | 2 | Tyr | 3 |
| Ile | 6 | Val | 5 |
| Аминокислота | Количество различных последовательностей, переносящих аминокислоту | Аминокислота | Количество различных последовательностей, переносящих аминокислоту |
|---|---|---|---|
| Ala | 3 | Lys | 3 |
| 5 | Met | 1 | |
| Arg | 1 | 3 | |
| 4 | 4 | ||
| 3 | Phe | 2 | |
| 1 | 1 | ||
| Asn | 4 | Pro | 3 |
| Asp | 5 | 1 | |
| Cys | 4 | Ser | 4 |
| Gln | 5 | 1 | |
| Glu | 3 | 2 | |
| 1 | Thr | 2 | |
| Gly | 8 | 2 | |
| 1 | 1 | ||
| 1 | 1 | ||
| His | 1 | Trp | 2 |
| 1 | Tyr | 2 | |
| Ile | 5 | 1 | |
| 1 | Val | 3 | |
| Leu | 1 | 1 | |
| 7 | 1 | ||
| 2 | |||
| 1 | |||
| 2 |
| Аминокислота | Последовательности, переносящие аминокислоту | Количество последовательностей, переносящих аминокислоту |
|---|---|---|
| Glu | GTCCCGTTCGTCTAGAGGCCTAGGACACCGCCCTTTCACGGCGGTAACAG GGGTTCGACTCCCCTACGGGATACCA |
3 |
| GTCCCGTTCGTCTAGAGGCCTCAGGACACCGCCCTTTCACGGCGGTAACA GGGGTTCGACTCCCCTACGGGATACCA |
1 |
| Аминокислота | Последовательности, переносящие аминокислоту | Количество последовательностей, переносящих аминокислоту |
|---|---|---|
| Phe | GCCCGGATAGCTCAGTCGGTAGAGCAGAGGATTGAAAATCCTCGTGTCGG TGGTTCGATTCCGCCTCCGGGCACCA |
2 |
| GCCTCGATAGCTCAGTCGGTAGAGCAGAGGATTGAAAATCCTCGTGTCGG TGGTTCGATTCCGCCTCGAGGCACCA |
1 |
Сопроводительные материалы
- Виды: Vibrio panuliri - ссылка на страницу [2], откуда взята дополнительная информация о бактерии.
- Vibrio panuliri - NCBI - NLM - ссылка на таксономию Vibrio panuliri.
- Index of /genomes/ - ссылка на сайт с которого взяты последовательности генома.
- CDS from genome of Vibrio panuliri - таблица с гистограммой распределения длин белков.
- Start codons - таблица с диаграммой, показывающей количество различных старт-кодонов.
- tRNA.py - программа для определения аминокислот, переносимых последовательностями тРНК, и подсчете различных последовательностей тРНК.
- tRNA - таблицы с информацией об аминокислотах, переносимых тРНК
Список литературы
- Kumari P, Poddar A, Schumann P, Das SK. Vibrio panuliri sp. nov., a marine bacterium isolated from spiny lobster, Panulirus penicillatus and transfer of Vibrio ponticus from Scophthalmi clade to the newly proposed Ponticus clade. Res Microbiol. 2014 Dec;165(10):826-35;
- Parte, A.C., Sardà Carbasse, J., Meier-Kolthoff, J.P., Reimer, L.C. and Göker, M. (2020). List of Prokaryotic names with Standing in Nomenclature (LPSN) moves to the DSMZ. International Journal of Systematic and Evolutionary Microbiology, 70, 5607-5612