Практикум 8
В этом практикуме были опробованы разные алгоритмы BLAST через веб-интерфейс и локально.
Задание 1
Заданиие: найти в геноме эукариота ген, кодирующий δ-субъединицу АТФ-синтазы
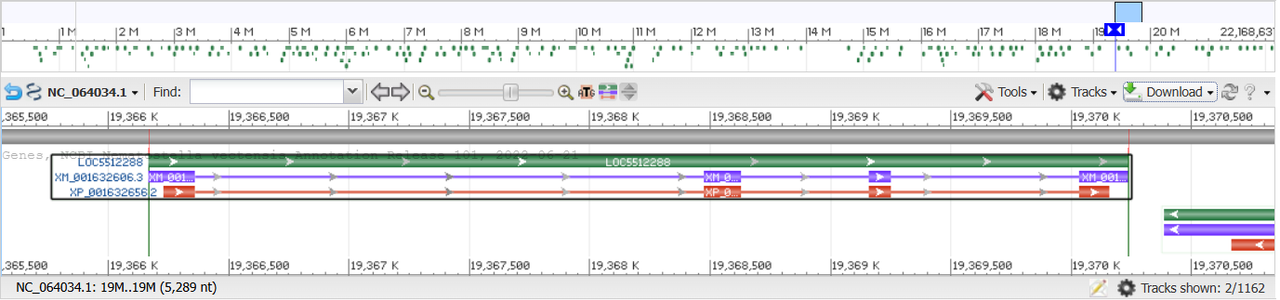
С помощью текстового поиска была найдена последовательность белка, аннотированного как δ-субъединица АТФ-синтазы с идентификатором XP_001632656.2 в организме Nematostella vectensis. Сохрананён в файле: FASTA.
Идентификатор нуклеотидной записи, к которой оттносится ген: NC_064034
В геном браузере был получен ген, кодирующий дельту δ-субъединицу АТФ-синтазы (XP_001632656.2) и сохранён в файл
Задание 2
Задание: попробовать разные варианты BLAST для фрагмента ДНК
Выбранное семейство: Собачьи (Canidae), так как анемона-стерлядь, выбранная в практикуме 7, является первичноротой, и в задании, в таком случае предоставлялся выбор между Кошачьи (Felidae) или Собачьи (Canidae).
Для первого поиска BLAST по последовательностям Собачьих для белок-кодирующей последовательности δ-субъединицы АТФ-синтазы анемоны-стерляди NC_064034 по методу blastn, так как организмы очень удалены друг от друга эволюционно. База данных для поиска refseq_genomes, число сборок в ней — 7 штук. Оптимальные параметры для запуска: длина слова 7 (для увеличения совпадения) и Е-value 0.001 (для исключения плохих совпадениЙ), все остальные параметры стандартны.
Результат: 25 последовательностей, но у всех Query cover 1%, что не удивительно, так как алгорит пытается найти совпадения не учитывая избыточность генетического кода. (В файле написано 27 найденных совподений, однака две пары этих совпадений являются находками в одной последовательности с небольшим перекрытием в одной паре и разрывом в два нуклеотида во второй)
Файл с результатом: blastn
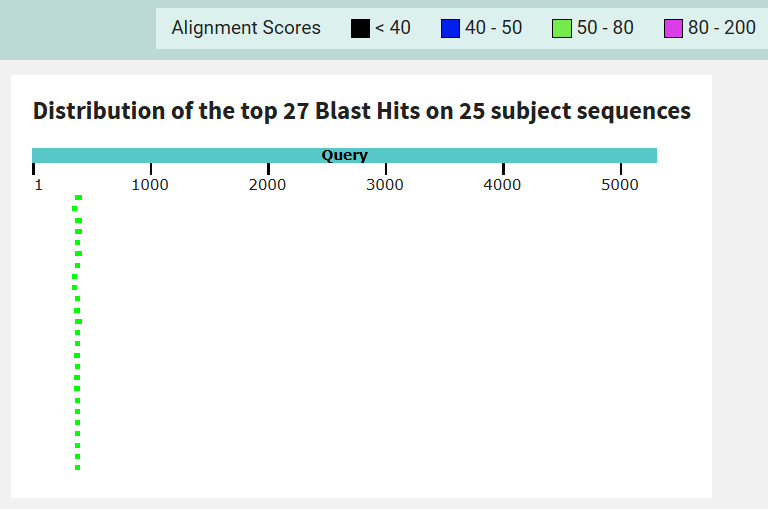
Для второго поиска BLAST по последовательностям Собачьих для белок-кодирующей последовательности δ-субъединицы АТФ-синтазы анемоны-стерляди NC_064034 по методу tblastn, в котором на вход подаётся последовательность белка, и алгоритм ищет совпадения уже по шестирамочно переведенной нуклеотидной базе данных, так как он работает быстрее чем tblastx и менее чувствителен к ошибкам секвенирования. База данных для поиска refseq_genomes, число сборок в ней — 7 штук. Оптимальные параметры для запуска: длина слова 2 (для увеличения совпадения), все остальные параметры стандартны.
Результат: 13 последовательностей, с Query выше 65%. (В файле написано 26 найденных совподений, однака девять пар и две тройки этих совпадений являются находками в одной последовательности)
Файл с результатом: tblastn
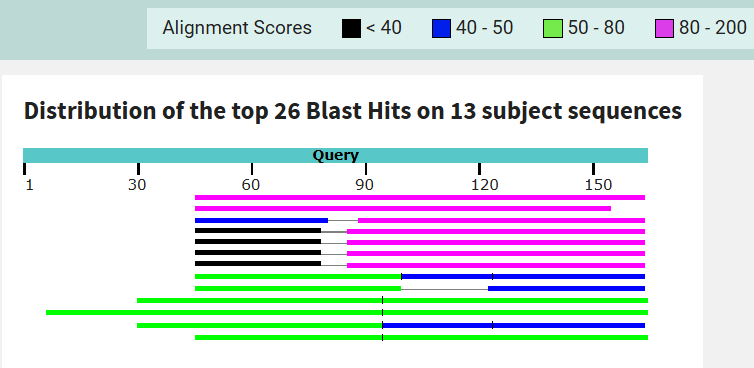
Задание 3
Задание: найти в геноме эукариота гены основных рибосомальных РНК по далекому гомологу.
На локальном компьютере с помощью скаченных утильт BLAST+ была проиндексирована последовательность анемоны-стерляди для последующей работы локального BLAST.
Команда для выполнения:
makeblastdb -in .\GCF_932526225.1_jaNemVect1.1_genomic.fna -dbtype nucl -out anemone_db, где после "-in" указана последовательность в fasta формате, а после "-dbtype" указан тип последовательности
Далее для каждой скаченной рРНК последовательности Escherichia coli был проведён локальный поиск BLAST по полученной базе данных по методу blastn (так как он отлично подходит для поиска далёких последовательностей).
Команда для выполнения 16S рРНК:
blastn -task blastn -query rRNA_ecoli_16S.txt -db anemone_db -out anemone_16S.txt -evalue 0.01 -outfmt 7.
Выдача команды: anemone_16S.txt.
Blastn нащёл 94 гомологичных участков, в 10 разных нуклеотидных последовательностях анемоны-стерляди, но все эти выравнивания на одном участке 1494-1536 (кроме одного, где выравнивание с 1493). В остальном почти все параметры совпадают, E-value очень низок, из чего можно сделать вывод, что в последовательности анемоны очень много повторов.
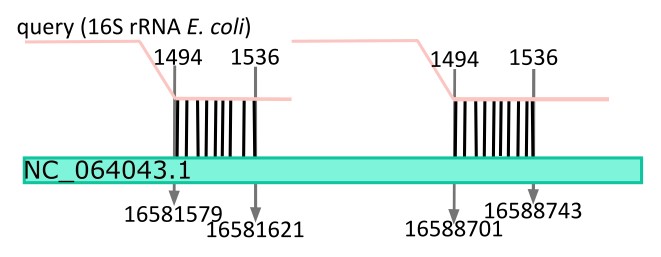
Команда для выполнения 23S рРНК:
blastn -task blastn -query rRNA_ecoli_23S.txt -db anemone_db -out anemone_23S.txt -evalue 0.01 -outfmt 7
Выдача команды: anemone_32S.txt.
Blastn нащёл 305 гомологичных участков, в 12 разных нуклеотидных последовательностях анемоны-стерляди, причём у E. coli выравнивались 1899-1991, 185-279, 2442-2613 и 451-528 (иногда чуть меньше нуклеотидов, и небольшой сдвиг, исключение 679-829, но она единственная).
Задание 4
Задание: подберать пару геномов и построить карты их локального сходства.
Для этого задания были выбраны митохондиальные геномы человека и дрозофилы, NC_012920.1 (Homo sapiens mitochondrion, complete genome) и NC_024511.2 (Drosophila melanogaster mitochondrion, complete genome), потому что митохондиальные геномы содержат как высококонсервативные гены, так и высоковариабельные, так же они компакты а значит на карте будет не слишком много точек.
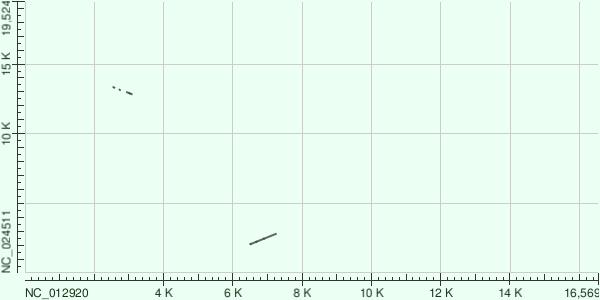
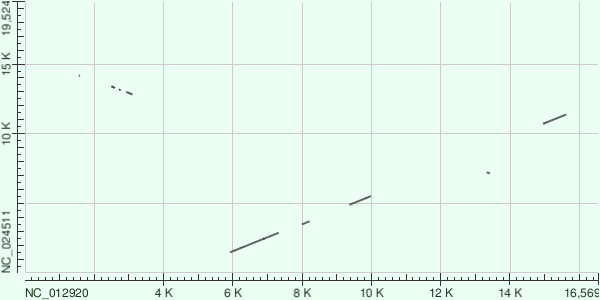
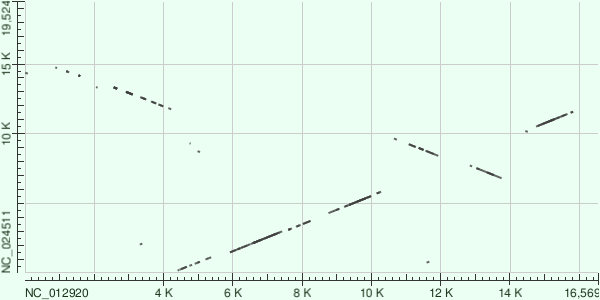
Карты локального сходства были построены на основе трёх алгоритмов: blastn, megablast и tblastx (для blastn и tblastx параметры по умолчанию, для megablast длина слова была сокращена до 16 так как при 28, небыло найделно совпадений).
Изучая самую подробную карту, построенную tblastx, можно хорошо увидеть две инверсии в последовательнсти человека (нижней) на участках 1000-4200 и 10450-13850 (участок 1000-4200 находится не внизу, так как митохондриальные геномы кольцевые, и видимо секвинировались с разных точек)